Разделы презентаций
- Разное
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Геометрия
- Детские презентации
- Информатика
- История
- Литература
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Bacterial Plasmids in Fundamental and Applied Science Внехромосомные
Содержание
- 1. Bacterial Plasmids in Fundamental and Applied Science Внехромосомные
- 2. Плазмиды - внехромосомные элементы наследственности, способные к
- 3. F - плазмида – прототип “fertility factor”
- 4. ПлазмидыСтруктура - кольцевые или линейные молекулы ДНК
- 5. ПлазмидыКонтролируемый фенотип: Устойчивость к антибиотикам, тяжелым металлам.
- 6. Конъюгация – процесс обмена генетической информацией между
- 7. Обозначение плазмидПрефикс “p”«Исторические» обозначения: RSF1010, R100, NAH7.R100 фенотип – Tc Cm Sm Su TraГенотип – tet+cat+aadA+sul+tra+
- 8. Молекулярная организация плазмид Базовый репликон (basic replicon):ori
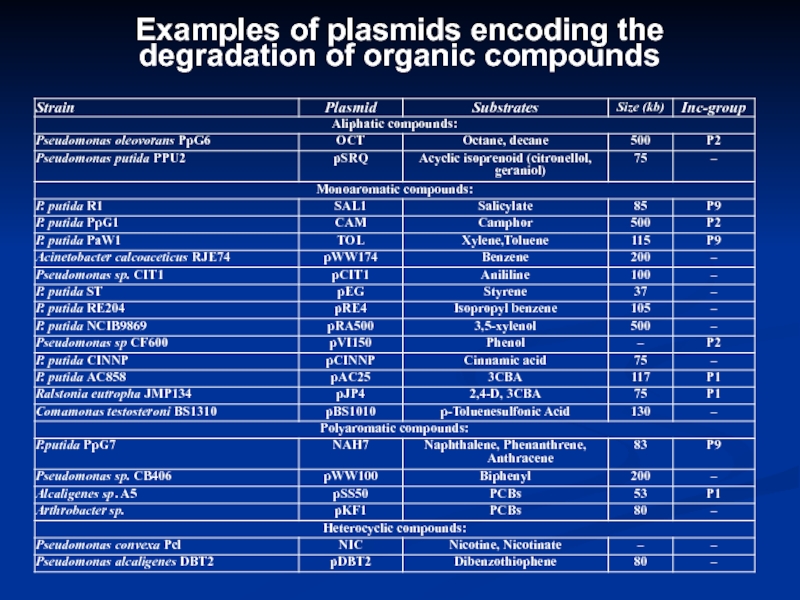
- 9. Examples of plasmids encoding the degradation of organic compounds
- 10. Incompatibility Groups of Pseudomonas Degradative Plasmids
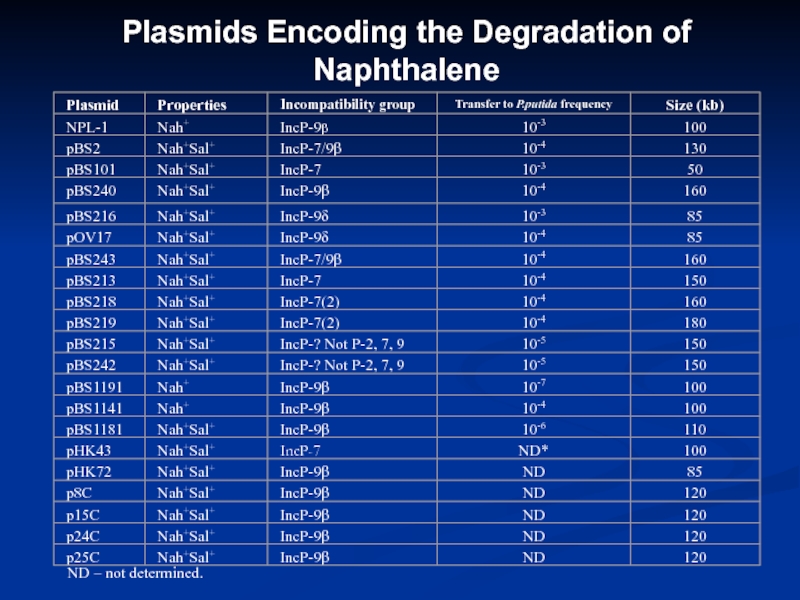
- 11. Plasmids Encoding the Degradation of NaphthaleneND – not determined.
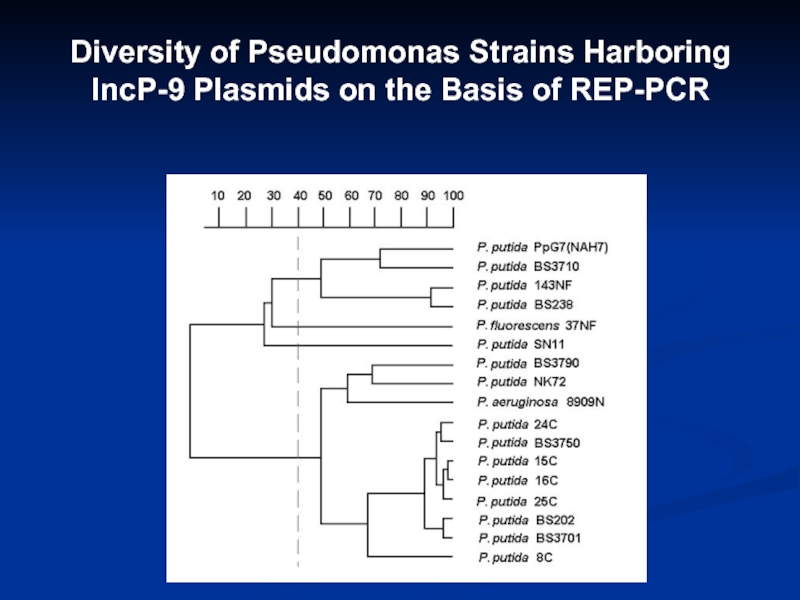
- 12. Diversity of Pseudomonas Strains Harboring IncP-9 Plasmids on the Basis of REP-PCR
- 13. Diversity of the Naphthalene Catabolic Plasmids Belonging to IncP-9 Group on the Basis of RFLP Analysis
- 14. Phylogenetic Tree of IncP-9 Plasmid Group Created on the Basis of rep-Gene Sequences
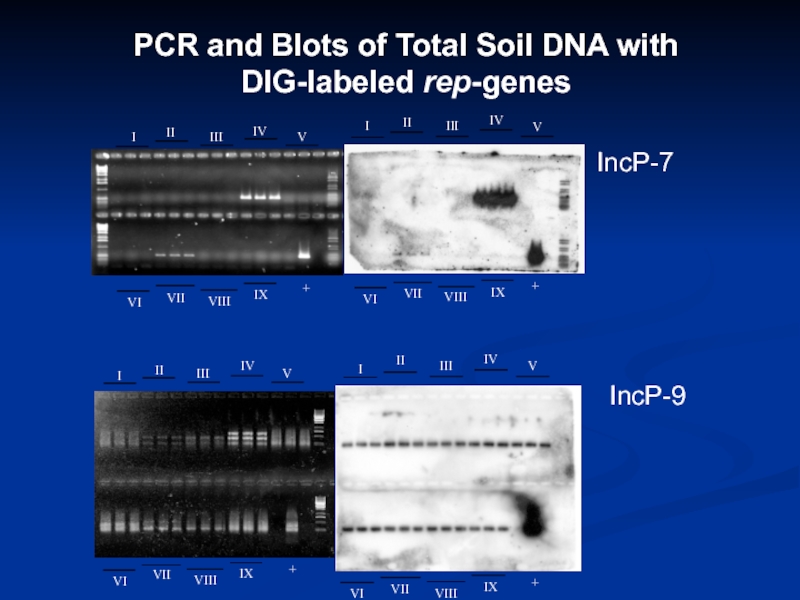
- 15. PCR and Blots of Total Soil DNA with DIG-labeled rep-genesIncP-7IncP-9
- 16. Phylogenetic Tree of IncP-7 Naphthalene Degrading Plasmids Based on RFLP
- 17. Pathways of Naphthalene and Phenanthrene Degradationnaphthalenephenanthrene1,2-dihydroxynaphthalene1-hydroxy-2-naphthoic acidNADНsalycilatecatecholprotocatechuic
- 18. NAH Catabolic Gene Organization and RegulationABCFDERGHINLJKUpper pathwayLower pathwayPiruvate + AcetaldehydeSalicylate -> 2-oxo-4-hydroxypentanoateNaphthalene -> SalicylateNahR Regulatory proteinSalicylate
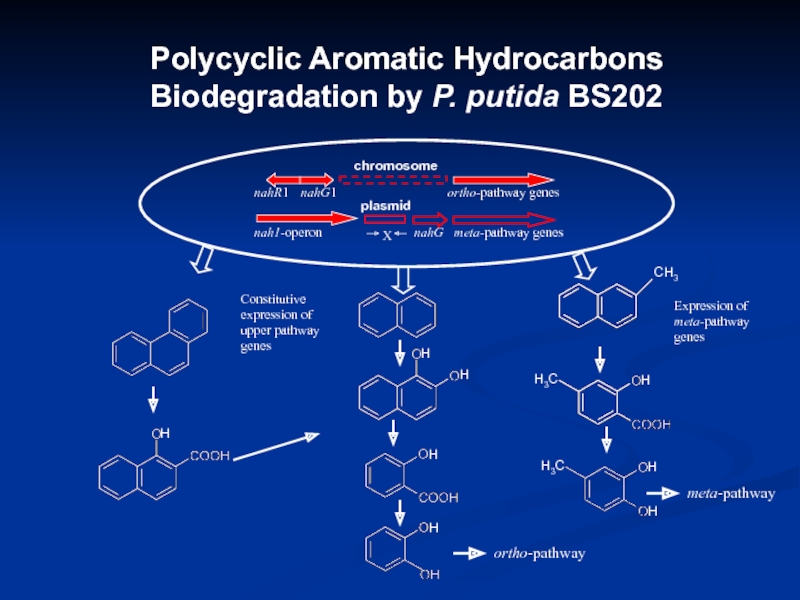
- 19. Polycyclic Aromatic Hydrocarbons Biodegradation by P. putida BS202meta-pathway
- 20. Organization of PAH catabolic genes in some Pseudomonas putida strainsnahG1nahGmeta-pathwaynah1-operonBS3790 chromosomepBS1191pBS1192
- 21. Diversity of Pseudomonas nahAc genes (RFLP analysis)
- 22. Distance Tree of Classic NahG Subtypes Based on Cluster Analisys of AA Sequences
- 23. SgpIMpi?SgpK+OHCOOHsalicylate gentisate
- 24. Is there a degradative (D) “superplasmid” capable
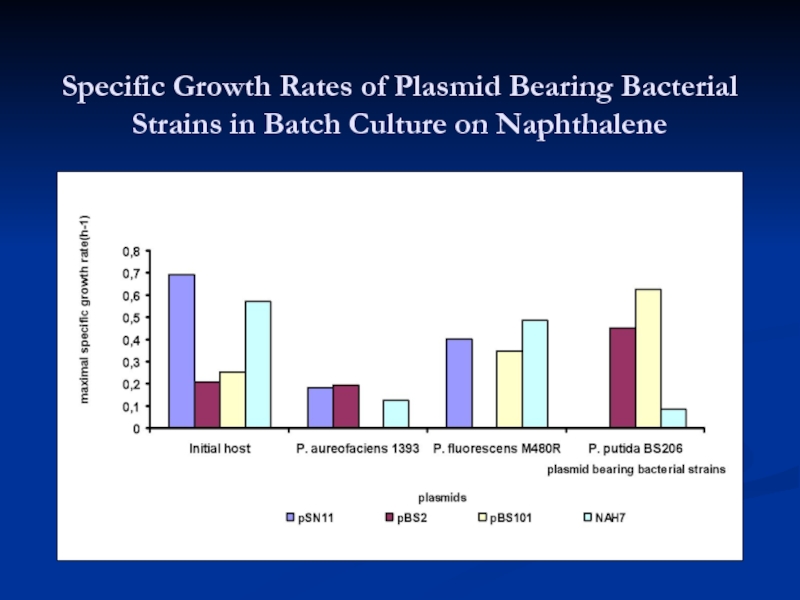
- 25. Specific Growth Rates of Plasmid Bearing Bacterial Strains in Batch Culture on Naphthalene
- 26. Diversity of Microorganisms from Oil Slimes
- 27. Two nahU Gene (salicylate hydroxylase) Subtypes Restriction
- 28. Natural rhizosphere strains combining both degradative abilities
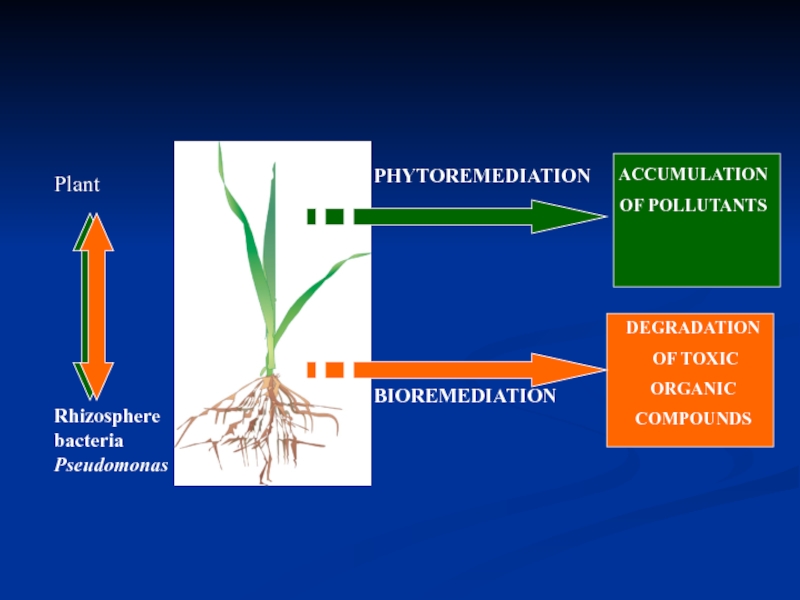
- 29. PlantPHYTOREMEDIATIONBIOREMEDIATIONRhizosphere bacteriaPseudomonasDEGRADATION OF TOXICORGANICCOMPOUNDSACCUMULATIONOF POLLUTANTS
- 30. Effect of naphthalene degradative plasmids on biosynthesis
- 31. Mechanisms of arsenic resistance in microorganisms
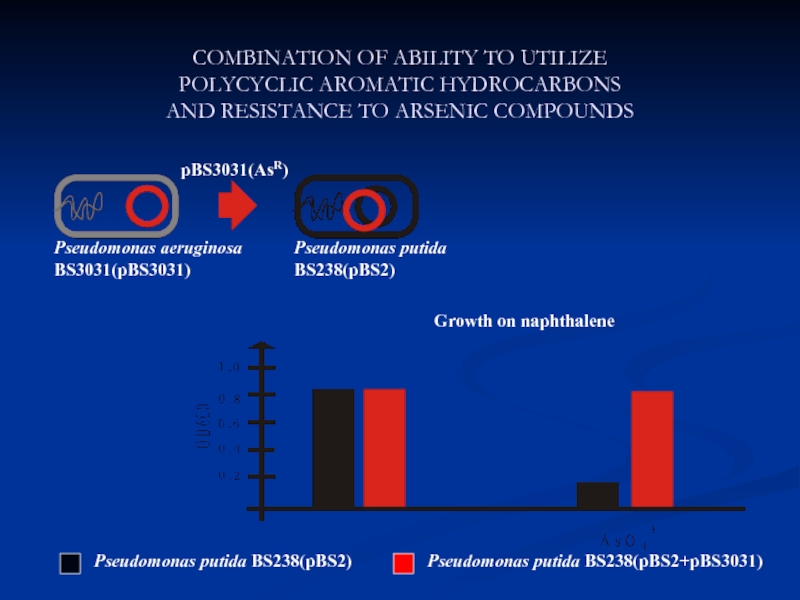
- 32. COMBINATION OF ABILITY TO UTILIZE POLYCYCLIC
- 33. Oil Contamination in Western Siberia The view of oil-contaminated site. July, 1999.
- 34. Extreme Environmental FactorsToxic chemical agentsHeavy metalsRadionucleidesUV-lightLow or high pH valuesHigh salinityWater deficiencyOxygen deficiency
- 35. Dendrogram of Oil-degrading Strains Based on Their Catabolic and Physiological Properties
- 36. Слайд 36
- 37. Слайд 37
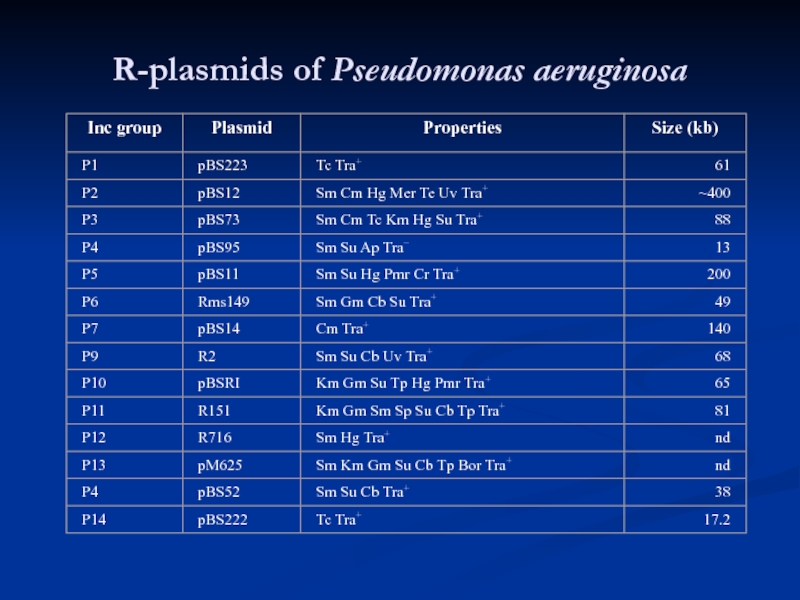
- 38. R-plasmids of Pseudomonas aeruginosa
- 39. The construction of multifunctional PGPR Pseudomonas
- 40. The effect of crude oil hydrocarbons on
- 41. The History of Bacterial Genetics (S.E. Luria,
- 42. Thank you!
- 43. Скачать презентанцию
Слайды и текст этой презентации
Слайд 1 Bacterial Plasmids in Fundamental and Applied Science Внехромосомные генетические элементы у
бактерий
State University, Pushchino, Russia.Слайд 2Плазмиды - внехромосомные элементы наследственности, способные к автономной репликации.
Термин введен Ледербергом в 1952 году.
Эписомы – генетические
элементы, способные реплицироваться в двух альтернативных состояниях: в интегрированном в хромосому и независимо от хромосомы.Конъюгативные плазмиды – способны обеспечивать перенос ДНК путем конъюгации.
Неконъюгативные плазмиды – не способны обеспечивать перенос ДНК путем конъюгации.
Мобилизуемые плазмиды – способны передаваться в реципиентные клетки с помощью конъюгативных плазмид.
Криптические плазмиды – плазмиды, фенотипические признаки для которых не установлены.
Плазмиды
Слайд 3F - плазмида – прототип “fertility factor” – ответственный за
конъюгационный перенос в штаммах E. coli.
F’ – плазмида –
дериват F – плазмиды, содержащий сегменты бактериальной хромосомы. Hfr –штаммы – (high frequency of recombination) – штамм, несущий в составе хромосомы плазмиду и, следовательно, способный осуществлять ориентированный перенос хромосомных генов в подходящий реципиент.
R – плазмиды
RTF – фактор – часть плазмиды резистентности, способная осуществлять конъюгационный перенос генов резистентности.
Плазмиды
Слайд 4Плазмиды
Структура - кольцевые или линейные молекулы ДНК размером от 2
до 600 т.п.н.
Число копий – от 1 до 1000
(малокопийные и мультикопийные) Круг хозяев – узкий круг (nhr – narrow host range), широкий круг хозяев (bhr – broad host range)
Способность к конъюгационному переносу (конъюгативные, неконъюгативные, мобилизуемые)
Группы несовместимости (Inc – incompatibility):
14 групп в системе Pseudomonas (IncP-1 – IncP-14)
30 групп в системе Enterobacteriaceae
Слайд 5Плазмиды
Контролируемый фенотип:
Устойчивость к антибиотикам, тяжелым металлам.
R – плазмиды
(resistance).
Деградация органических соединений.
D – плазмиды (degradative)
Конъюгационный перенос. F
– factor (fertility) Продукция токсических соединений (антибиотики, бактериоцины, колицины). ColE1 – плазмида (colicin)
Криптические плазмиды (фенотип неизвестен и плазмиды малого размера)
Взаимодействие с эукариотами. Ti – плазмиды (tumor inducing)
Другие свойства – устойчивость к UV, продукция H2S, чувствительность к NaCl, системы рестрикции – модификации, фиксация азота.
Слайд 6Конъюгация – процесс обмена генетической информацией между бактериальными клетками, обеспечиваемый
плазмидами, путем переноса генетического материала из клетки донора в клетку
реципиента.Трансконъюгант – бактериальная клетка, получившая генетический материал путем конъюгации.
Поверхностное исключение – конъюгационный перенос между плазмидосодержащими клетками происходит с меньшей на 2 порядка эффективностью, чем между донором и бесплазмидной клеткой. За этот эффект отвечают гены tra – системы (traS и traT).
Несовместимость плазмид (Inc) – неспособность двух разных плазмид стабильно сосуществовать в одной бактериальной клетке.
Донор – специфические фаги – фаги, инфицирующие только те штаммы, которые содержат конъюгативные плазмиды.
Число копий плазмиды – количество молекул плазмиды на бактериальный геном.
Строгий контроль репликации – плазмидная репликация связана с репликацией хромосомы.
Плазмиды
Слайд 7Обозначение плазмид
Префикс “p”
«Исторические» обозначения:
RSF1010, R100, NAH7.
R100 фенотип – Tc
Cm Sm Su Tra
Генотип – tet+cat+aadA+sul+tra+
Слайд 8Молекулярная организация плазмид
Базовый репликон (basic replicon):
ori (origin)
inc/cop - ген
(ы)
rep – ген
Жизненно – важные структуры плазмиды (backbone segment)
система разрешения
коинтегратов (mrs - multimer resolution system), система активного распределения плазмид (par – partitioning),
система постсегрегационной гибели клетки (PSK – post segregational killing).
система рестрикции – модификации (RM).
Система конъюгационного переноса.
Гены резистентности.
Гены биодеградации.
Другие гены.
Слайд 13Diversity of the Naphthalene Catabolic Plasmids Belonging to IncP-9 Group
on the Basis of RFLP Analysis
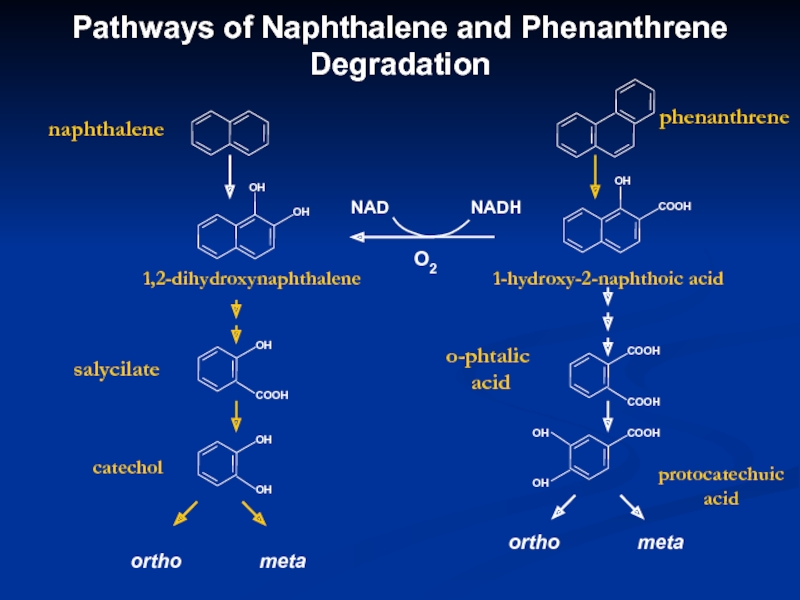
Слайд 17Pathways of Naphthalene and Phenanthrene Degradation
naphthalene
phenanthrene
1,2-dihydroxynaphthalene
1-hydroxy-2-naphthoic acid
NADН
salycilate
catechol
protocatechuic
acid
о-phtalic
acid
ortho
meta
ortho
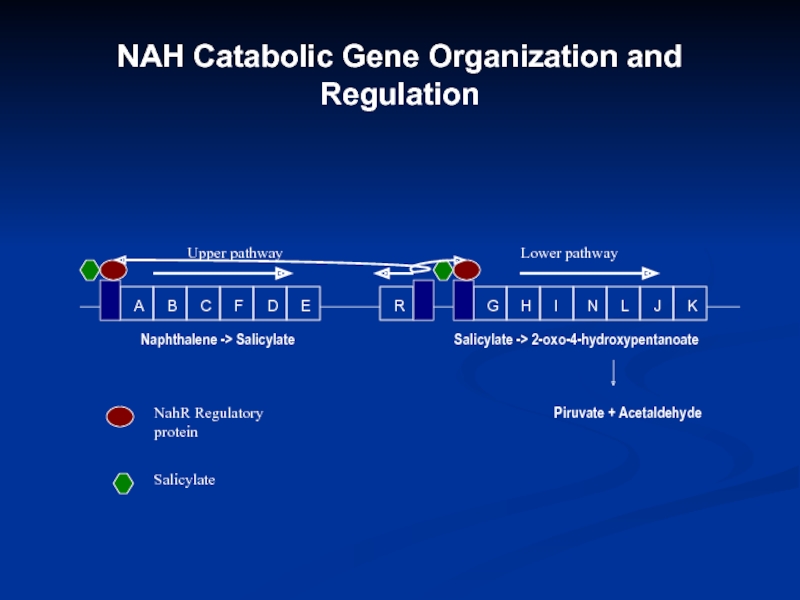
metaСлайд 18NAH Catabolic Gene Organization and Regulation
A
B
C
F
D
E
R
G
H
I
N
L
J
K
Upper pathway
Lower pathway
Piruvate + Acetaldehyde
Salicylate
-> 2-oxo-4-hydroxypentanoate
Naphthalene -> Salicylate
NahR Regulatory protein
Salicylate
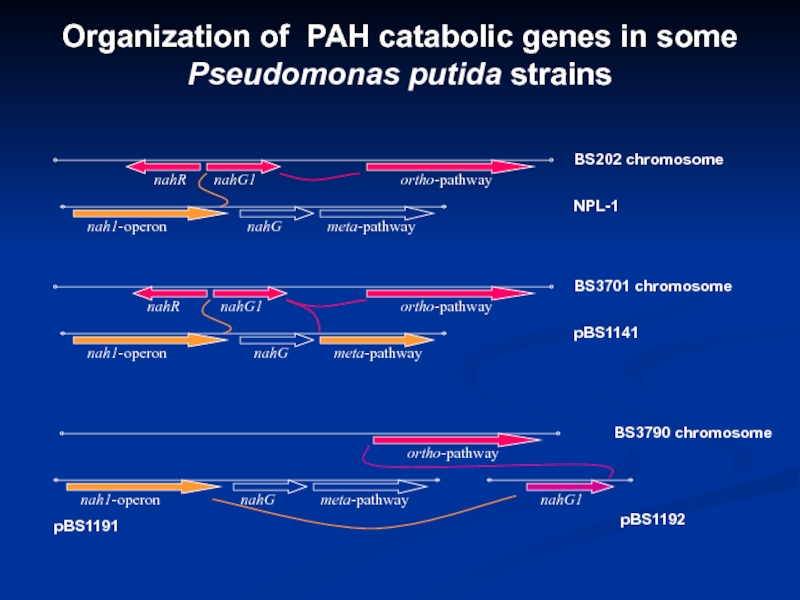
Слайд 20Organization of PAH catabolic genes in some Pseudomonas putida strains
nahG1
nahG
meta-pathway
nah1-operon
BS3790
chromosome
pBS1191
pBS1192
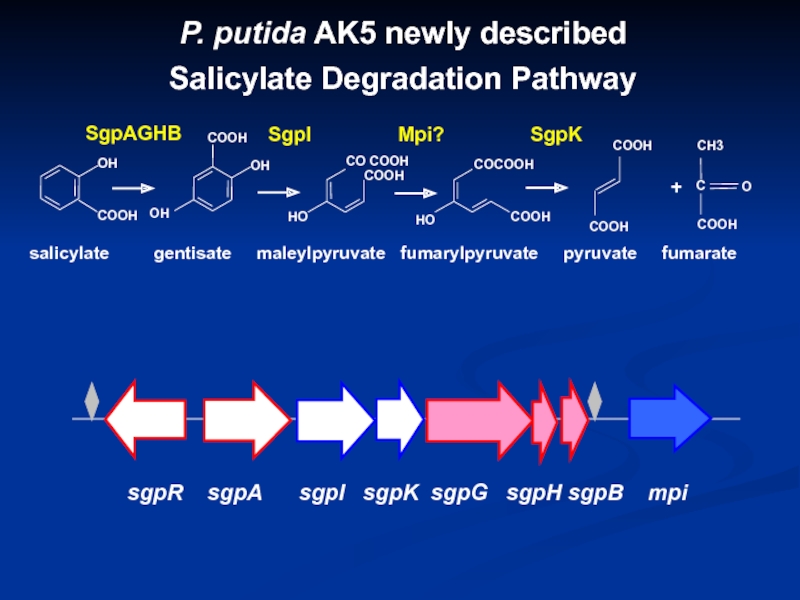
Слайд 23SgpI
Mpi?
SgpK
+
OH
COOH
salicylate gentisate maleylpyruvate
fumarylpyruvate pyruvate fumarate
СОOH
OH
OH
SgpAGHB
P. putida AK5
newly described Salicylate Degradation Pathway
Слайд 24Is there a degradative (D) “superplasmid” capable of determining the
most efficient degradation of a particular organic compound?
Is there a
bacterium which is the optimal host for that plasmid from the viewpoint of the expression of degradative genes?
Is there an “ideal” combination of a D-plasmid and a bacterial host?
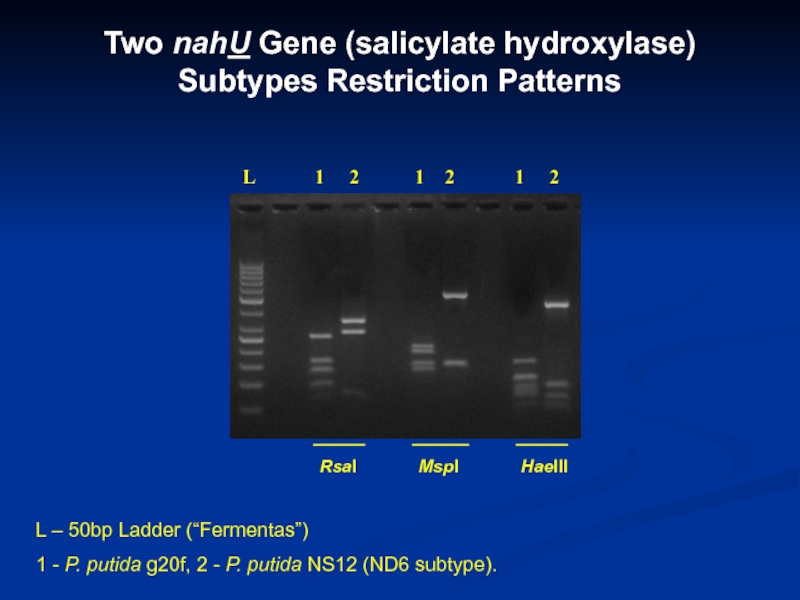
Слайд 27Two nahU Gene (salicylate hydroxylase) Subtypes Restriction Patterns
L – 50bp
Ladder (“Fermentas”)
1 - P. putida g20f, 2 - P. putida
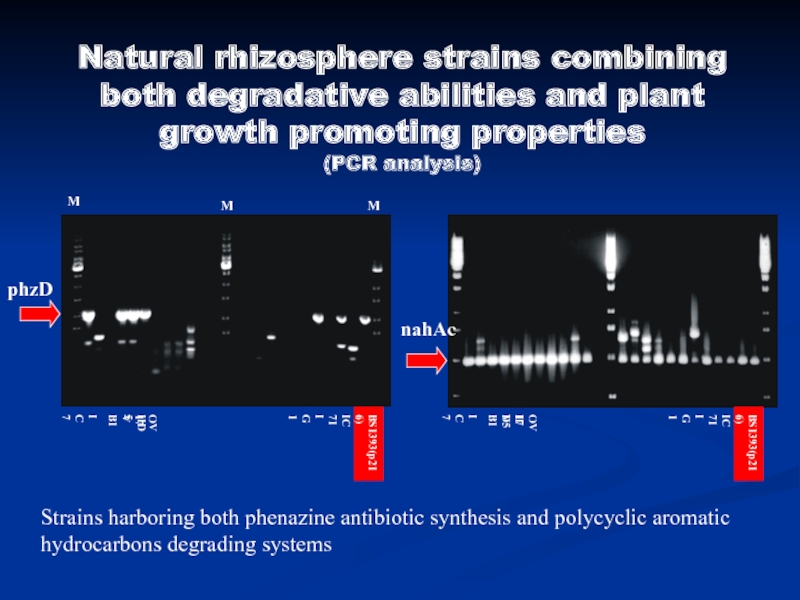
NS12 (ND6 subtype).Слайд 28Natural rhizosphere strains combining both degradative abilities and plant growth
promoting properties
(PCR analysis)
Strains harboring both phenazine antibiotic synthesis and polycyclic
aromatic hydrocarbons degrading systemsphzD
nahAc
M
IC7
VB1
M
M
IID5
OV17
IG1
IC71
BS1393(p216)
IC7
VB1
IID5
OV17
IG1
IC71
BS1393(p216)
Слайд 29Plant
PHYTOREMEDIATION
BIOREMEDIATION
Rhizosphere bacteria
Pseudomonas
DEGRADATION
OF TOXIC
ORGANIC
COMPOUNDS
ACCUMULATION
OF POLLUTANTS
Слайд 30Effect of naphthalene degradative plasmids on biosynthesis of phenazine antibiotics
by PGPR Pseudomonas
1 – P. fluorescenc 2-79, 2 - P.
fluorescenc 2-79(pBS216)3 – P. chlororaphis PCL1391, 4 - P. chlororaphis PCL1391(pBS216)
5 – P. aureofaciens 1217, P. aureofaciens 1217(pBS216)
1 - P. aureofaciens BS1393, 2 - P. aureofaciens BS1393(NAH7), 3 - P. aureofaciens BS1393(pBS216), 4 - P. aureofaciens BS1393(pBS3), 5 - P. aureofaciens BS1393(SAL)
1 – plasmid less variant of the P. aureofaciens strain OV17,
2 - P. aureofaciens OV17(pOV17),
3 - P. aureofaciens OV17(pBS216)
Слайд 32COMBINATION OF ABILITY TO UTILIZE POLYCYCLIC AROMATIC HYDROCARBONS AND RESISTANCE TO
ARSENIC COMPOUNDS
pBS3031(AsR)
Pseudomonas aeruginosa BS3031(pBS3031)
Pseudomonas putida BS238(pBS2)
Pseudomonas putida BS238(pBS2)
Pseudomonas putida BS238(pBS2+pBS3031)
Growth
on naphthaleneСлайд 34Extreme Environmental Factors
Toxic chemical agents
Heavy metals
Radionucleides
UV-light
Low or high pH values
High
salinity
Water deficiency
Oxygen deficiency
Слайд 40The effect of crude oil hydrocarbons on microbial processes in
soils, providing CO2 emission into atmosphere, has been studied. The
total CO2 emission from oil-containing soil samples to atmosphere during 47 days of observation was 6.8-fold more then from native soil. At the same time, the amount of metabolic CO2 produced due to soil organic matter mineralization was about 38 % of the total CO2 flow and that due to utilization of oil hydrocarbons reached 62 %.Слайд 41The History of Bacterial Genetics (S.E. Luria, 1968)
The Stone Age
or the Luria-Delbrück Age (1943-1946)
The Bronze or Lederberg Age (1946-1953)
The
Golden or Hayes-Wollman-Jacob Age (1953-1961)The Desperate Age, which is the present one, in which lots of things happen, lots of biochemists and ultracentrifugologists appear, and the philosophers get desperate.