Разделы презентаций
- Разное
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Геометрия
- Детские презентации
- Информатика
- История
- Литература
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Эволюционные модели и дистанции между последовательностями
Содержание
- 1. Эволюционные модели и дистанции между последовательностями
- 2. Слайд 2
- 3. BEAST Software Bayesian Evolutionary Analysis Sampling TreesПрограммы
- 4. Урок 1: Использование последовательностей сэмплированные в различные
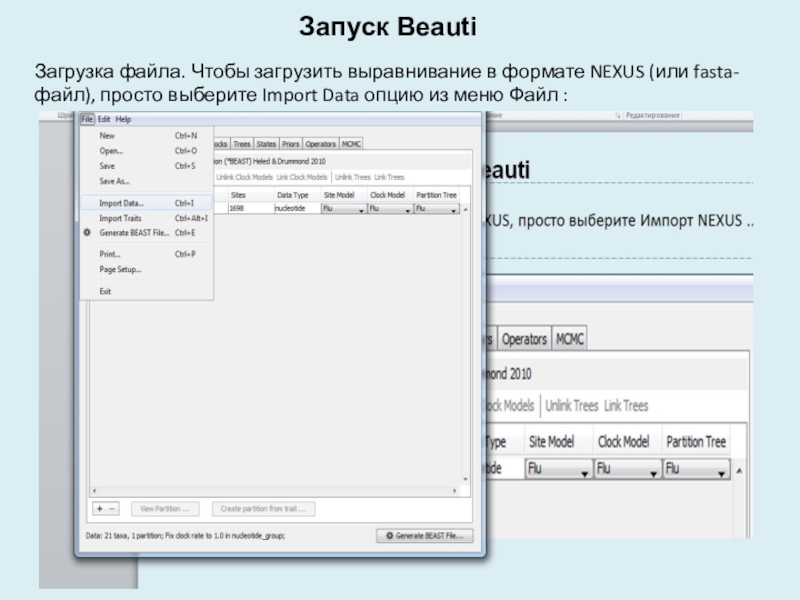
- 5. Запуск BeautiЗагрузка файла. Чтобы загрузить выравнивание в
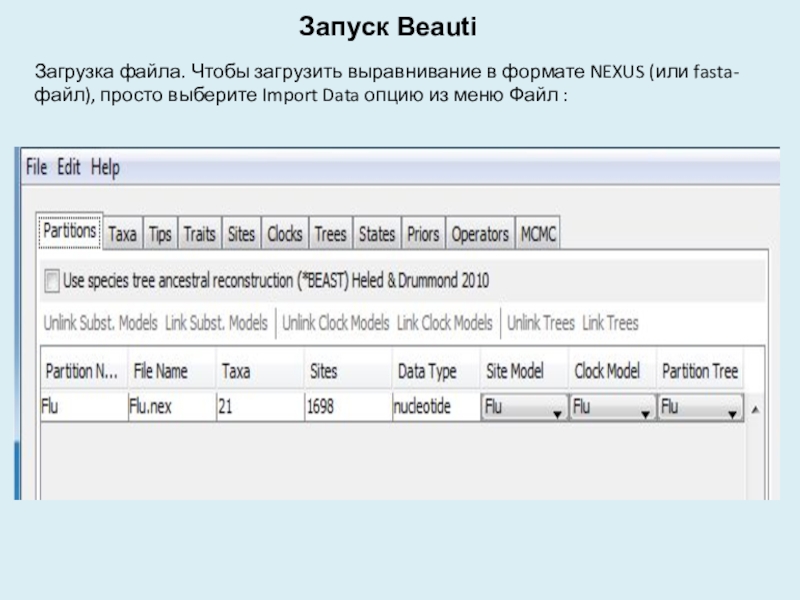
- 6. Запуск BeautiЗагрузка файла. Чтобы загрузить выравнивание в
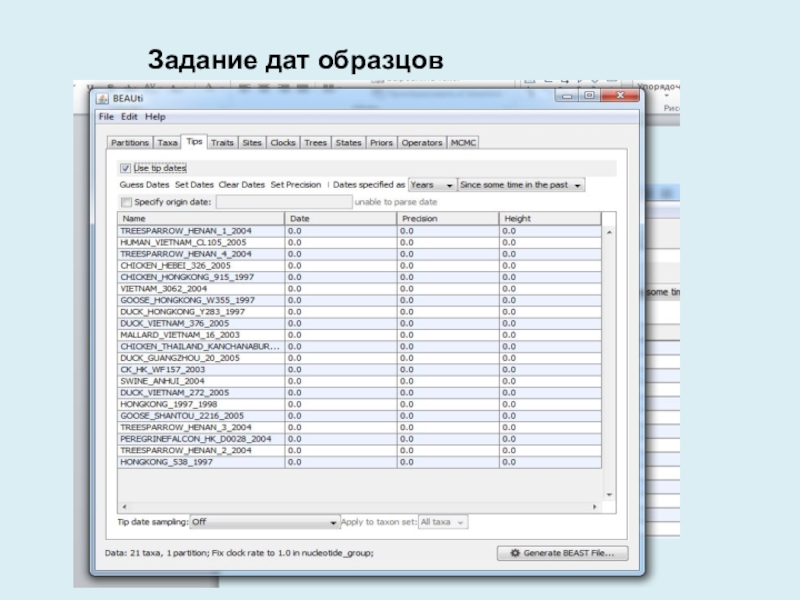
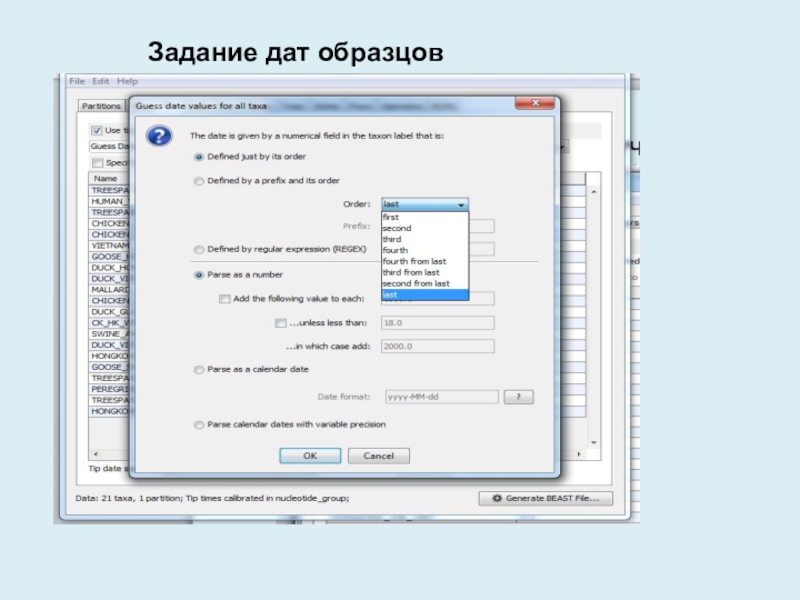
- 7. Задание дат образцов
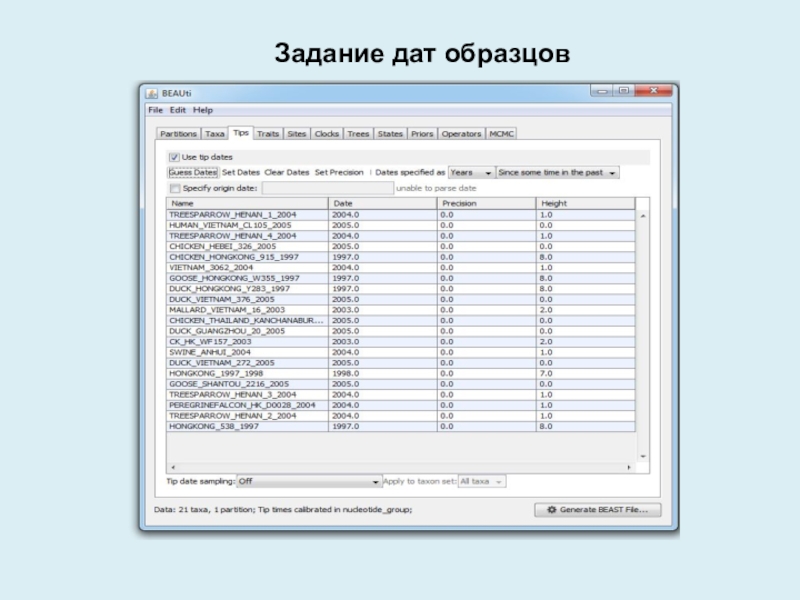
- 8. Задание дат образцов
- 9. Задание дат образцов
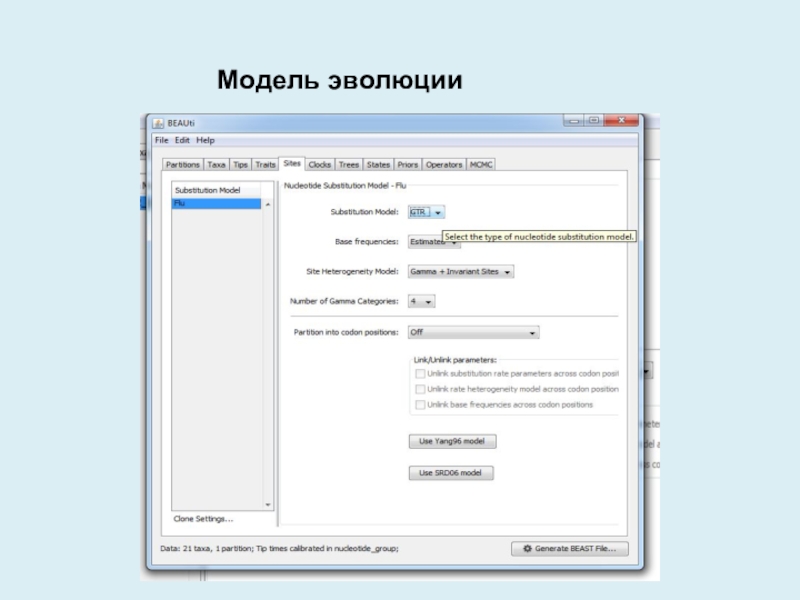
- 10. Модель эволюции
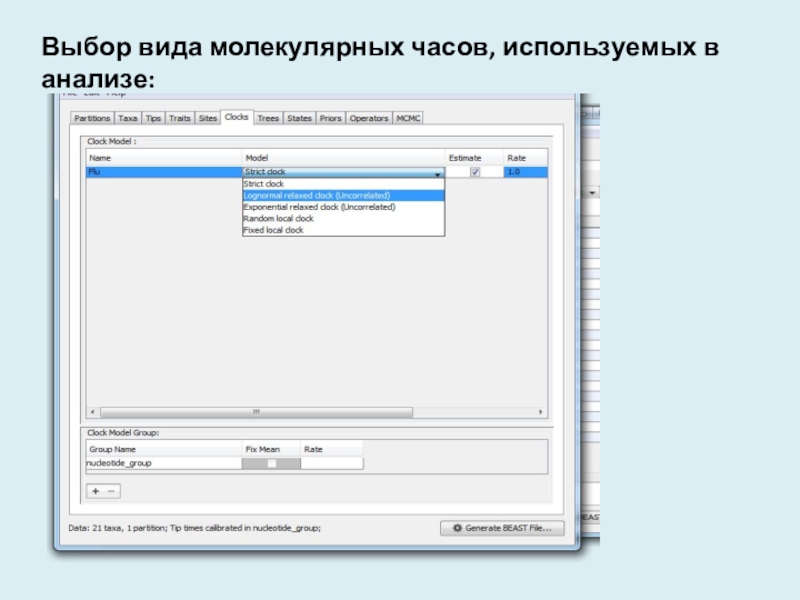
- 11. Выбор вида молекулярных часов, используемых в анализе:
- 12. BEAST предлагает ряд предварительных распределений для моделирования
- 13. Выбор параметров анализа:
- 14. Слайд 14
- 15. Слайд 15
- 16. Создаем XML (Beast) файл Генерация XML файла
- 17. Запуск Beast
- 18. Запуск Beast
- 19. Для изучения дает ли адекватный результат конкретная
- 20. Слайд 20
- 21. Или в случае достаточного объема выборки (ESS):
- 22. Mean Среднее (исключая burn-in).Stdev of mean Стандартная
- 23. Слайд 23
- 24. Мы получили большую выборку филогенетических деревьев BEAST.
- 25. Слайд 25
- 26. Слайд 26
- 27. Просмотр дереваИ, наконец, мы можем посмотреть на
- 28. Слайд 28
- 29. Слайд 29
- 30. Слайд 30
- 31. Coalescent: GMRF Bayesian SkyrideЭта модель предполагает фиксированное
- 32. https://rega.kuleuven.be/cev/ecv/software/SpreaD3_tutorialSpreaD3
- 33. https://rega.kuleuven.be/cev/ecv/software/SpreaD3_tutorialSpreaD3Epidemiological statistics estimated for Foot-and-mouth disease (Serotype
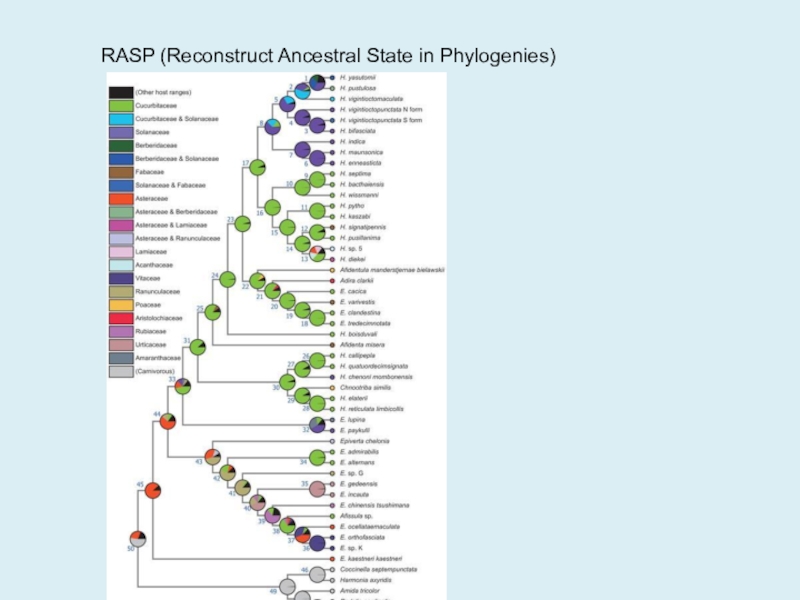
- 34. RASP (Reconstruct Ancestral State in Phylogenies)
- 35. Для предсказания вторичной структуры ДНК и РНК
- 36. Слайд 36
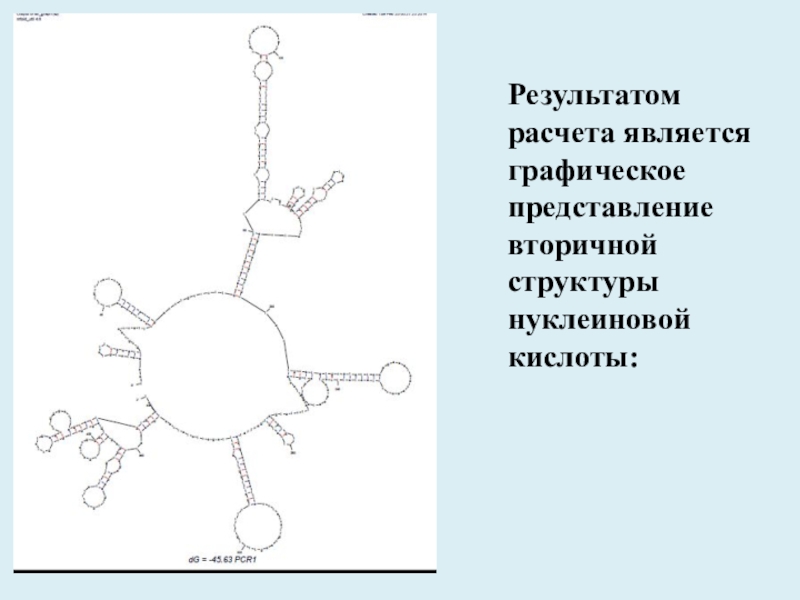
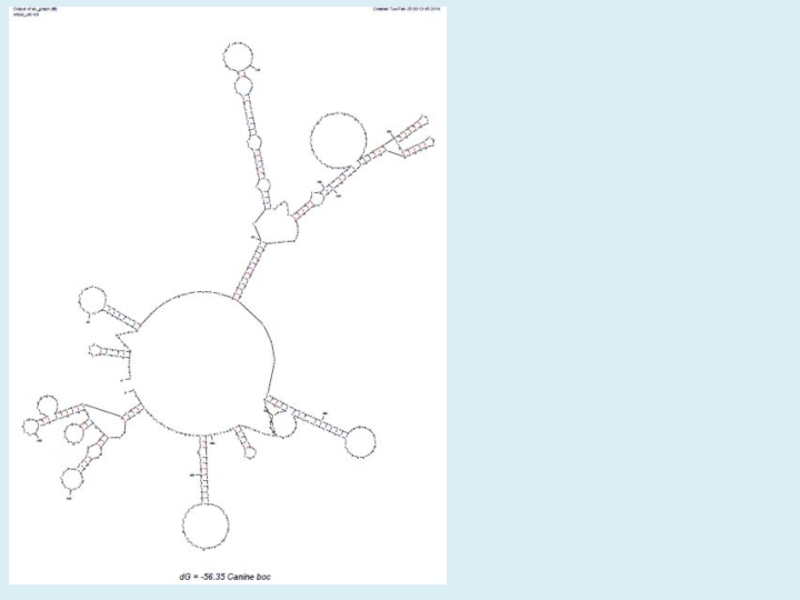
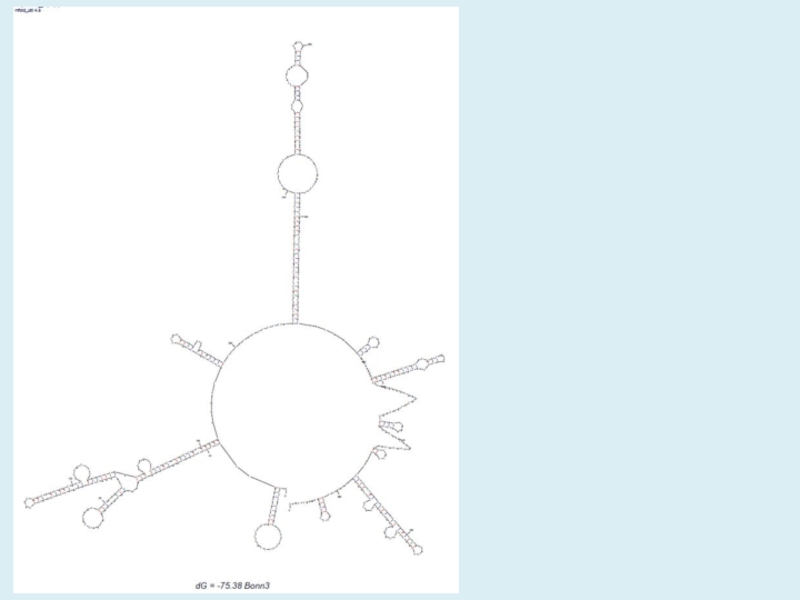
- 37. Результатом расчета является графическое представление вторичной структуры нуклеиновой кислоты:
- 38. Слайд 38
- 39. Слайд 39
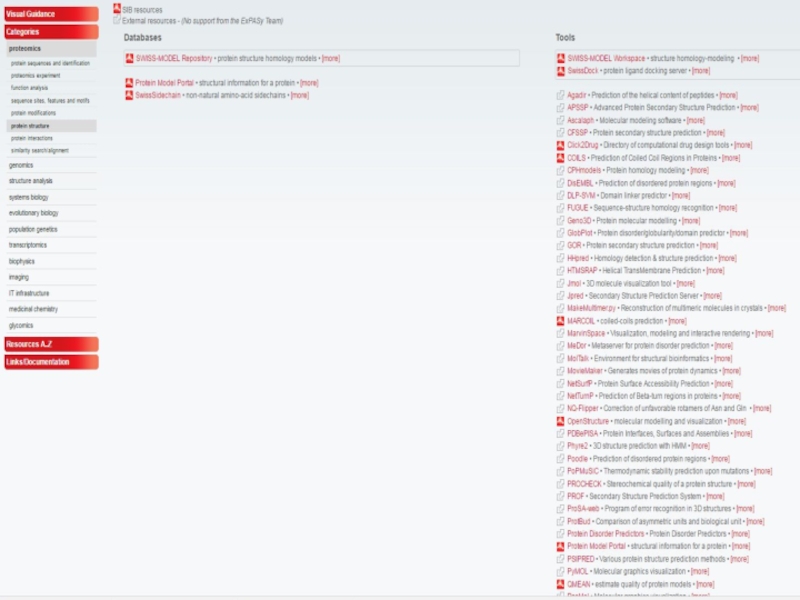
- 40. Предсказание свойств и структуры белковБольшое количество такого рода программ представлено на ресурсе http://www.expasy.org/:
- 41. Слайд 41
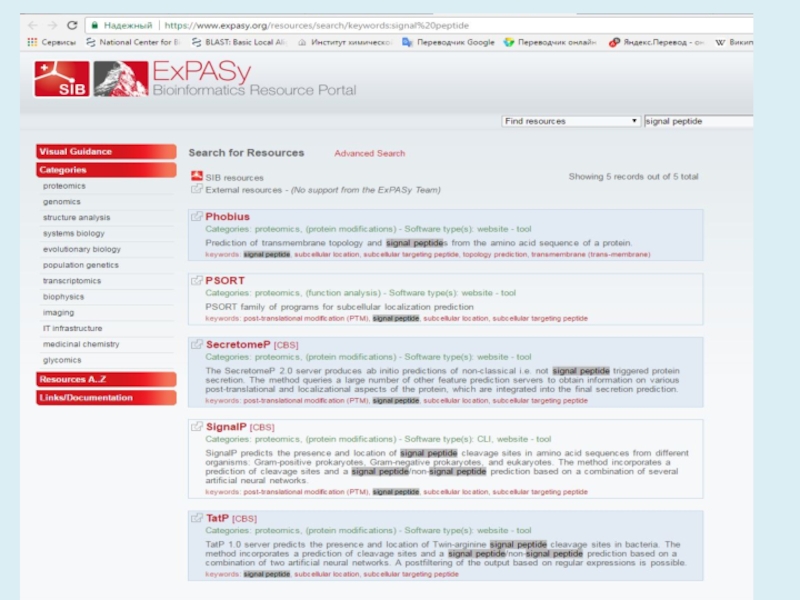
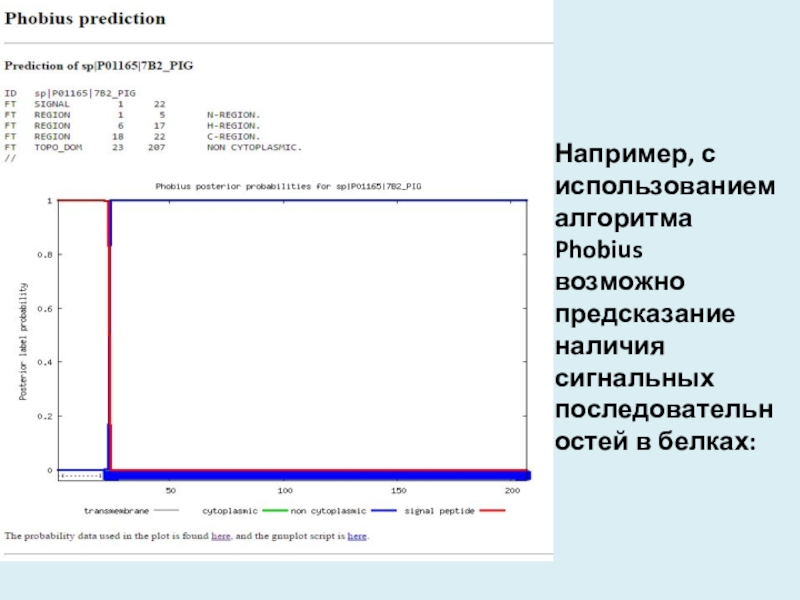
- 42. Например, с использованием алгоритма Phobius возможно предсказание наличия сигнальных последовательностей в белках:
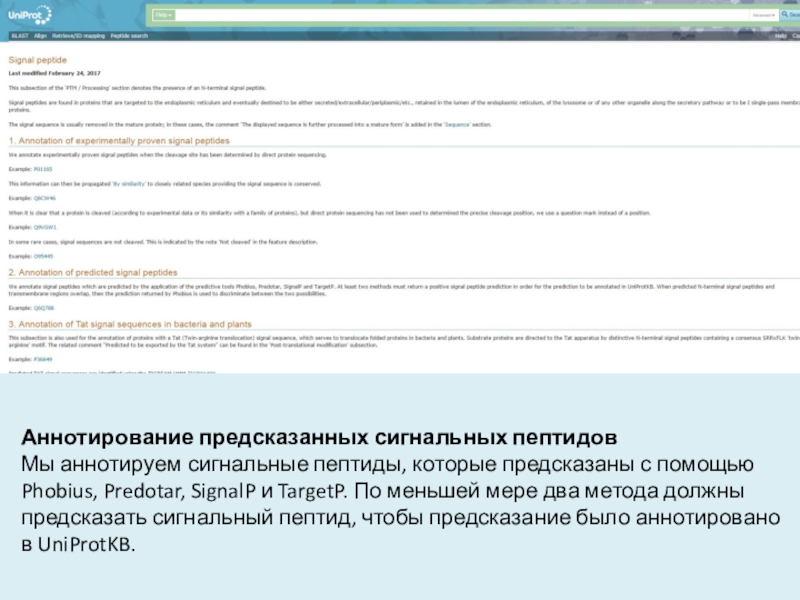
- 43. Аннотирование предсказанных сигнальных пептидовМы аннотируем сигнальные пептиды,
- 44. Слайд 44
- 45. Слайд 45
- 46. Скачать презентанцию
Слайды и текст этой презентации
Слайд 1Эволюционные модели и дистанции между последовательностями биополимеров
Молекулярная филогения
Статистические методы
оценки деревьев. Анализ молекулярных часов.
Слайд 3BEAST Software
Bayesian Evolutionary Analysis Sampling Trees
Программы поставляемые в составе
пакета BEAST:
BEAST Bayesian Evolutionary Analysis Sampling Trees
BEAUti простой пользовательский интерфейс для создания
входных файлов для запуска BEAST.LogCombiner Программа для объединения log и tree файлов из нескольких запусков BEAST
TreeAnnotator A program for summarizing the information in a sample of trees produced by BEAST Программа для обобщения информации, в единое дерево
Программы не поставляемые в составе пакета BEAST:
Tracer Программа для анализа результатов программ байесовских MCMC, таких как BEAST & MrBayes
FigTree программа для просмотра деревьев, включая обобщенную информацию, производимую TreeAnnotator
http://beast.bio.ed.ac.uk
/
beast.bio.ed.ac.uk
Слайд 4Урок 1: Использование последовательностей сэмплированные в различные периоды времени, чтобы
оценить темпы эволюции
Урок 2: Оценка времени дивергенции монофилетической группы с
использованием известной частоты мутацийСлайд 5Запуск Beauti
Загрузка файла. Чтобы загрузить выравнивание в формате NEXUS (или
fasta-файл), просто выберите Import Data опцию из меню Файл :
Слайд 6Запуск Beauti
Загрузка файла. Чтобы загрузить выравнивание в формате NEXUS (или
fasta-файл), просто выберите Import Data опцию из меню Файл :
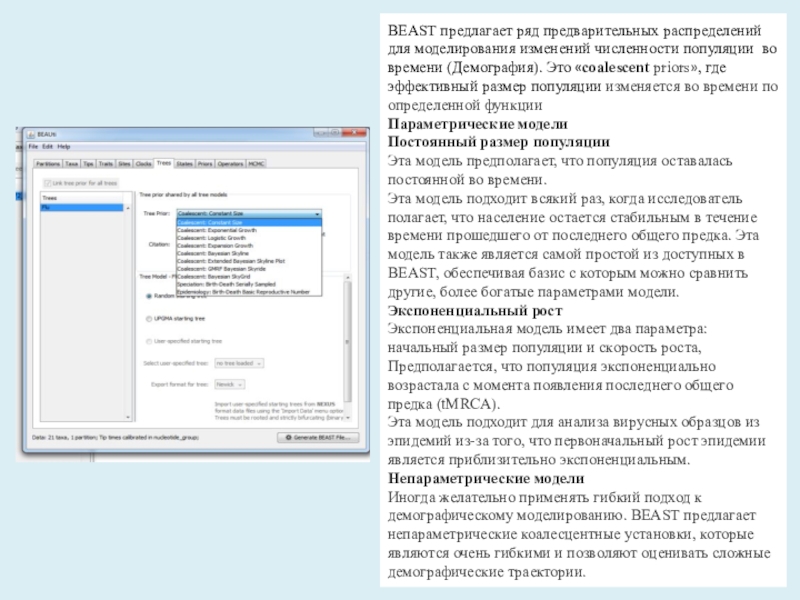
Слайд 12BEAST предлагает ряд предварительных распределений для моделирования изменений численности популяции
во времени (Демография). Это «coalescent priors», где эффективный размер популяции изменяется во времени по
определенной функции Параметрические моделиПостоянный размер популяции
Эта модель предполагает, что популяция оставалась постоянной во времени.
Эта модель подходит всякий раз, когда исследователь полагает, что население остается стабильным в течение времени прошедшего от последнего общего предка. Эта модель также является самой простой из доступных в BEAST, обеспечивая базис с которым можно сравнить другие, более богатые параметрами модели.
Экспоненциальный рост
Экспоненциальная модель имеет два параметра: начальный размер популяции и скорость роста, Предполагается, что популяция экспоненциально возрастала с момента появления последнего общего предка (tMRCA).
Эта модель подходит для анализа вирусных образцов из эпидемий из-за того, что первоначальный рост эпидемии является приблизительно экспоненциальным.
Непараметрические модели
Иногда желательно применять гибкий подход к демографическому моделированию. BEAST предлагает непараметрические коалесцентные установки, которые являются очень гибкими и позволяют оценивать сложные демографические траектории.
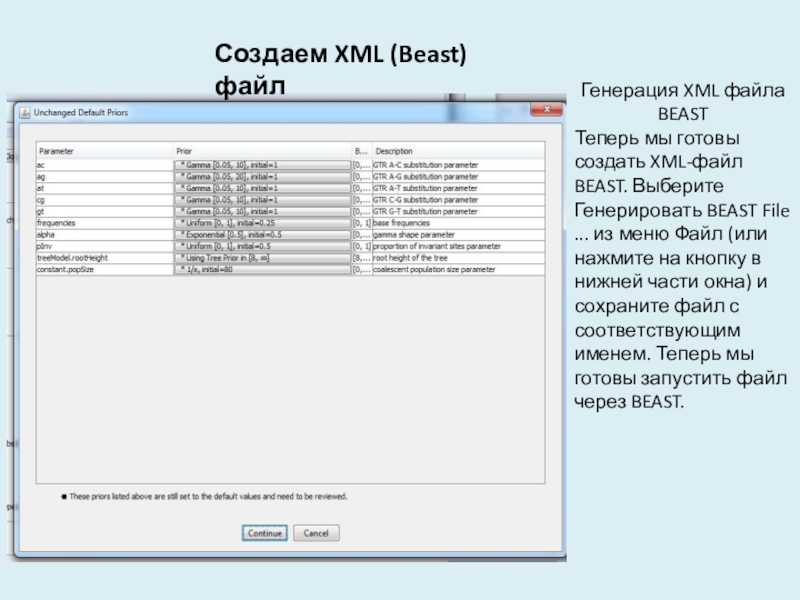
Слайд 16Создаем XML (Beast) файл
Генерация XML файла BEAST
Теперь мы готовы
создать XML-файл BEAST. Выберите Генерировать BEAST File ... из меню Файл
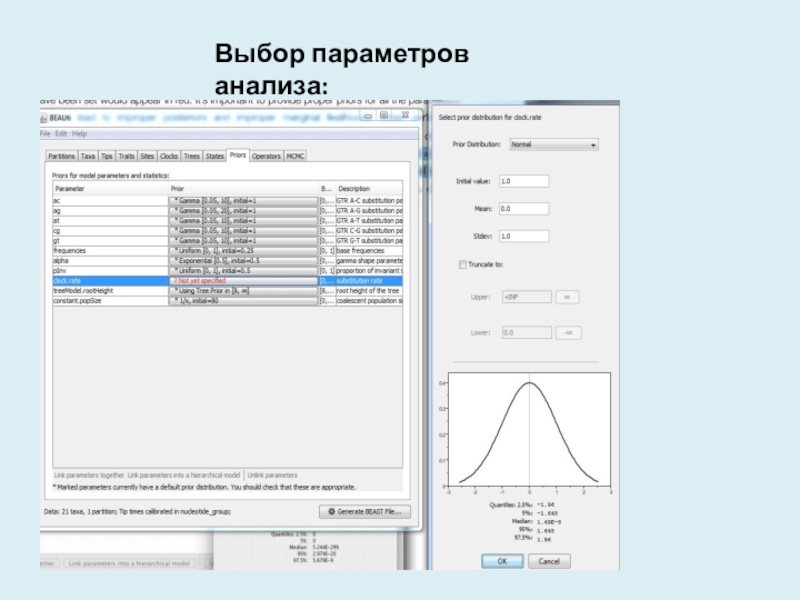
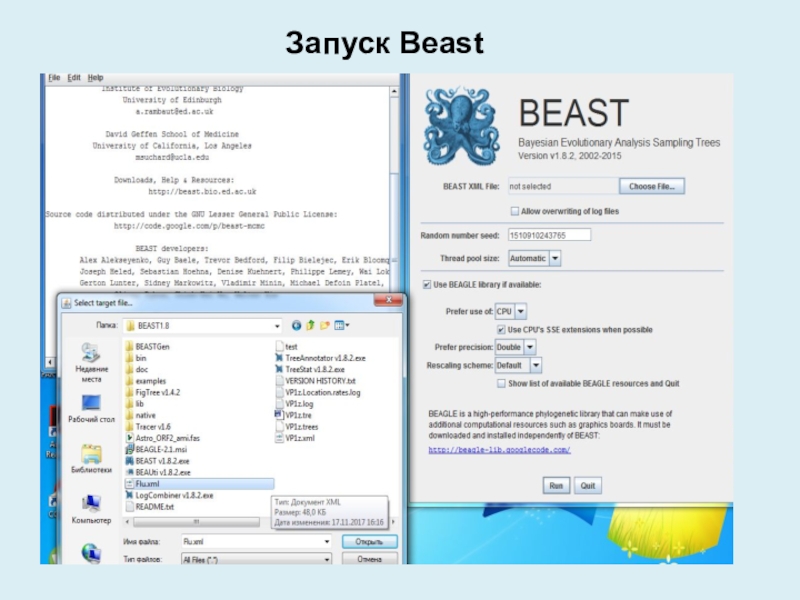
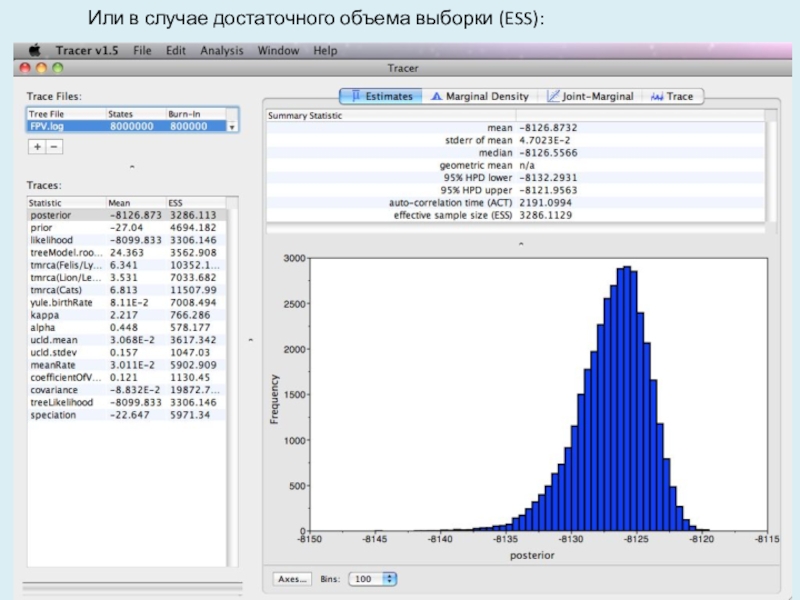
(или нажмите на кнопку в нижней части окна) и сохраните файл с соответствующим именем. Теперь мы готовы запустить файл через BEAST.Слайд 19Для изучения дает ли адекватный результат конкретная длина цепи проведенного
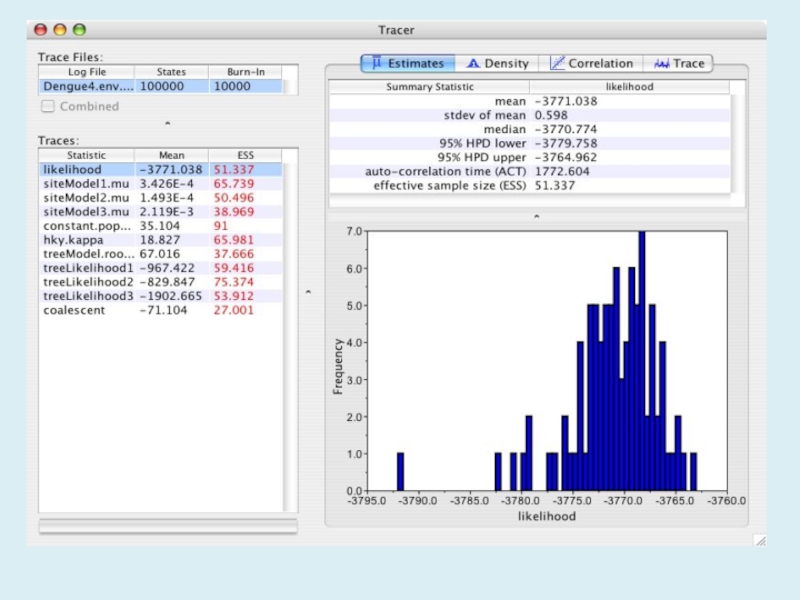
анализа, полученный log-файл можно анализировать с помощью Tracer. Цель установки
длины цепи является достижение разумного эффективного объема выборки (ESS).Анализ результатов
Запустите программу под названием Tracer для анализа результатов BEAST . Когда главное окно открылось, выберите Импорт файла трассировки ... в меню Файл и выберите файл, который BEAST создал называется FPV.log.txt. Теперь вы должны увидеть окно, похожее на следующее (В случае недостаточного объема выборки (ESS)):
Слайд 22Mean Среднее (исключая burn-in).
Stdev of mean Стандартная ошибка среднего. Она
учитывает эффективный размер выборки, поэтому небольшая ESS даст большую стандартную
ошибку.Median Медиана (исключая burn-in).
Geometric mean Центральная тенденция или типичное значение набора образцов (исключая burn-in).
95% HPD Lower Нижняя граница доверительного интервала (HPD). HPD - это самый короткий интервал, который содержит 95% значений.
95% HPD Upper Верхняя граница доверительного интервала (HPD).
Effective Sample Size (ESS) Эффективный размер выборки
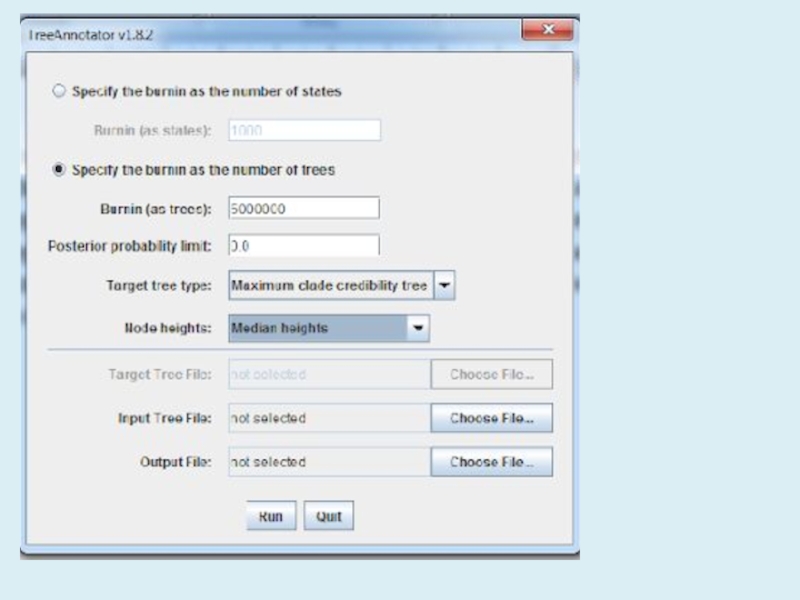
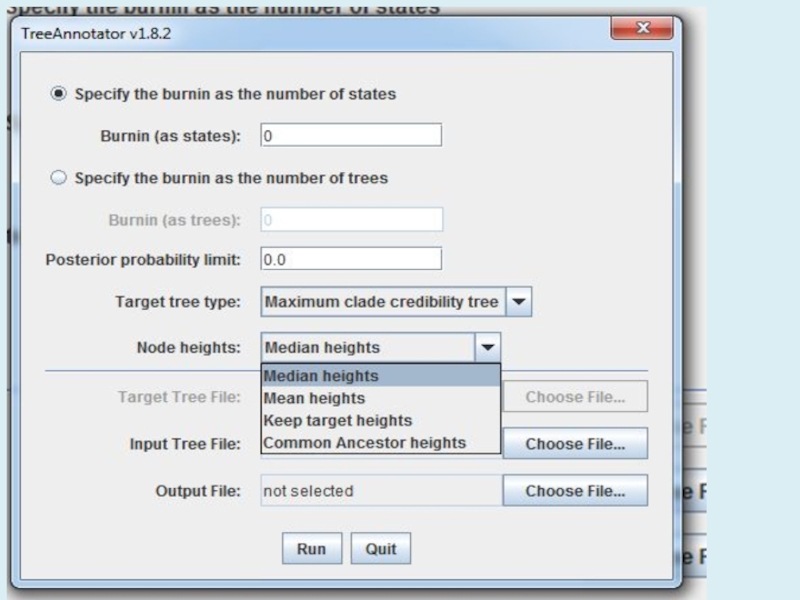
Слайд 24Мы получили большую выборку филогенетических деревьев BEAST. Их необходимо суммировать
с помощью программы TreeAnnotator. Она найдет дерево с наилучшей поддержкой.
Она также вычислит значения вероятности для каждого узла.TreeAnnotator создает одно целевое дерево и аннотирует его суммированной информацией из всех деревьев.
Запустите программу TreeAnnotator и настройте ее так:
Слайд 27Просмотр дерева
И, наконец, мы можем посмотреть на дерево в другой
программе под названием Figtree.
В результате мы видим хронограмму, отражающую время
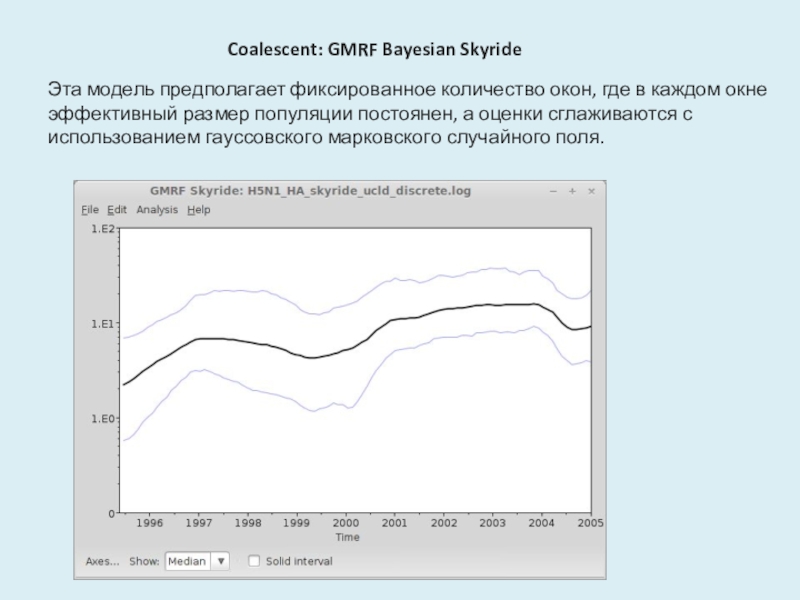
происхождения исследуемых видов от общего предка. Слайд 31Coalescent: GMRF Bayesian Skyride
Эта модель предполагает фиксированное количество окон, где
в каждом окне эффективный размер популяции постоянен, а оценки сглаживаются
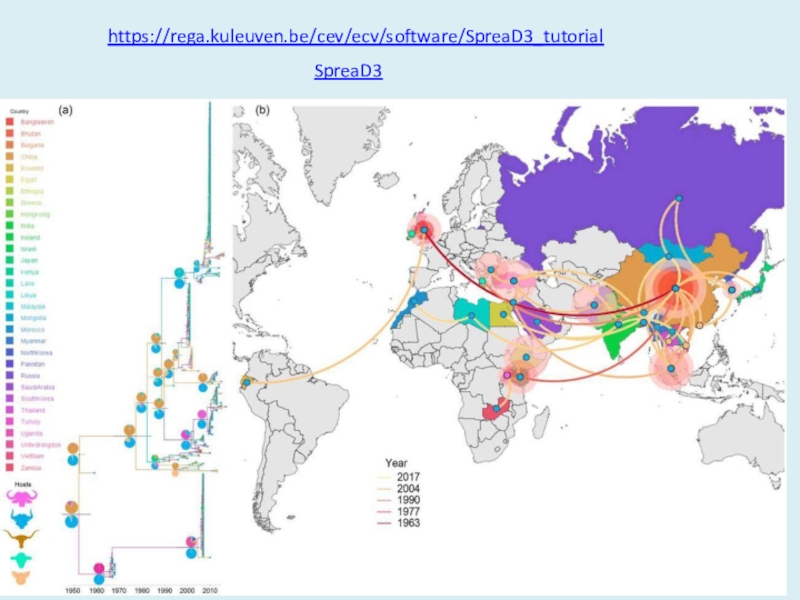
с использованием гауссовского марковского случайного поля.Слайд 33https://rega.kuleuven.be/cev/ecv/software/SpreaD3_tutorial
SpreaD3
Epidemiological statistics estimated for Foot-and-mouth disease (Serotype A) epidemic. (a)
Maximum clade credibility phylogeny of the FMDV epidemic colored according
to the countries where the disease has been registered. Dots at the end of the branches represent the host species (buffalo, cattle, sheep, and swine). The probabilities of ancestral states (inferred from the Bayesian MCMC discrete traits analysis) are shown in pie charts at each node, where charts above branches represent locations and charts below branches most likely hosts. (b) Reconstructed spatiotemporal diffusion of FMD serotype A spread, where diameters of the colored circles are proportional to the square root of the number of MCC branches maintaining a particular location state at each time period. The color of the branches represents the age of the internal nodes, where darker red colors represent older spread events.Слайд 35Для предсказания вторичной структуры ДНК и РНК в большинстве случаев
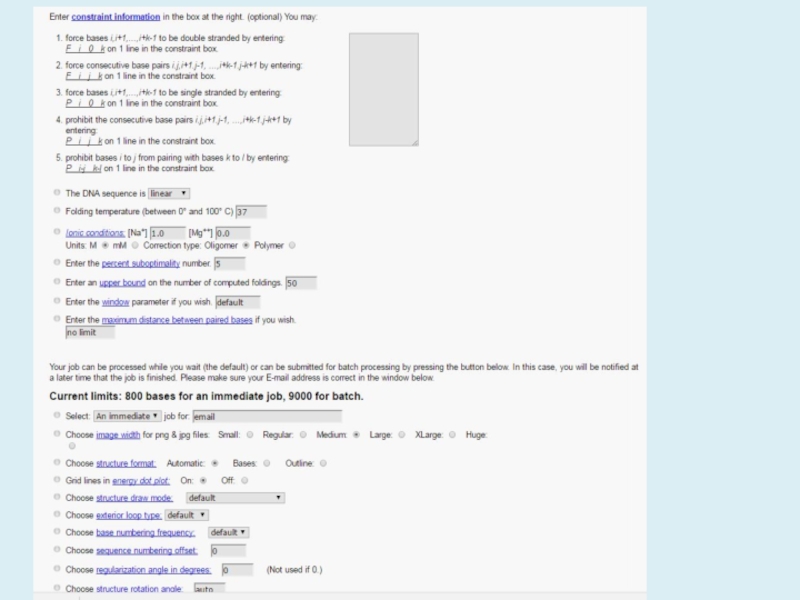
используется The mfold Web Server (http://unafold.rna.albany.edu/?q=mfold/DNA-Folding-Form).
Данная программа отображает вторичные структуры одноцепочечных нуклеиновых
кислот определяет их свободную энергию, которая является критерием их стабильности. Текущие ограничения программы: 800 нуклеотидов для немедленной работы, 9000 для пакетного режима.Окно программы выглядит следующим образом:
Предсказание вторичной структуры ДНК и РНК