Разделы презентаций
- Разное
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Геометрия
- Детские презентации
- Информатика
- История
- Литература
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Биоинформатические подходы к анализу РНК. Экспрессия генов: анализ
Содержание
- 1. Биоинформатические подходы к анализу РНК. Экспрессия генов: анализ
- 2. Кафедра биоинформатики МБФ РНИМУМиллер с коллегами (1970,
- 3. Кафедра биоинформатики МБФ РНИМУВ то время как
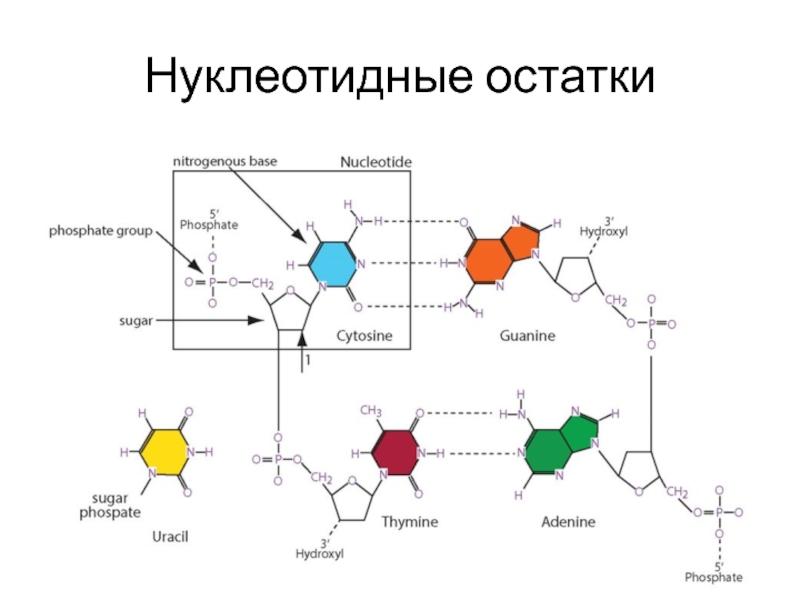
- 4. Нуклеотидные остаткиКафедра биоинформатики МБФ РНИМУ
- 5. Некодирующие РНККафедра биоинформатики МБФ РНИМУНесколько ключевых дат:1993
- 6. Некодирующие РНК (нкРНК)Кафедра биоинформатики МБФ РНИМУБольшое число
- 7. Малые некодирующие РНККафедра биоинформатики МБФ РНИМУНаибольшее функциональное
- 8. Кафедра биоинформатики МБФ РНИМУ
- 9. Кафедра биоинформатики МБФ РНИМУ
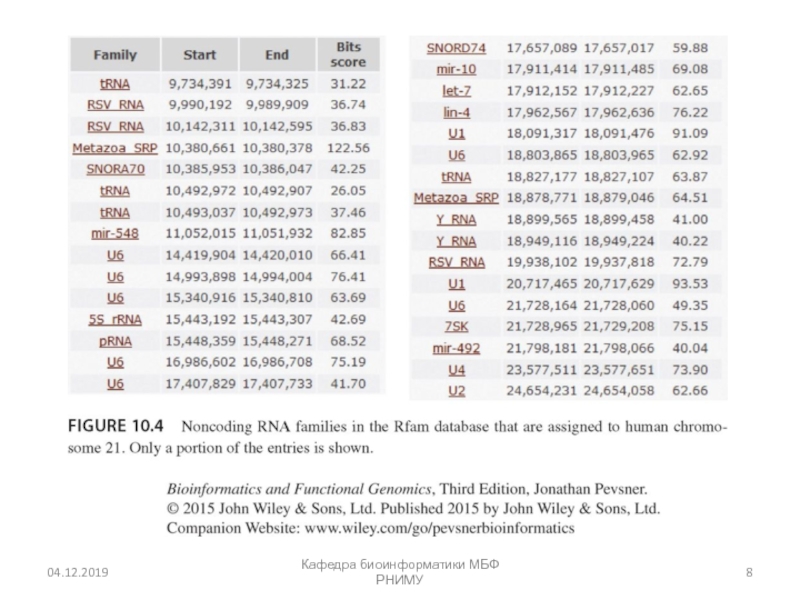
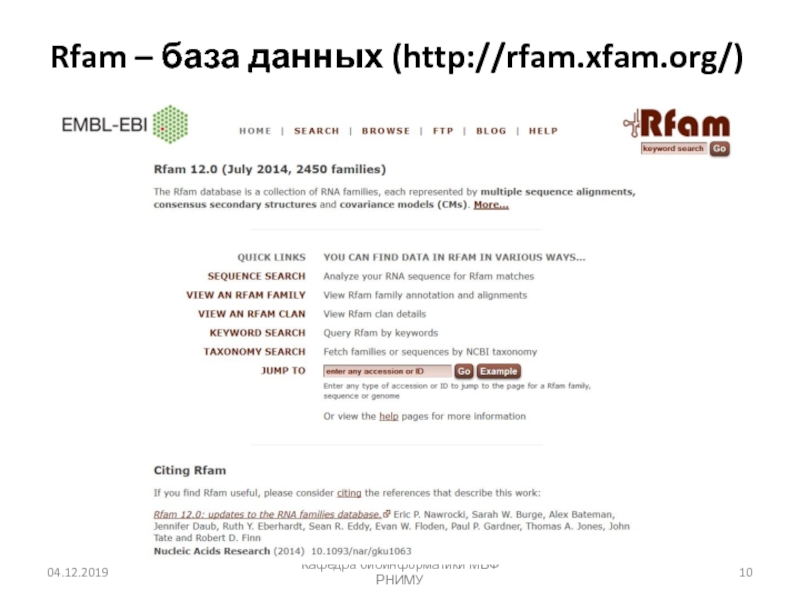
- 10. Rfam – база данных (http://rfam.xfam.org/) Кафедра биоинформатики МБФ РНИМУ
- 11. Некодируещие РНККафедра биоинформатики МБФ РНИМУ
- 12. Некодируещие РНККафедра биоинформатики МБФ РНИМУ
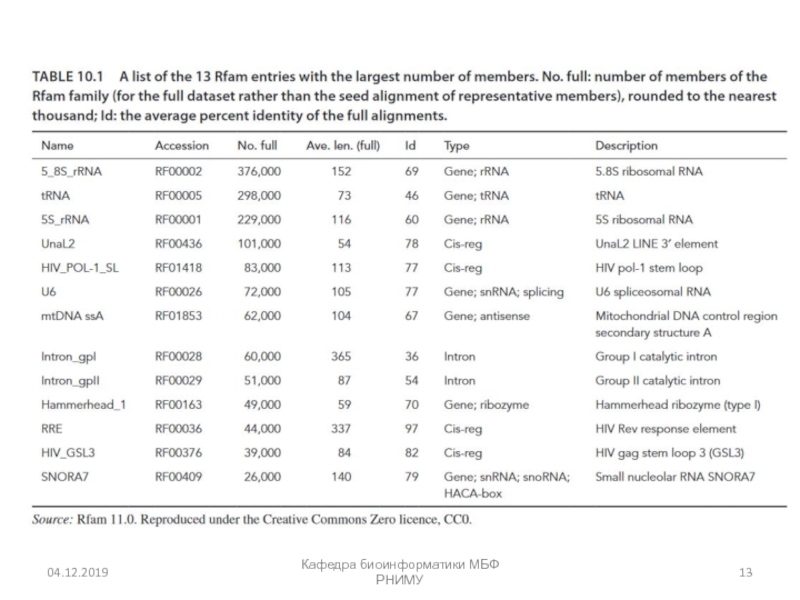
- 13. Кафедра биоинформатики МБФ РНИМУ
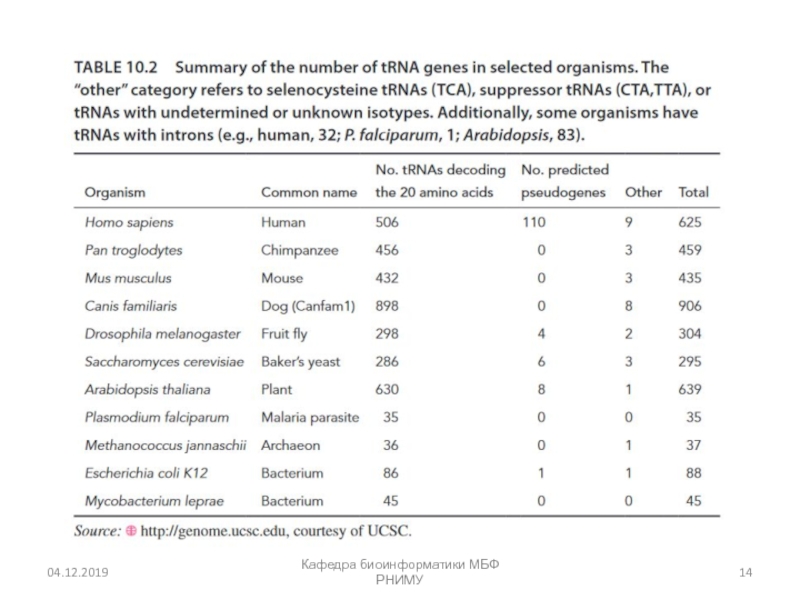
- 14. Кафедра биоинформатики МБФ РНИМУ
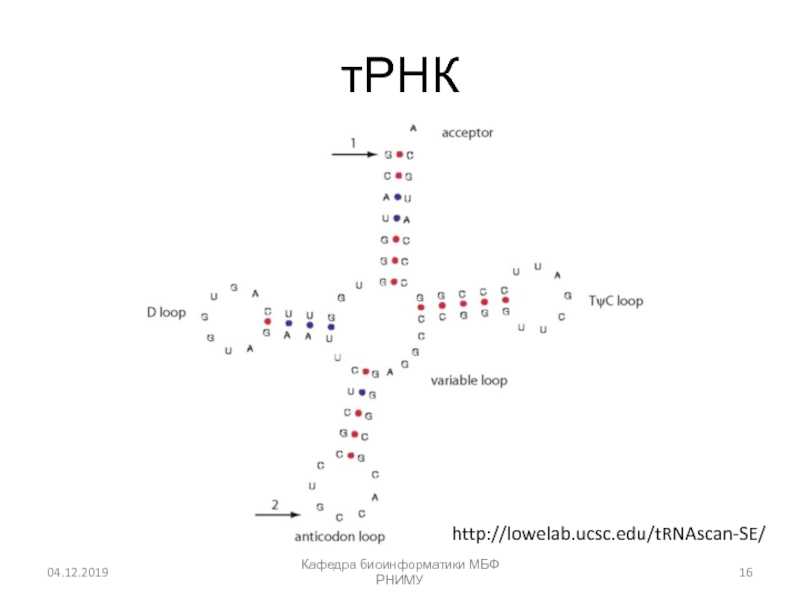
- 15. тРНККафедра биоинформатики МБФ РНИМУhttp://lowelab.ucsc.edu/tRNAscan-SE/
- 16. тРНККафедра биоинформатики МБФ РНИМУhttp://lowelab.ucsc.edu/tRNAscan-SE/
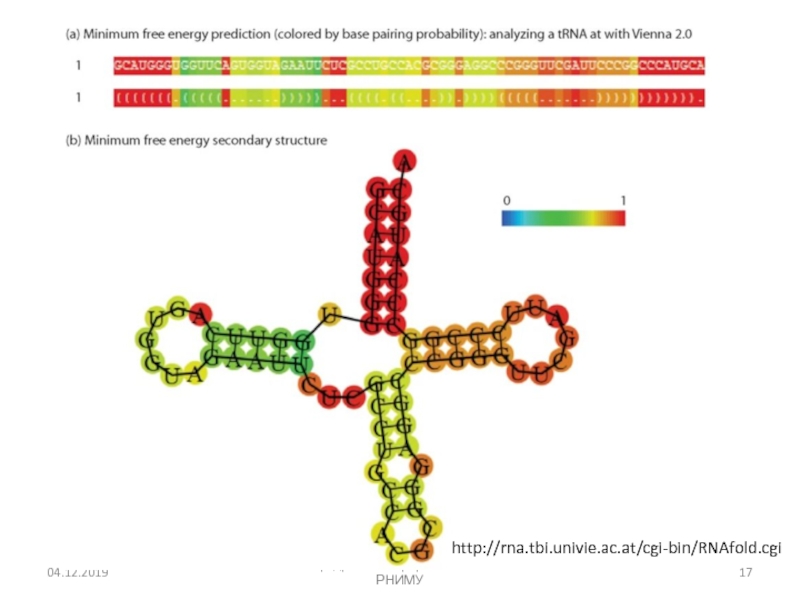
- 17. Кафедра биоинформатики МБФ РНИМУhttp://rna.tbi.univie.ac.at/cgi-bin/RNAfold.cgi
- 18. Экспрессия генов: анализ микроэрейных данныхЭкспрессия геновМикрочипы (Microarrays)Предварительная обработканормализациядиаграммы рассеянияСтатистический анализТ-тестANOVAрасстояниякластеризацияанализ главных компонентов (PCA)Кафедра биоинформатики МБФ РНИМУ
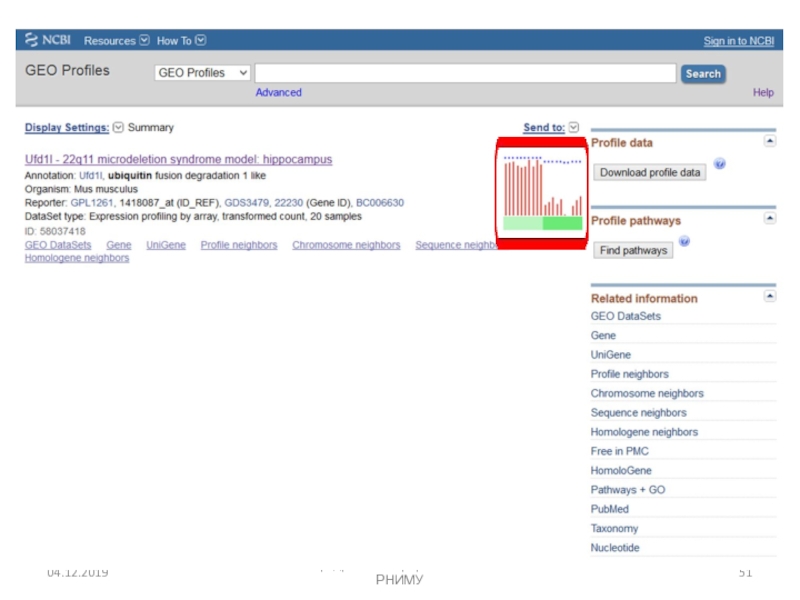
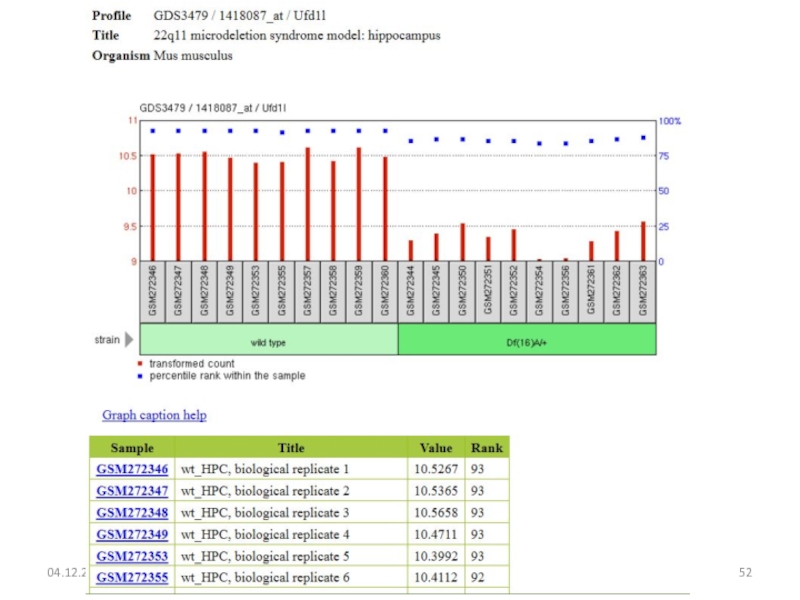
- 19. Кафедра биоинформатики МБФ РНИМУСравнение экспрессии генов в
- 20. Экспрессия генов зависит от контекста, и регулируется
- 21. Кафедра биоинформатики МБФ РНИМУDNAРНКcDNAбелокDNAРНКcDNAбелокUniGeneSAGEmicroarraynext-generation sequencing!!!
- 22. UniGene: уникальные гены представленные ESTsUnigene в
- 23. Кафедра биоинформатики МБФ РНИМУ
- 24. Кафедра биоинформатики МБФ РНИМУ
- 25. Слайд 25
- 26. Microarrays: инструмент для измерения экспрессии геновмикрочипом
- 27. Кафедра биоинформатики МБФ РНИМУMicroarrays: инструмент для
- 28. Преимущества микроэррейных экспериментовСкорость: данные о > 20000
- 29. Недостатки микроэррейных экспериментовЦена: Некоторые исследователи не могут
- 30. Кафедра биоинформатики МБФ РНИМУЭтап 1: Экспериментальный дизайнЭтап
- 31. Этап 1: Экспериментальный дизайн[1] Биологические образцы: технические
- 32. Один образец на массив (Affymetrix или
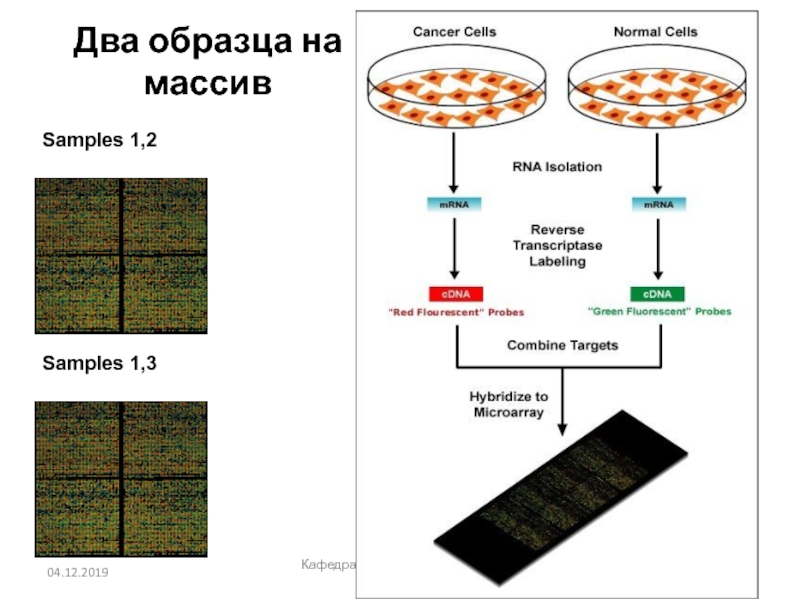
- 33. Два образца на массивКафедра биоинформатики МБФ РНИМУSamples 1,2Samples 1,3
- 34. Этап 2: РНК и пробо подготовкаДля Affymetrix
- 35. Этап 3: Гибридизация с ДНК чипомМассив состоит
- 36. Поверхность чипаКафедра биоинформатики МБФ РНИМУ
- 37. Этап 4: Анализ изображенийУровни РНК-транскриптов являются количественнымиИнтенсивность
- 38. Дифференциальная генная экспрессия на кДНК микроэррее
- 39. Этап 5: Анализ микроэррейных данныхПроверка гипотезыКак можно
- 40. Кафедра биоинформатики МБФ РНИМУЗначения экспрессии генов из
- 41. Этап 6: Биологическое подтверждениеМикроэррейные эксперименты можно рассматривать
- 42. Этап 7: Микроэррейные базы данныхЕсть две основных
- 43. Кафедра биоинформатики МБФ РНИМУ
- 44. Кафедра биоинформатики МБФ РНИМУ
- 45. Кафедра биоинформатики МБФ РНИМУ
- 46. Кафедра биоинформатики МБФ РНИМУ
- 47. Кафедра биоинформатики МБФ РНИМУ
- 48. Кафедра биоинформатики МБФ РНИМУ
- 49. Кафедра биоинформатики МБФ РНИМУ
- 50. Кафедра биоинформатики МБФ РНИМУ
- 51. Кафедра биоинформатики МБФ РНИМУ
- 52. Кафедра биоинформатики МБФ РНИМУ
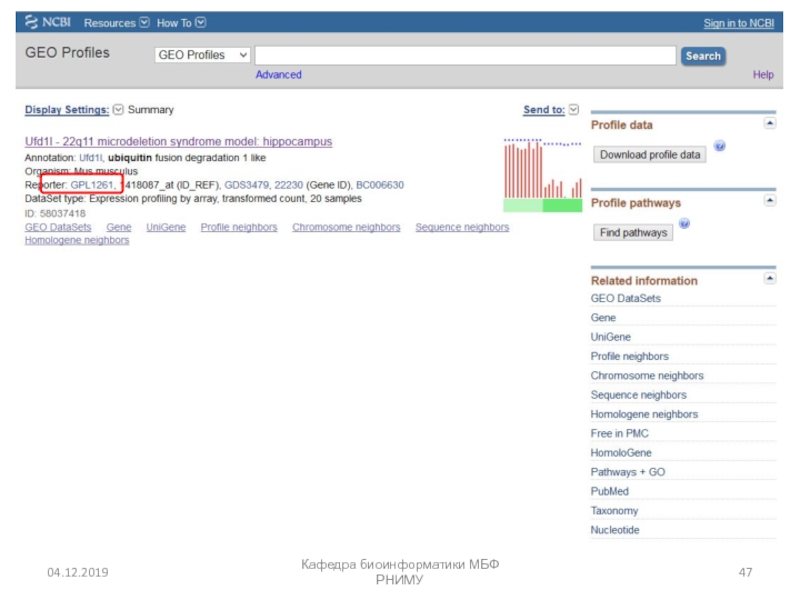
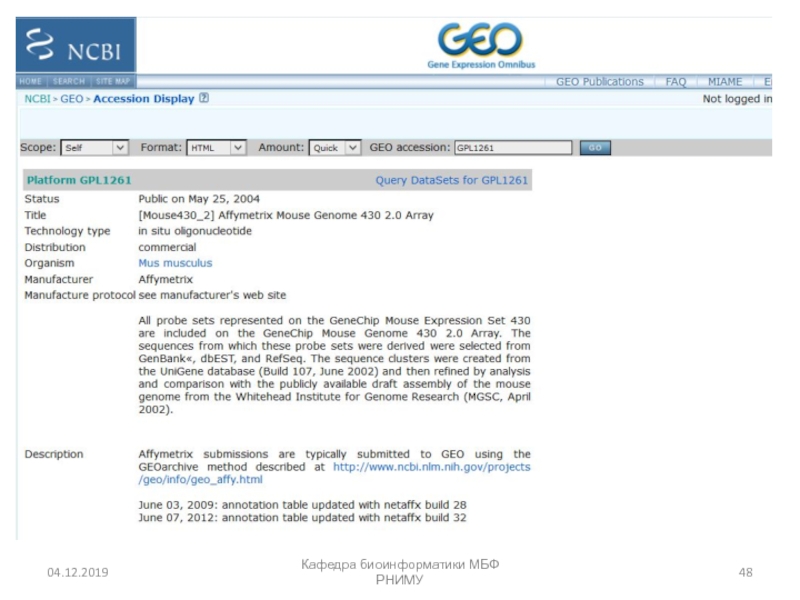
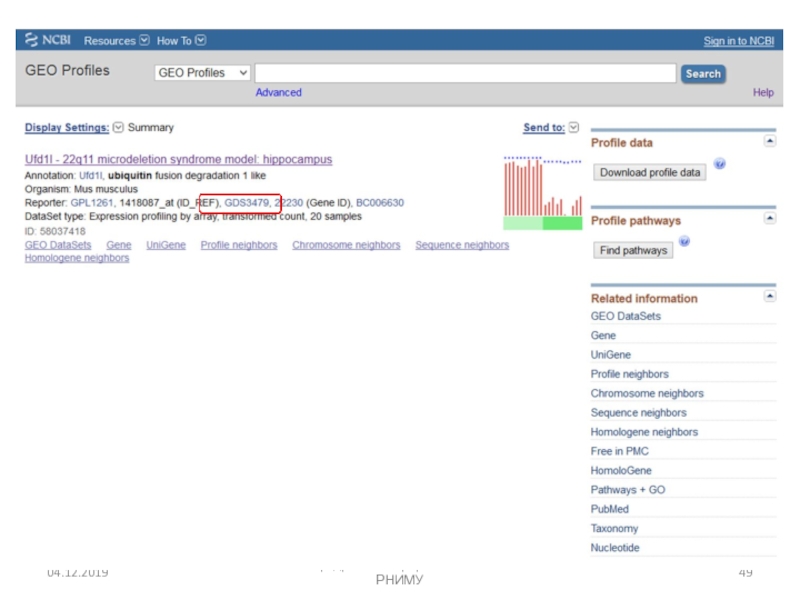
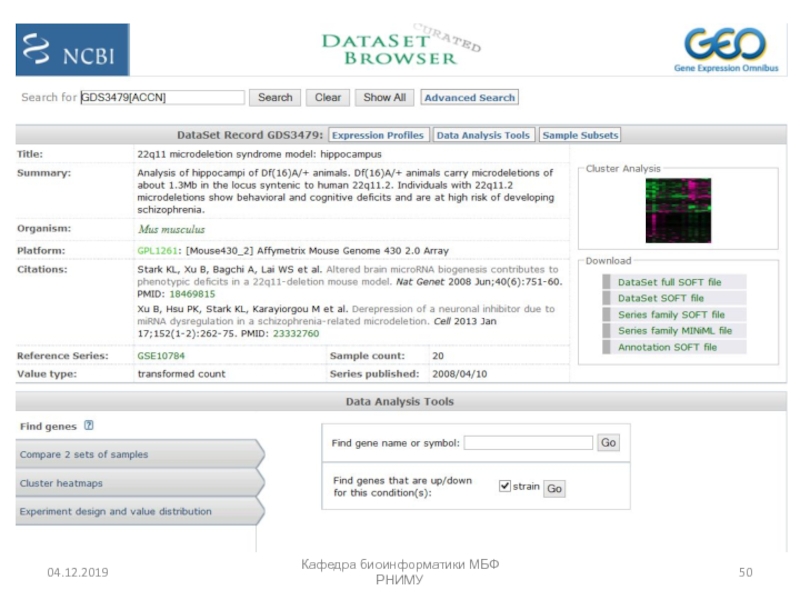
- 53. Array Express в European Bioinformatics Institute http://www.ebi.ac.uk/arrayexpress/Кафедра биоинформатики МБФ РНИМУ
- 54. Кафедра биоинформатики МБФ РНИМУ
- 55. Кафедра биоинформатики МБФ РНИМУ
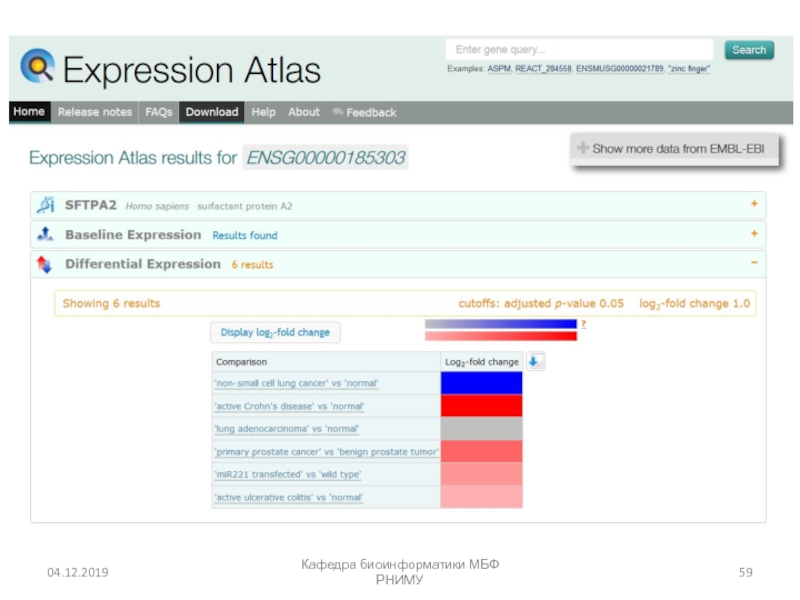
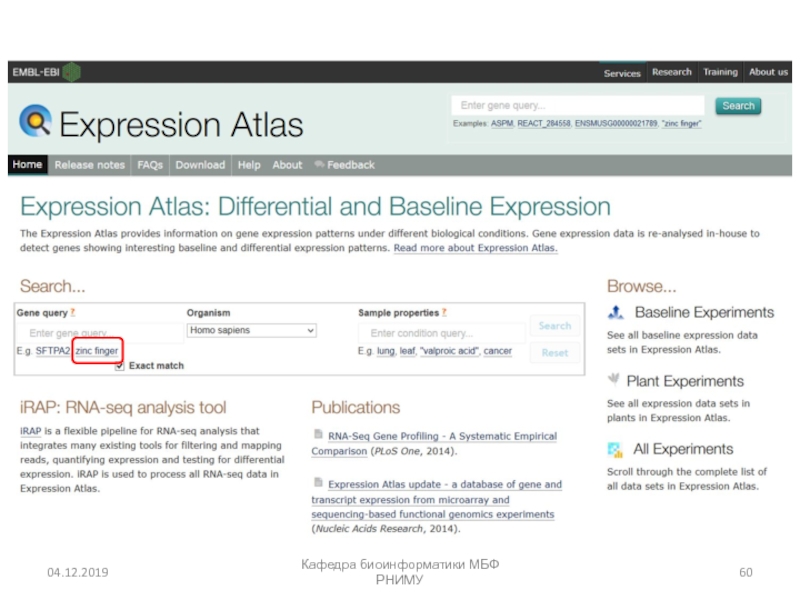
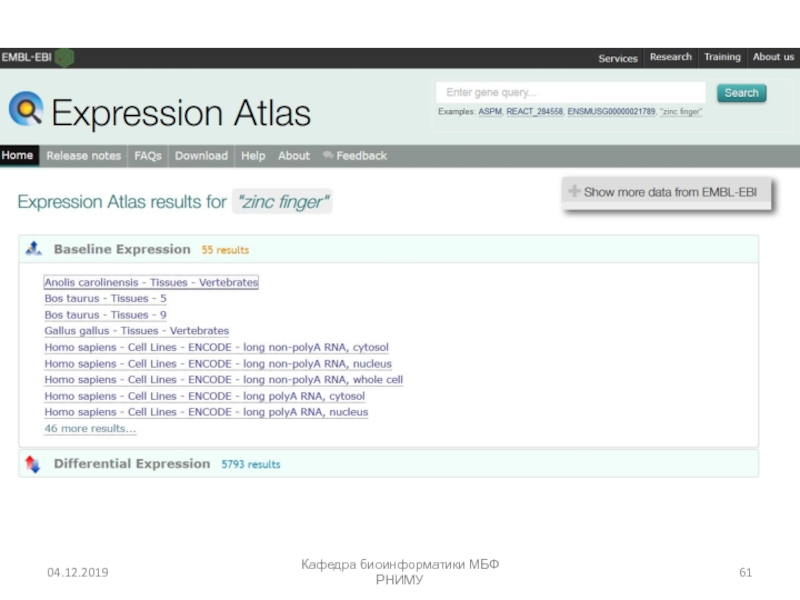
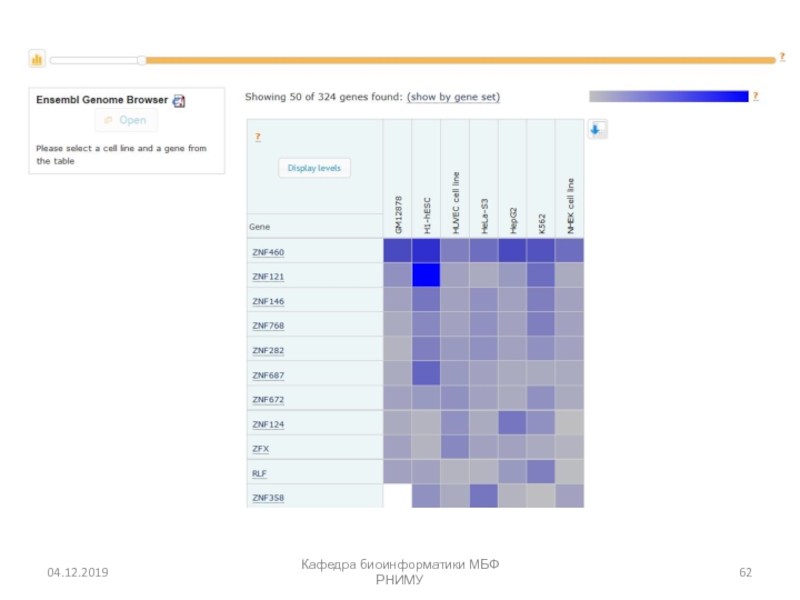
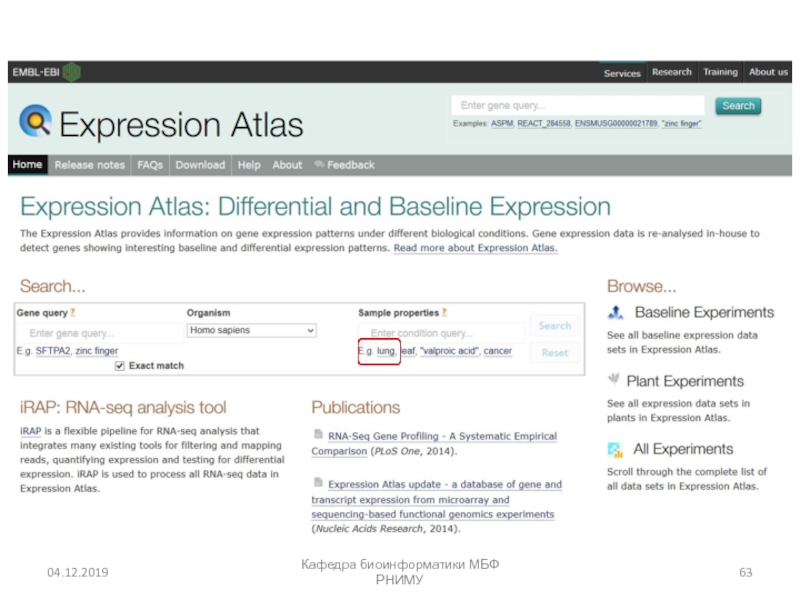
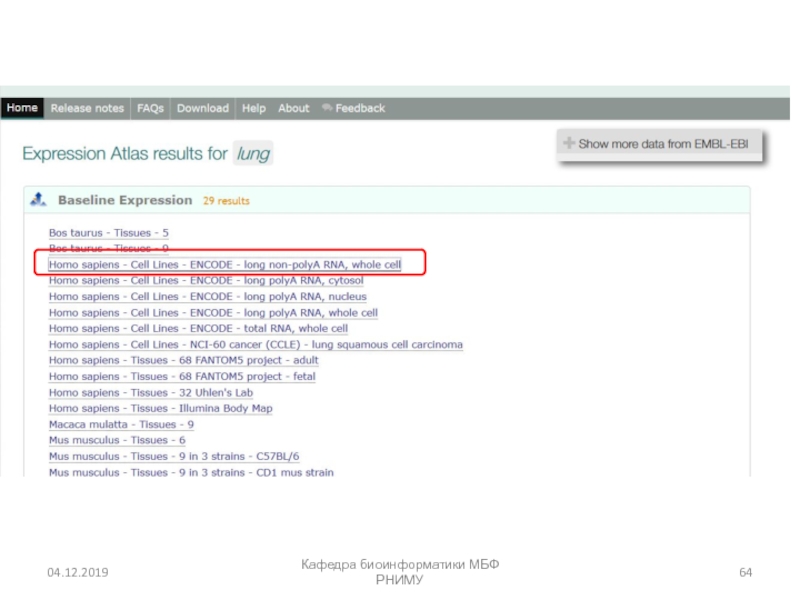
- 56. Expression Atlas (EMBL) https://www.ebi.ac.uk/gxa/homeКафедра биоинформатики МБФ РНИМУ
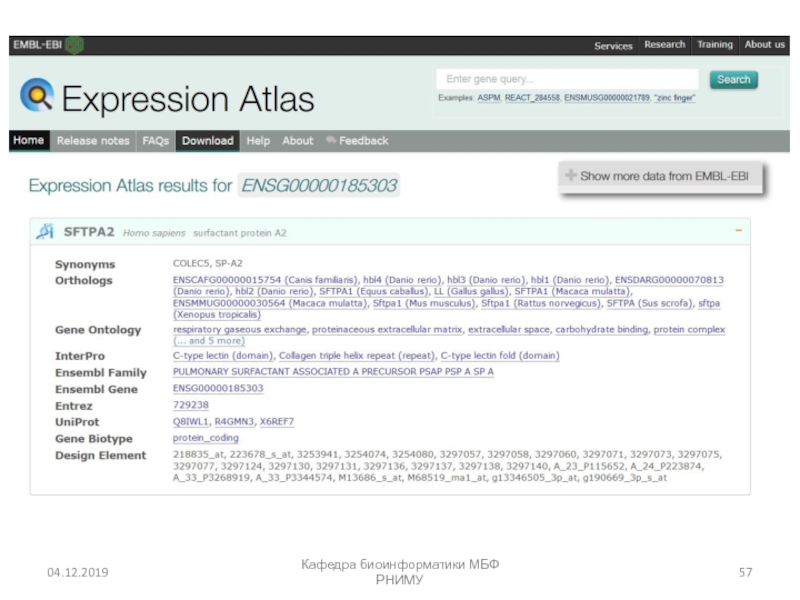
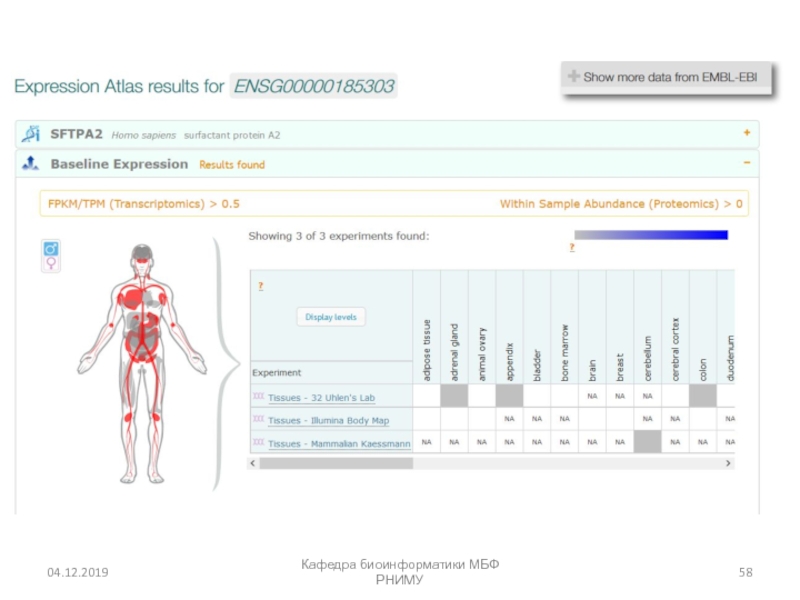
- 57. Кафедра биоинформатики МБФ РНИМУ
- 58. Кафедра биоинформатики МБФ РНИМУ
- 59. Кафедра биоинформатики МБФ РНИМУ
- 60. Кафедра биоинформатики МБФ РНИМУ
- 61. Кафедра биоинформатики МБФ РНИМУ
- 62. Кафедра биоинформатики МБФ РНИМУ
- 63. Кафедра биоинформатики МБФ РНИМУ
- 64. Кафедра биоинформатики МБФ РНИМУ
- 65. Кафедра биоинформатики МБФ РНИМУ
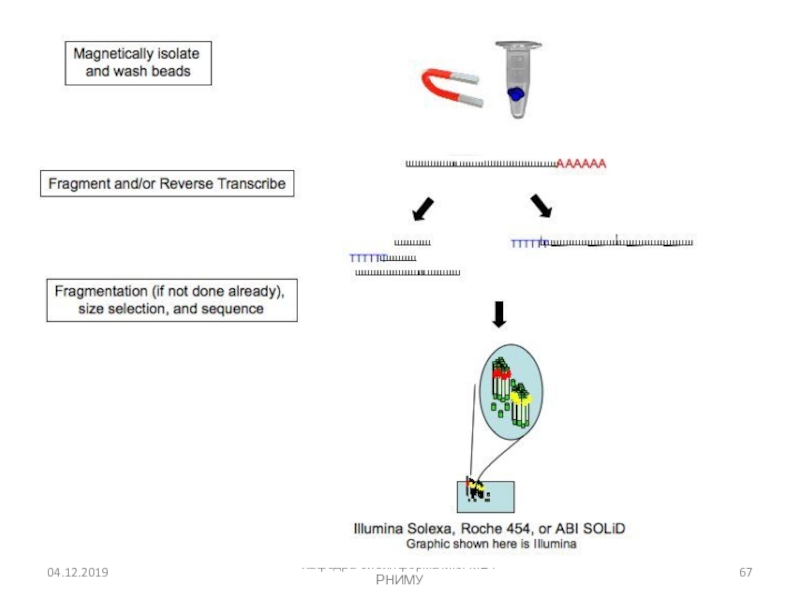
- 66. RNASeqКафедра биоинформатики МБФ РНИМУ(РНК Секвенирование), также называемый
- 67. Кафедра биоинформатики МБФ РНИМУ
- 68. Кафедра биоинформатики МБФ РНИМУ
- 69. Лекарственно-индуцированное изменение генной экспрессииКафедра биоинформатики МБФ
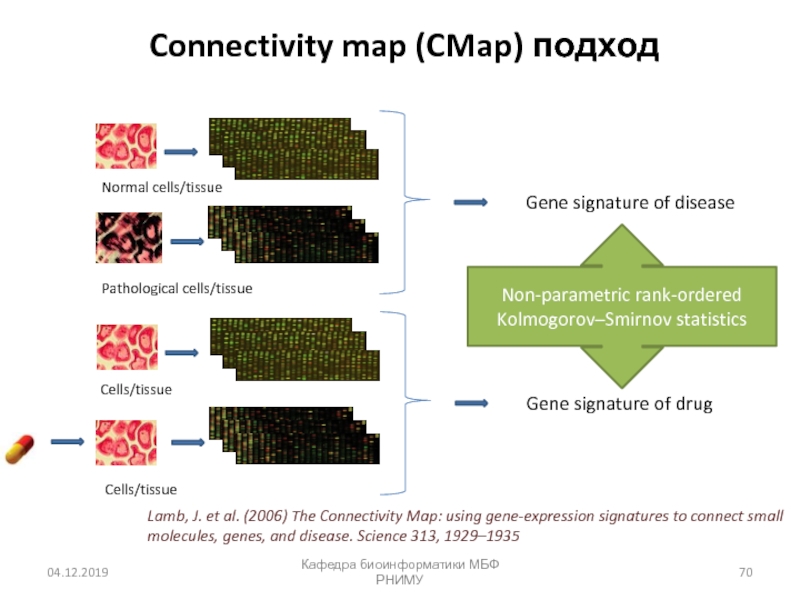
- 70. Connectivity map (CMap) подходКафедра биоинформатики МБФ РНИМУNormal
- 71. Применение CMap подходаКафедра биоинформатики МБФ РНИМУHuman Disease-Drug
- 72. MIAMEВ целях стандартизации представления и анализ данных
- 73. Экспрессия генов: анализ микроэрейных данныхЭкспрессия геновМикрочипы (Microarrays)Предварительная
- 74. Анализ микроэррейных данныхКафедра биоинформатики МБФ РНИМУНачинаем с
- 75. Предварительная обработка (препроцессинг) Наблюдаемые различия в
- 76. Основная цель предварительной обработки данных заключается в
- 77. Глобальная нормализация данных Глобальная нормализация используется для коррекции
- 78. Глобальная нормализация используется для коррекции двух или
- 79. Глобальная процедура нормализацииШаг 1: вычесть значения интенсивности
- 80. Диаграммы рассеяния (Scatter plots) Полезны для представления значений
- 81. Дифференциальная генная экспрессия в различных тканях и клеткахКафедра биоинформатики МБФ РНИМУМозгАстроцитАстроцитФибробласт
- 82. Кафедра биоинформатики МБФ РНИМУУровень экспрессииhighlowupdownУровень экспрессии (образец 1)Уровень экспрессии (образец 2)регуляция
- 83. Кафедра биоинформатики МБФ РНИМУЛогарифмическая трансформация данных
- 84. Диаграммы рассеяния (Scatter plots)Кафедра биоинформатики МБФ РНИМУОбычно
- 85. Кафедра биоинформатики МБФ РНИМУhttp://www.r-project.org
- 86. Эффект нормализацииКафедра биоинформатики МБФ РНИМУAAMMПосле RMA (Robust
- 87. Экспрессия генов: анализ микроэрейных данныхЭкспрессия геновМикрочипы (Microarrays)Предварительная
- 88. T-тестКафедра биоинформатики МБФ РНИМУT-тест широко используется для
- 89. Скачать презентанцию
Слайды и текст этой презентации
Слайд 1Биоинформатические подходы к анализу РНК. Экспрессия генов: анализ микроэррейных данных
презентации взяты из книги Bioinformatics and Functional Genomics by Jonathan Pevsner Copyright © 2009 by John Wiley & Sons, Inc. и соответствующего курса по биоинформатики Johns Hopkins School of Medicine
Слайд 2Кафедра биоинформатики МБФ РНИМУ
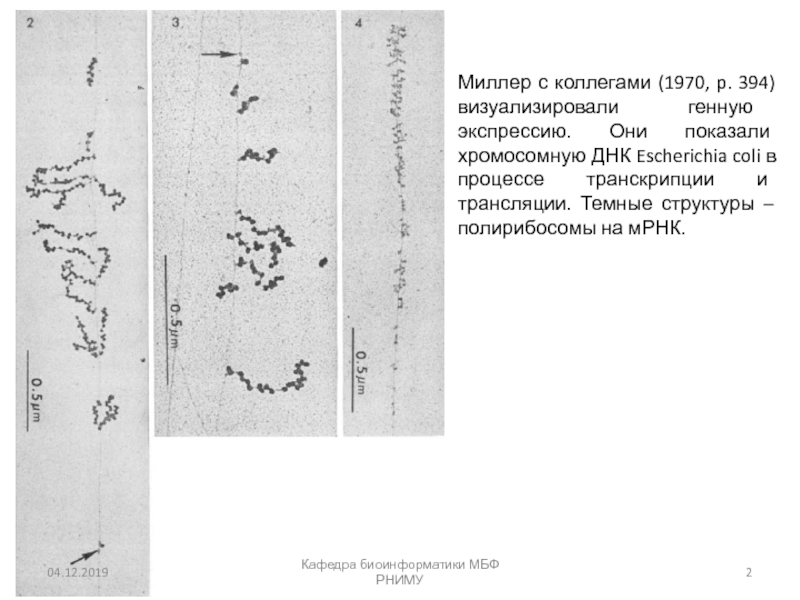
Миллер с коллегами (1970, p. 394) визуализировали
генную экспрессию. Они показали хромосомную ДНК Escherichia coli в процессе
транскрипции и трансляции. Темные структуры – полирибосомы на мРНК.Слайд 3Кафедра биоинформатики МБФ РНИМУ
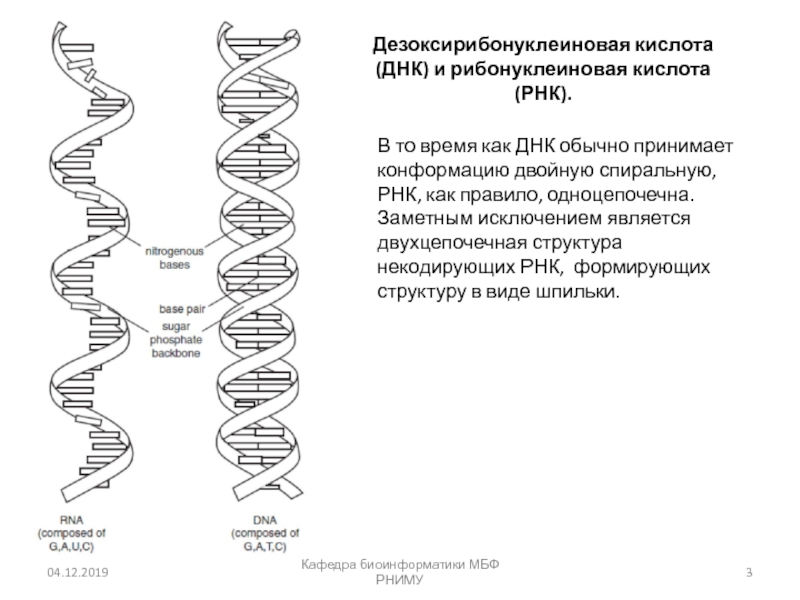
В то время как ДНК обычно принимает
конформацию двойную спиральную, РНК, как правило, одноцепочечна. Заметным исключением является
двухцепочечная структура некодирующих РНК, формирующих структуру в виде шпильки.Дезоксирибонуклеиновая кислота (ДНК) и рибонуклеиновая кислота (РНК).
Слайд 5Некодирующие РНК
Кафедра биоинформатики МБФ РНИМУ
Несколько ключевых дат:
1993 г: демонстрация нового
механизма подавления экспрессии:
C.elegans: гены lin-4 и lin-14 (белок LIN-14) антагонисты,
но lin-4 не кодирует белок.1999 г: открытие малых интерферирующих РНК (doi: 10.1126/science.286.5441.950)
2000 г: открытие второй микроРНК - lin-7 (doi: 10.1038/35002607)
2001 г: открытие сотни новых малых РНК, появление термина микроРНК (doi: 10.1126/science.1065329)
Первоначально все некодирующие РНК считали малыми; дальнейшие исследования выявили существенные различия по размерам и по функциям.
Слайд 6Некодирующие РНК (нкРНК)
Кафедра биоинформатики МБФ РНИМУ
Большое число и разные функции
нкРНК
Классификация по размерам:
малые нкРНК (sncRNA) - размер до 200 нт
(в н.в. известны тысячи)длинные нкРНК (lncRNA, lincRNA) - размер 200-50 тыс. нт (в н.в. известны десятки тысяч)
очень длинные нкРНК (vlincRNA) - размер от 50 тыс.нт до ~700 тыс.нт (в н.в. известны ~2000-3000) (doi: 10.1186/gb-2013-14-7-r73)
Малые нкРНК - высококонсервативны, длинные нкРНК - низкоконсервативные.
Наиболее изучены следующие малые нкРНК:
малые интерферирующие РНК;
малые ядерные РНК;
малые ядрышковые РНК;
малые РНК, образующие комплексы с piwi-белками;
малые РНК, образующие шпильки;
микроРНК.
Слайд 7Малые некодирующие РНК
Кафедра биоинформатики МБФ РНИМУ
Наибольшее функциональное значение имеют малые
РНК, вовлеченные в процессы генной регуляции - малые интерферирующие РНК
(siRNA) и микроРНК (miRNA):короткие интерферирующие РНК - длина 20-25 нт
микроРНК - длина 18-24 нт
~60% генов человека регулируются микроРНК (doi:10.1016/j.cell.2004.12.035);
2014 г: аннотировано ~1900 микроРНК человека; общее число микроРНК может достичь десятков тысяч;
огромное разнообразие вариантов регуляции:
одна miRNA -> несколько mRNA
несколько miRNA -> одну mRNA
влияние степени комплементарности
Существует значительное число биоинформационных программ для поиска микроРНК и их генов-мишеней
=> необходимо совершенствование расчетных методов поиска микроРНК, их генов-мишеней и других регуляторных участков
Слайд 18Экспрессия генов: анализ микроэрейных данных
Экспрессия генов
Микрочипы (Microarrays)
Предварительная обработка
нормализация
диаграммы рассеяния
Статистический анализ
Т-тест
ANOVA
расстояния
кластеризация
анализ
главных компонентов (PCA)
Кафедра биоинформатики МБФ РНИМУ
Слайд 19Кафедра биоинформатики МБФ РНИМУ
Сравнение экспрессии генов
в этом типе клеток …
…после
применения
лекарства
…в разное время жизни
…в различных участках
тела
…после вирусной
инфекции
…с
образцами от
пациентов… по отношению к
животному с нокаут геном
Слайд 20Экспрессия генов зависит от контекста, и регулируется несколькими основными способами
по
тканям и органам (например, мозг по сравнению с почкой)
в процессе
развития (например плода по сравнению с взрослой ткани)в динамическом ответ на сигналы внешней среды (например, лекарств)
при патологических состояниях
с помощью активности других генов
эпигенетически
Кафедра биоинформатики МБФ РНИМУ
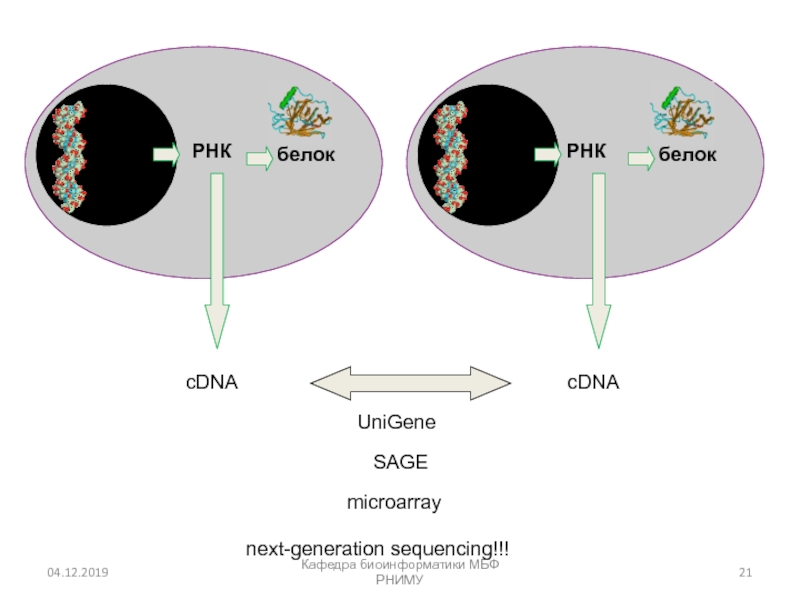
Слайд 21Кафедра биоинформатики МБФ РНИМУ
DNA
РНК
cDNA
белок
DNA
РНК
cDNA
белок
UniGene
SAGE
microarray
next-generation sequencing!!!
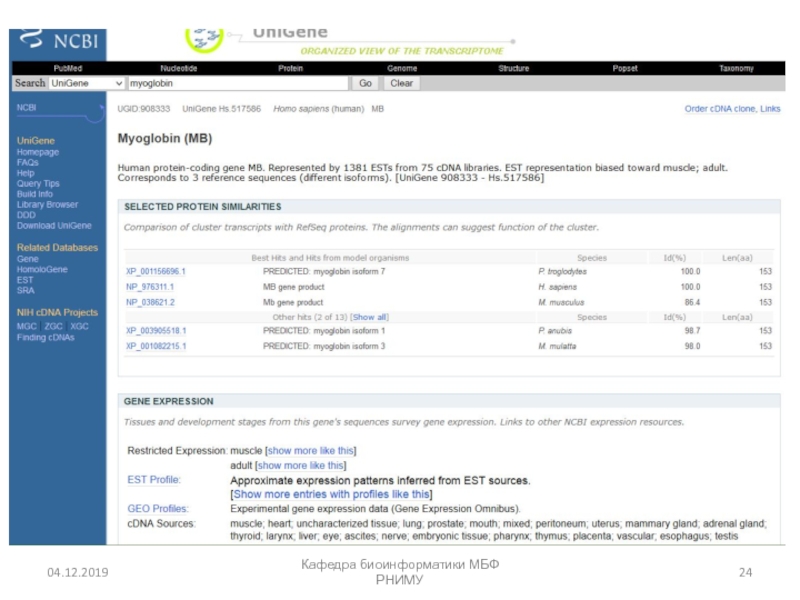
Слайд 22UniGene:
уникальные гены представленные ESTs
Unigene в NCBI:
www.ncbi.nlm.nih.gov/UniGene
Unigene кластеры содержат
много ESTs
(Expressed Sequence Tags - короткая подпоследовательность кДНК последовательности)
Данные
Unigene приходят из многих библиотек кДНК.Таким образом, когда вы смотрите на ген в Unigene вы получите информацию о величине и месте его экспрессии.
Кафедра биоинформатики МБФ РНИМУ
Слайд 26Microarrays: инструмент
для измерения экспрессии генов
микрочипом является твердый носитель (такой
как мембрана или предметное стекло микроскопа), на котором ДНК известной
последовательности нанесена в виде решетки матрицы.Кафедра биоинформатики МБФ РНИМУ
Слайд 27Кафедра биоинформатики МБФ РНИМУ
Microarrays: инструмент
для измерения экспрессии генов
Наиболее распространенная
форма микрочипа используется для измерения экспрессии генов. РНК выделяют из
образцов, представляющих интерес. РНК, как правило, превращают в кДНК, помеченную флуоресцентной (или радиоактивной) меткой, а затем гибридизуют на микрочип для того, чтобы измерить уровни экспрессии тысяч генов.Слайд 28Преимущества микроэррейных экспериментов
Скорость: данные о > 20000 транскриптов несколько дней
Всесторонность
исследования: весь геном дрожжей или мыши на чипе
Гибкость: Пользовательские микроэрреи
могут быть сделаны, для представляющих интерес геновЛегкость создания: Добавление РНК в чип
Стоимость?: Чип, представляющий 20 000 генов за $ 300
Кафедра биоинформатики МБФ РНИМУ
Слайд 29Недостатки микроэррейных экспериментов
Цена: Некоторые исследователи не могут позволить себе сделать
статистически значимое количество измерений
Значимость РНК:
Отсутствие корреляции между экспрессией генов
и количеством белкаПолная транскрипция генома плохо понимаема
Много некодирующих РНК не представлены в микрочипах
Контроль качества:
Артефакты при анализе изображения
Артефакты при анализе данных
Недостаточно внимание к планированию эксперимента
Не хватает правильной статистической обработки
Кафедра биоинформатики МБФ РНИМУ
Слайд 30Кафедра биоинформатики МБФ РНИМУ
Этап 1: Экспериментальный дизайн
Этап 3: Гибридизация с
ДНК чипом
Этап 2: РНК и пробо подготовка
Этап 4: Анализ изображений
Этап
5: Анализ микроэррейных данныхЭтап 6: Биологическое подтверждение
Этап 7: Микроэррейные базы данных
Слайд 31Этап 1: Экспериментальный дизайн
[1] Биологические образцы: технические и биологические повторы:
определить подход к анализу данных с самого начала [2] Выделение РНК,
преобразование, маркировка, гибридизация [3] Расположение элементов массива на поверхности: рандомизации может уменьшить пространственные артефактыКафедра биоинформатики МБФ РНИМУ
Слайд 32Один образец на массив
(Affymetrix или платформы на радиоактивных метках)
Кафедра
биоинформатики МБФ РНИМУ
Sample 1
Sample 2
Sample 3
Слайд 34Этап 2: РНК и пробо подготовка
Для Affymetrix чипов, нужна полная
РНК (около 5 мкг)
Подтвердите чистоту, запустив ее в агарозном геле
Один
из самых больших источников ошибки, связанных с выделения РНК;Использование соответствующего сбалансированного, рандомизированого дизайна эксперимента.
Кафедра биоинформатики МБФ РНИМУ
Слайд 35Этап 3: Гибридизация с ДНК чипом
Массив состоит из кДНК или
олигонуклеотидов
Олигонуклеотиды могут быть нанесены с помощью фотолитографии
Образец преобразуется в кРНК
или кДНККафедра биоинформатики МБФ РНИМУ
Слайд 37Этап 4: Анализ изображений
Уровни РНК-транскриптов являются количественными
Интенсивность флуоресценции или радиоактивности
измеряют с помощью сканера
Кафедра биоинформатики МБФ РНИМУ
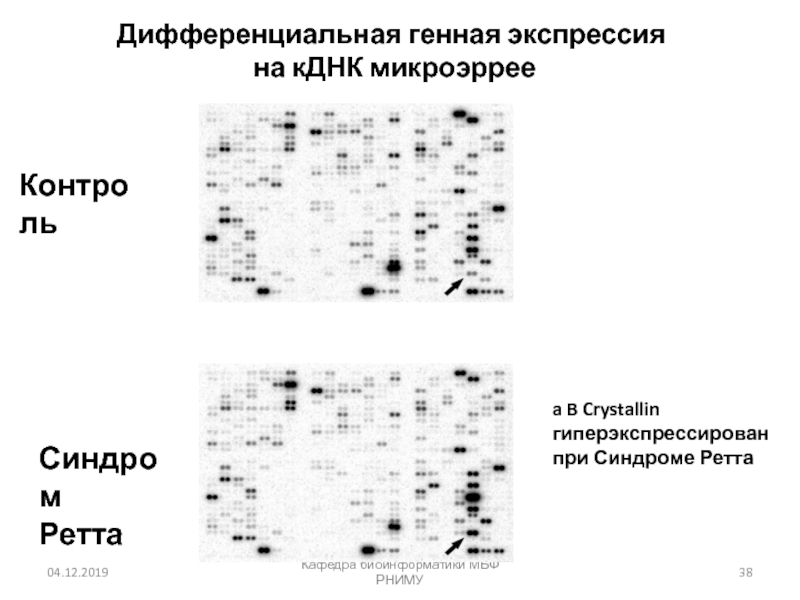
Слайд 38Дифференциальная генная экспрессия
на кДНК микроэррее
Кафедра биоинформатики МБФ РНИМУ
Синдром
Ретта
Контроль
a B Crystallin
гиперэкспрессирован при Синдроме Ретта
Слайд 39Этап 5: Анализ микроэррейных данных
Проверка гипотезы
Как можно сравнить массивы?
Какие РНК-транскрипты
(гены) регулируются?
Являются ли различия подлинным?
Каковы критерии для статистической значимости?
Кластеризация
Есть ли
значимые закономерности в данных (например, группы)?Классификация
Есть ли у РНК-транскриптов предсказанные заранее группы, такие как подтипы болезней?
Кафедра биоинформатики МБФ РНИМУ
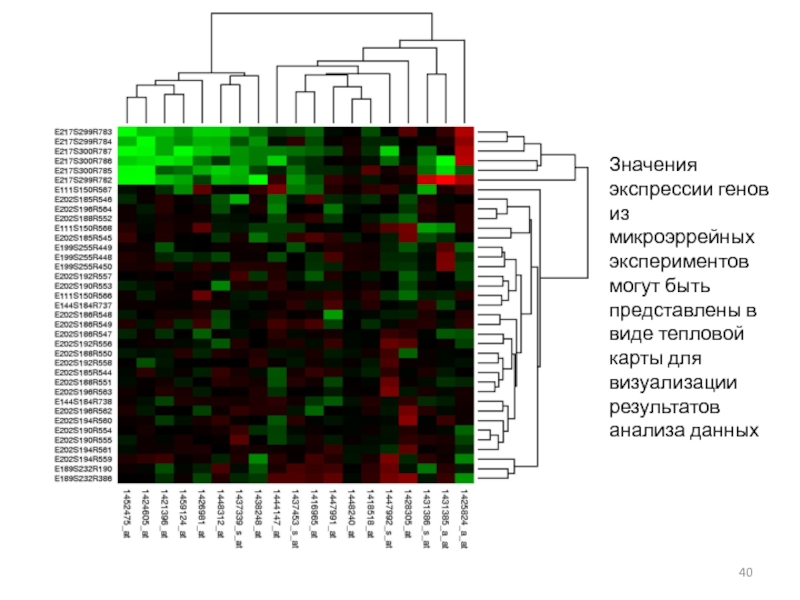
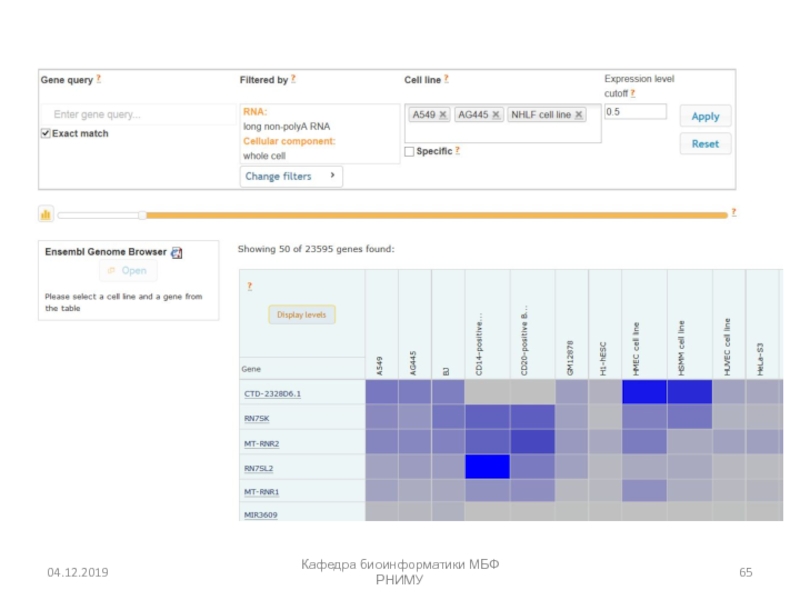
Слайд 40Кафедра биоинформатики МБФ РНИМУ
Значения экспрессии генов из микроэррейных экспериментов могут
быть представлены в виде тепловой карты для визуализации результатов анализа
данныхСлайд 41Этап 6: Биологическое подтверждение
Микроэррейные эксперименты можно рассматривать как «генераторы гипотез».
Дифференциальное
регулирования РНК-транскриптов может быть измерено с помощью независимых анализов, таких
какНозерн-блот
Полимеразная цепная реакция (ПЦР)
Гибридизация
Кафедра биоинформатики МБФ РНИМУ
Слайд 42Этап 7: Микроэррейные базы данных
Есть две основных базы данных
Gene expression
omnibus (GEO) в NCBI
ArrayExpress в European Bioinformatics Institute (EBI)
Кафедра биоинформатики
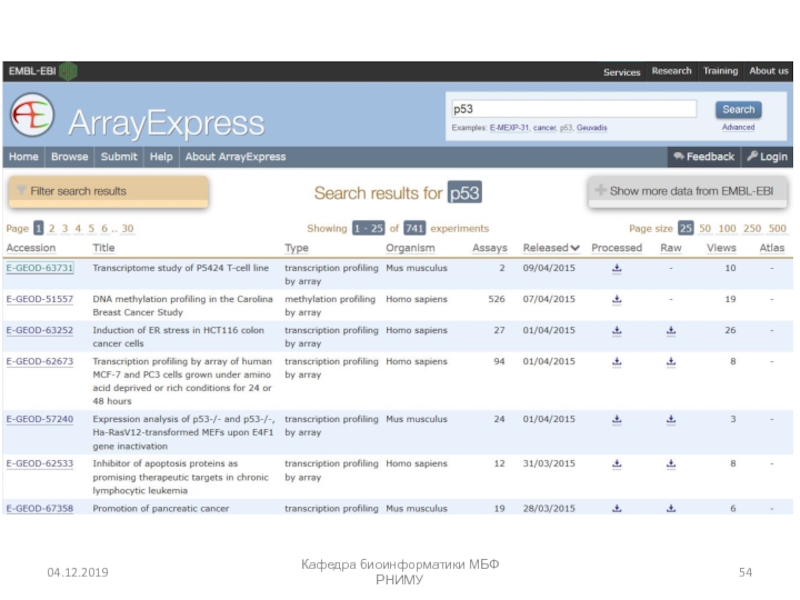
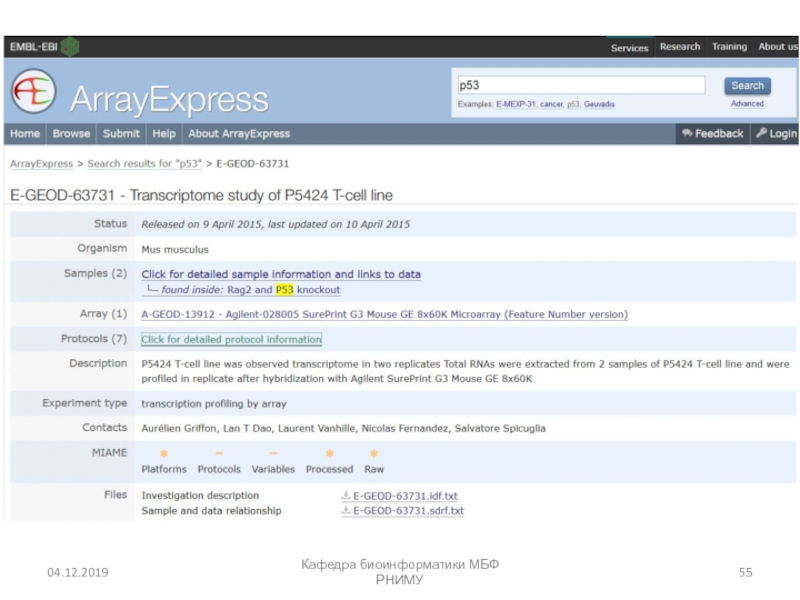
МБФ РНИМУСлайд 53Array Express в European Bioinformatics Institute
http://www.ebi.ac.uk/arrayexpress/
Кафедра биоинформатики МБФ РНИМУ
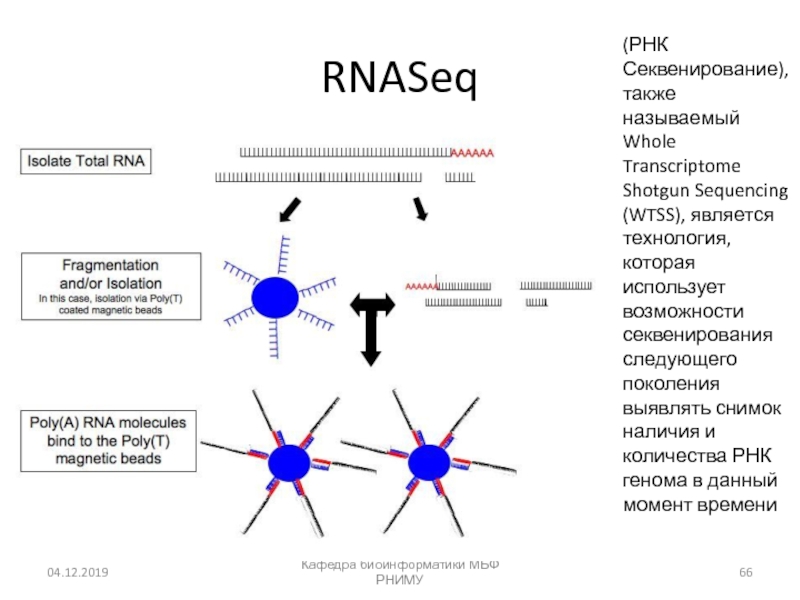
Слайд 66RNASeq
Кафедра биоинформатики МБФ РНИМУ
(РНК Секвенирование), также называемый Whole Transcriptome Shotgun
Sequencing (WTSS), является технология, которая использует возможности секвенирования следующего поколения
выявлять снимок наличия и количества РНК генома в данный момент времениСлайд 69Лекарственно-индуцированное
изменение генной экспрессии
Кафедра биоинформатики МБФ РНИМУ
Cells/tissue
Cells/tissue
Normalization
Comparative analysis by
statistical methods
(e.g. Student's t-test
or hypergeometric test)
Up regulated
genes
CASP3
CDKN1A
CDKN1B
FAS
GPX5
TP53
Down regulated
genes
CCND1
CCND2
MYC
NFKB1
PCNA
PTGS2
Исходные данные микроэррейных экспериментов
Лекарственно-индуцированное
изменение профиля генной экспрессии или генетическая подпись
Слайд 70Connectivity map (CMap) подход
Кафедра биоинформатики МБФ РНИМУ
Normal cells/tissue
Pathological cells/tissue
Gene signature
of disease
Cells/tissue
Gene signature of drug
Non-parametric rank-ordered Kolmogorov–Smirnov statistics
Cells/tissue
Lamb, J. et
al. (2006) The Connectivity Map: using gene-expression signatures to connect small molecules, genes, and disease. Science 313, 1929–1935Слайд 71Применение CMap подхода
Кафедра биоинформатики МБФ РНИМУ
Human Disease-Drug Network Based on
Genomic Expression Profiles - used Connectivity Map and GEO DataSets
for drug repositioning of 395 drugs (PLoS ONE, 2009, 4(8): e6536)Comprehensive gene expression profiles of NK cell neoplasms identify vorinostat as an effective drug candidate – used Connectivity Map data analysis for 6 drugs (Cancer Lett. 2013 pii: S0304-3835(13)00020-7)
Computational repositioning of the anticonvulsant topiramate for inflammatory bowel disease - GEO DataSets for 143 drugs (Sci Transl Med. 2011 August 17; 3(96): 96ra76)
Identification of Identical Transcript Changes in Liver and Whole Blood during Acetaminophen Toxicity – used in house data of gene expression profiles for acetaminophen. (Front Genet. 2012;3:162)
Prediction of synergistic effects of pairwise drug combinations from gene microarray data - used in house data of gene expression profiles from MCF-7 cells for docetaxel and gefitinib in different concentrations. (Bioinformatics, 2011, 27, i310-i316).
Слайд 72MIAME
В целях стандартизации представления и анализ данных микроэрреев, Alvis Brazma
и его коллеги из 17 учреждений ввели формат Minimum Information
About a Microarray Experiment (Минимальные сведения о микроэррейном эксперименте) - MIAME. В рамках MIAMI стандартизируется шесть областей информации:Планирование эксперимента
Дизайн микрочипа
Проба подготовка
Процедура гибридизации
Анализ изображений
Контроль в отношении нормализации
Кафедра биоинформатики МБФ РНИМУ
http://fged.org/projects/miame/
Слайд 73Экспрессия генов: анализ микроэрейных данных
Экспрессия генов
Микрочипы (Microarrays)
Предварительная обработка (препроцессинг)
нормализация
диаграммы рассеяния
(Scatter plots)
Статистический анализ
Т-тест
ANOVA
расстояния
кластеризация
анализ главных компонентов (PCA)
Кафедра биоинформатики МБФ РНИМУ
Слайд 74Анализ микроэррейных данных
Кафедра биоинформатики МБФ РНИМУ
Начинаем с матрицы данных
(значения
генной экспрессии в различных образцах)
гены
(уровни транскрипции РНК)
Обычно много генов
(>> 20,000)
и несколько образцов (~ 10)Слайд 75Предварительная обработка (препроцессинг)
Наблюдаемые различия в экспрессии генов могут
быть связаны с транскрипционными изменения, или же они могут быть
вызваны артефактами, такими, как:различная эффективность окрашивания Cy3 (зеленый), Cy5 (красный)
неравномерное распределение ДНК на поверхности массива
изменения связанные с чистотой или количеством РНК
изменения связанные с эффективностью отмывки
изменения связанные с эффективностью сканирования
Кафедра биоинформатики МБФ РНИМУ
Слайд 76Основная цель предварительной обработки данных заключается в как можно более
полном удалении систематической погрешности в данных, сохраняя при этом различия
в экспрессии генов, которое происходит из-за биологически соответствующих изменений в транскрипции.Основное предположение большинства процедур нормализации является то, что средний уровень экспрессии генов не меняется в эксперименте.
Кафедра биоинформатики МБФ РНИМУ
Предварительная обработка (препроцессинг)
Слайд 77Глобальная нормализация данных
Глобальная нормализация используется для коррекции двух или более
наборов данных. В одном общем случае образцы помеченные Cy3 (зеленый
краситель) или Cy5 (красный краситель) гибридизовали с ДНК-элементов на микрочипе. После промывки зонды возбуждаются с помощью лазера и исследуются с помощью сканирующего конфокального микроскопа.Кафедра биоинформатики МБФ РНИМУ
Слайд 78 Глобальная нормализация используется для коррекции двух или более наборов данных. Пример:
общая флуоресценция в Cy3 канал = 4 млн. единиц Су 5 канал
= 2 млн. единиц Тогда нескорректированное отношение для гена может показать 2000 единиц в сравнении 1000 единиц. Появление такого артефакта, приводит к тому, что показывает 2-кратное увеличение экспрессии.Кафедра биоинформатики МБФ РНИМУ
Глобальная нормализация данных
Слайд 79Глобальная процедура нормализации
Шаг 1: вычесть значения интенсивности фона (используется пустая
область массива)
Шаг 2: глобальная нормализация, так чтобы среднее соотношение было
равно 1 (применяется к 1-канальным или 2-канальным наборам данных)Кафедра биоинформатики МБФ РНИМУ
Слайд 80Диаграммы рассеяния (Scatter plots)
Полезны для представления значений экспрессии генов из
двух экспериментов микрочипов (например, контроль, эксперимент)
Каждая точка соответствует значению экспрессии
геновБольшинство точек находятся вдоль линии
Выбросы составляют гипо- и гиперэкспрессируемые гены
Кафедра биоинформатики МБФ РНИМУ
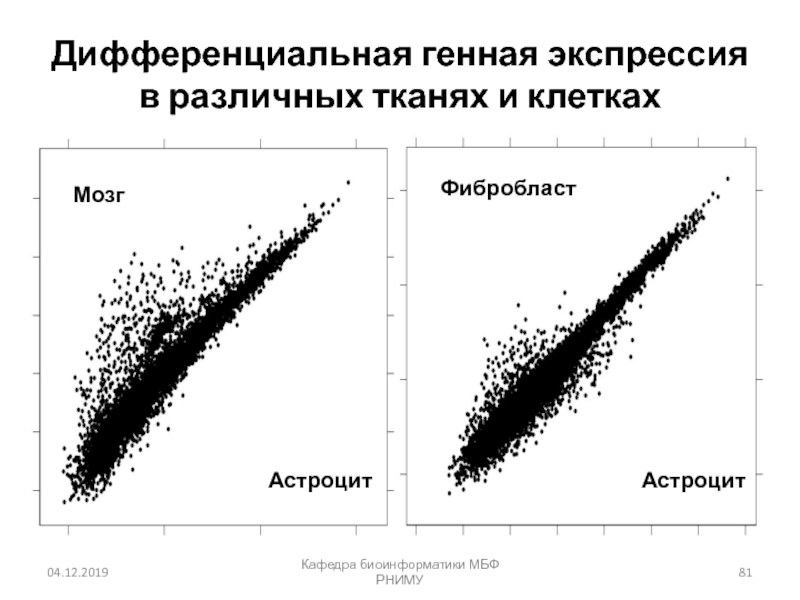
Слайд 81Дифференциальная генная экспрессия в различных тканях и клетках
Кафедра биоинформатики МБФ
РНИМУ
Мозг
Астроцит
Астроцит
Фибробласт
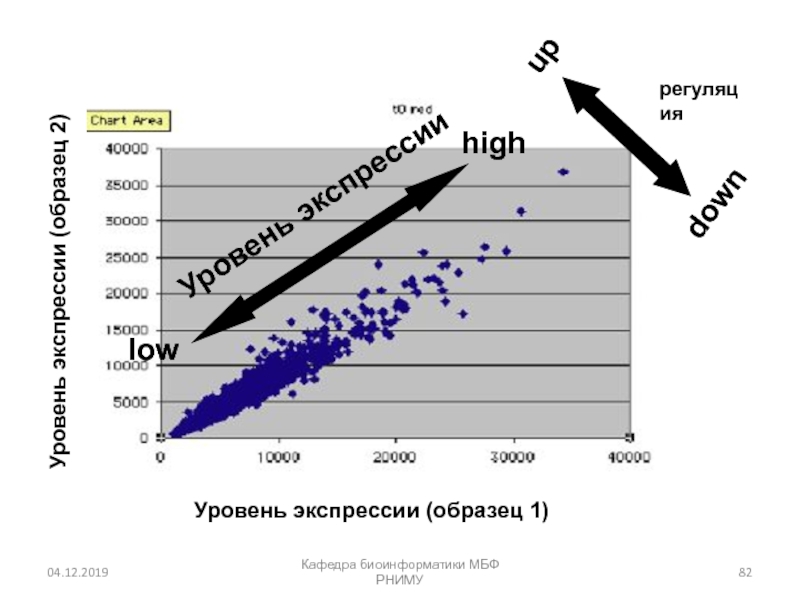
Слайд 82Кафедра биоинформатики МБФ РНИМУ
Уровень экспрессии
high
low
up
down
Уровень экспрессии (образец 1)
Уровень экспрессии (образец
2)
регуляция
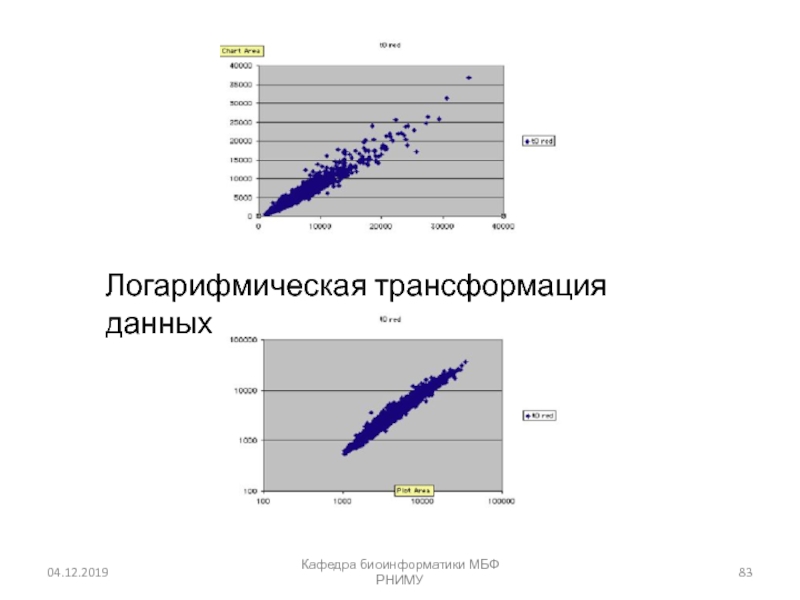
Слайд 84Диаграммы рассеяния (Scatter plots)
Кафедра биоинформатики МБФ РНИМУ
Обычно данные изображаются логарифмических
координатах
исходное log2
Время Изменение значение значение
t=0 начальное 1.0 0.0
t=1h нет изменений 1.0 0.0
t=2h 2-fold up 2.0 1.0
t=3h 2-fold down 0.5 -1.0
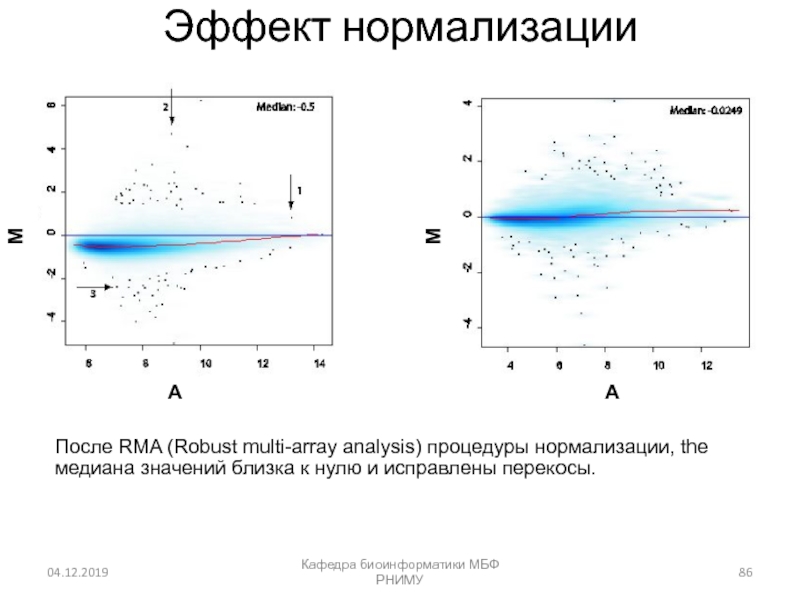
Слайд 86Эффект нормализации
Кафедра биоинформатики МБФ РНИМУ
A
A
M
M
После RMA (Robust multi-array analysis) процедуры
нормализации, the медиана значений близка к нулю и исправлены перекосы.
Слайд 87Экспрессия генов: анализ микроэрейных данных
Экспрессия генов
Микрочипы (Microarrays)
Предварительная обработка (препроцессинг)
нормализация
диаграммы рассеяния
(Scatter plots)
Статистический анализ
Т-тест
ANOVA
расстояния
кластеризация
анализ главных компонентов (PCA)
Кафедра биоинформатики МБФ РНИМУ
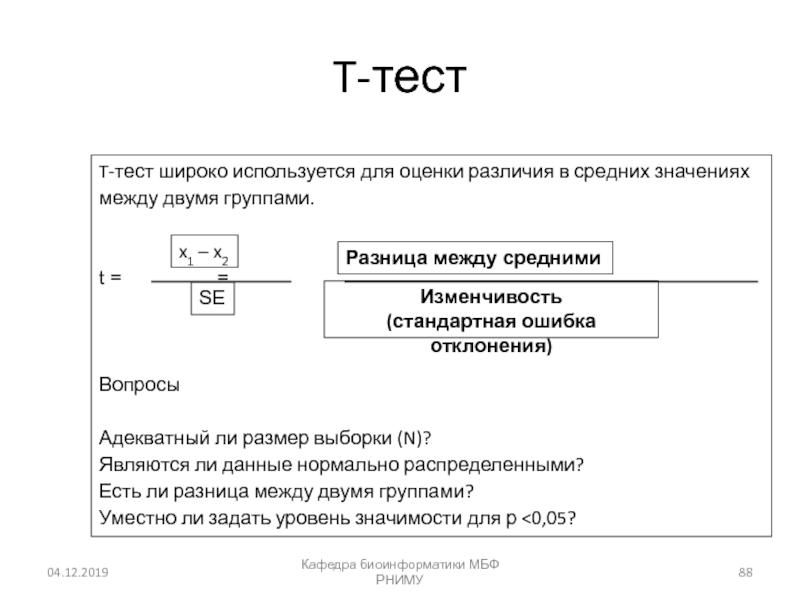
Слайд 88T-тест
Кафедра биоинформатики МБФ РНИМУ
T-тест широко используется для оценки различия в
средних значениях между двумя группами.
t = =
Вопросы
Адекватный ли размер выборки (N)? Являются ли данные нормально распределенными? Есть ли разница между двумя группами? Уместно ли задать уровень значимости для р <0,05?
x1 – x2
SE
Разница между средними
Изменчивость
(стандартная ошибка отклонения)
















![Биоинформатические подходы к анализу РНК. Экспрессия генов: анализ Этап 1: Экспериментальный дизайн[1] Биологические образцы: технические и биологические повторы: определить Этап 1: Экспериментальный дизайн[1] Биологические образцы: технические и биологические повторы: определить подход к анализу данных с самого](/img/thumbs/4df8a5a81c7c621b3f1fce2442794596-800x.jpg)