Разделы презентаций
- Разное
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Геометрия
- Детские презентации
- Информатика
- История
- Литература
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Эволюционные модели и дистанции между последовательностями
Содержание
- 1. Эволюционные модели и дистанции между последовательностями
- 2. Слайд 2
- 3. Слайд 3
- 4. Слайд 4
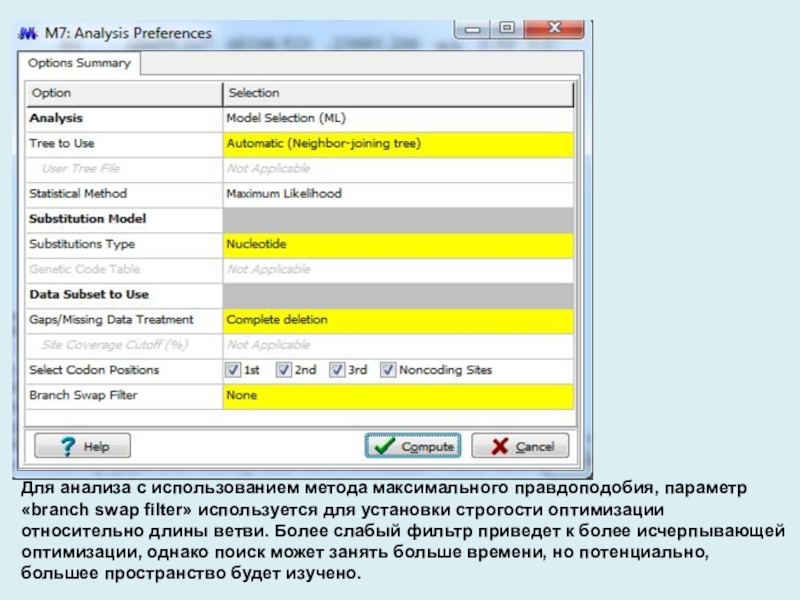
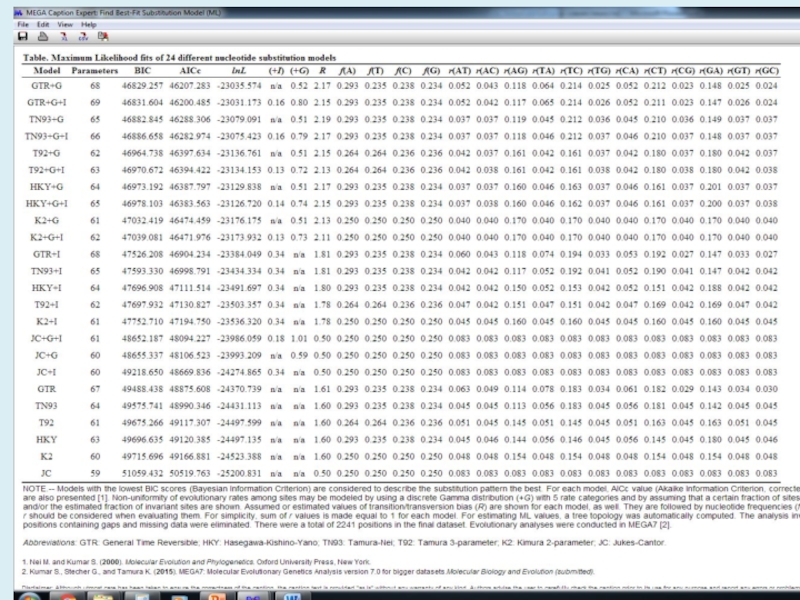
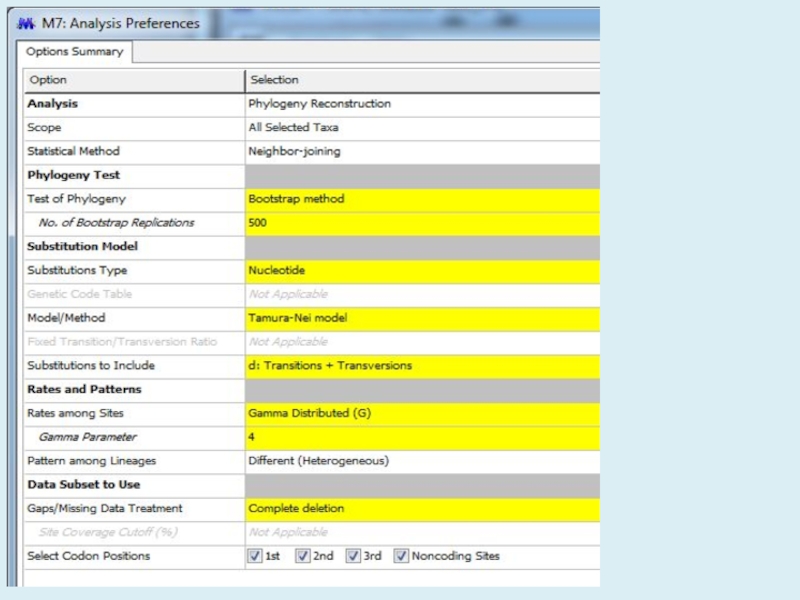
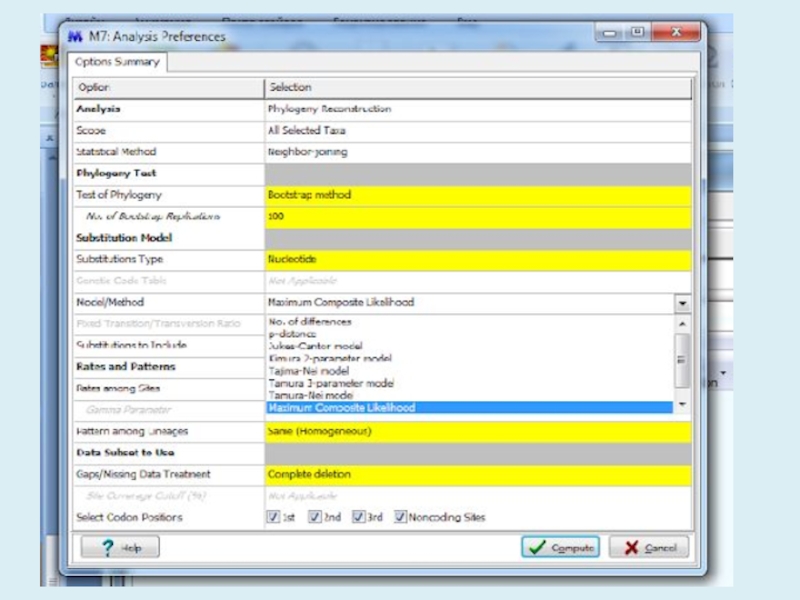
- 5. Для анализа с использованием метода максимального правдоподобия,
- 6. Слайд 6
- 7. Слайд 7
- 8. Слайд 8
- 9. Слайд 9
- 10. Слайд 10
- 11. Слайд 11
- 12. Слайд 12
- 13. Слайд 13
- 14. Слайд 14
- 15. Слайд 15
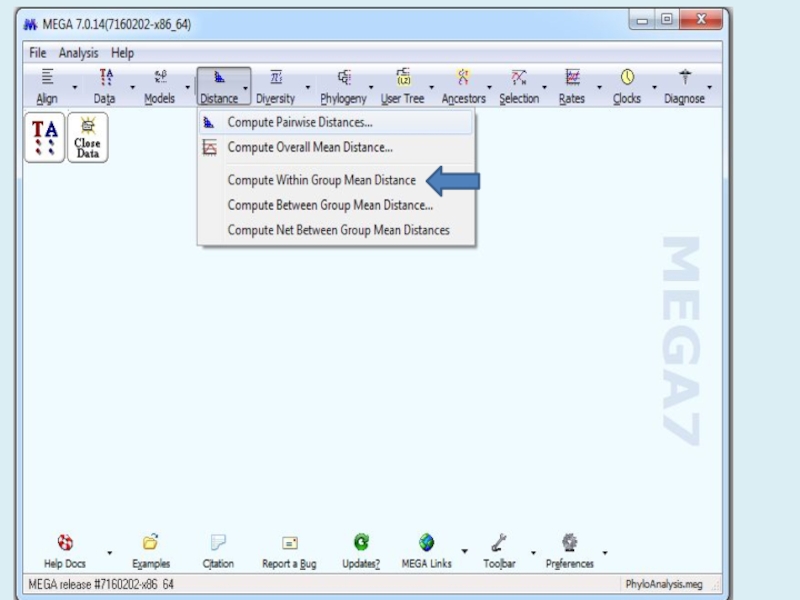
- 16. Gp 2 0.008 0.002Gp 1
- 17. [1] #Gp_2[2] #Gp_1[3] #4a[4] #4b[
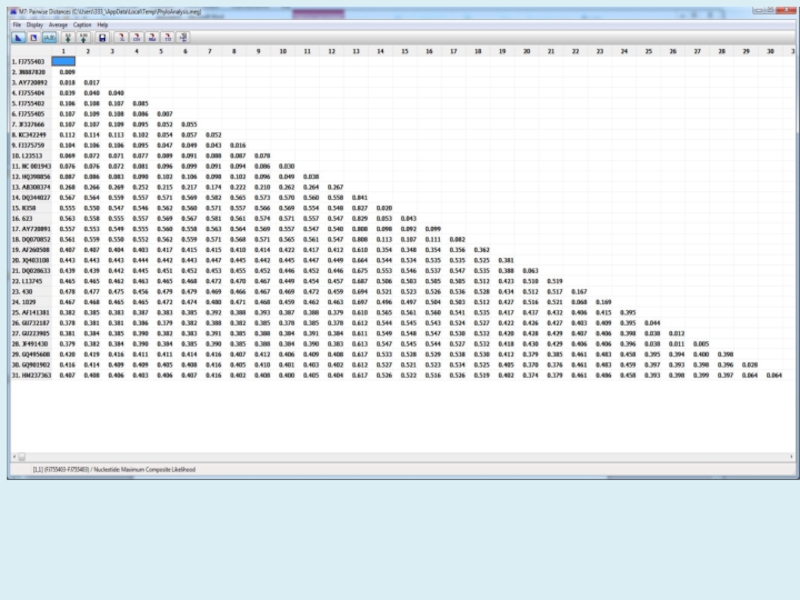
- 18. Таблица 1. Матрица средних значений генетических расстояний
- 19. Слайд 19
- 20. Слайд 20
- 21. Слайд 21
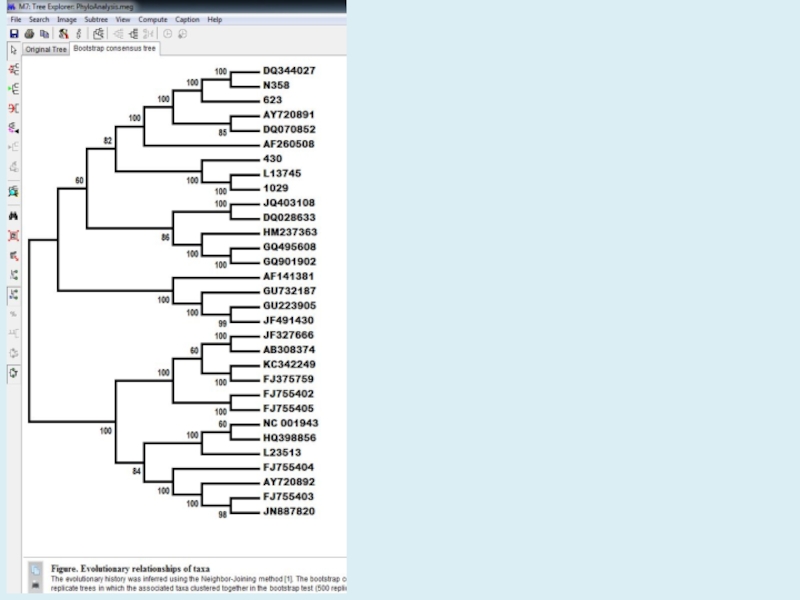
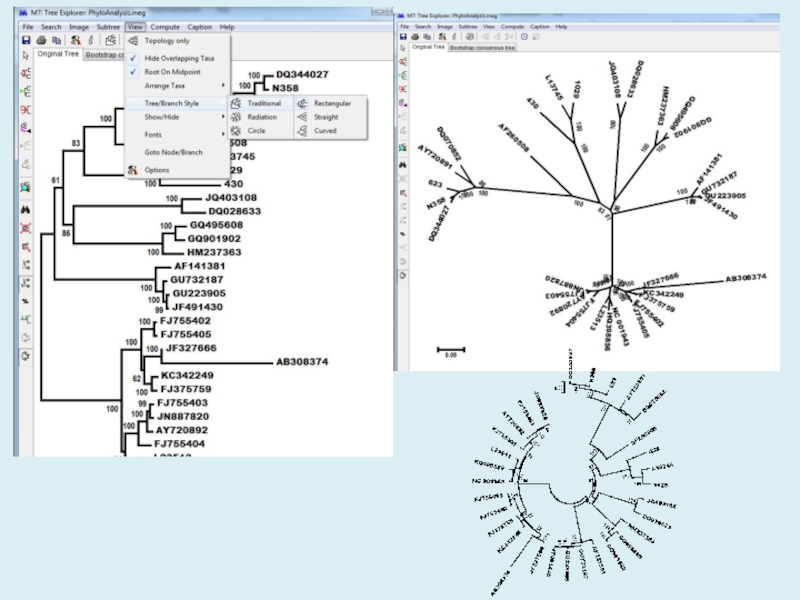
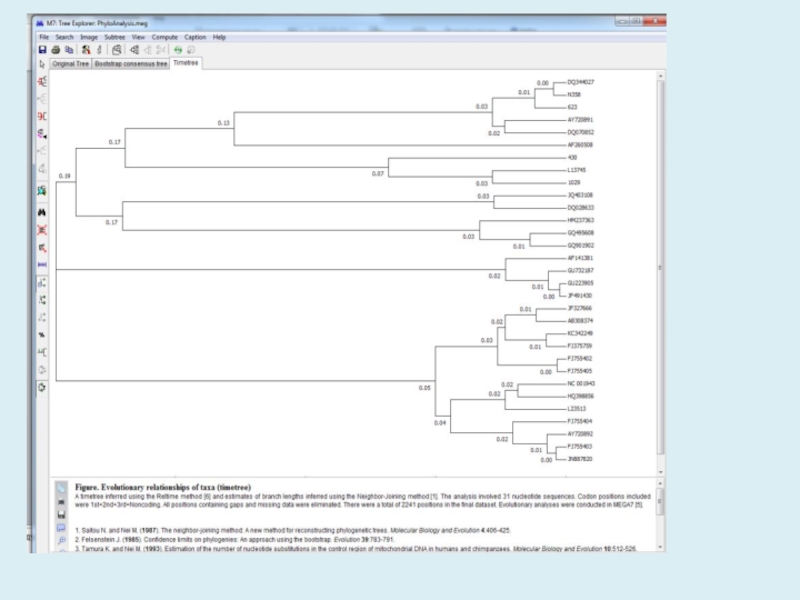
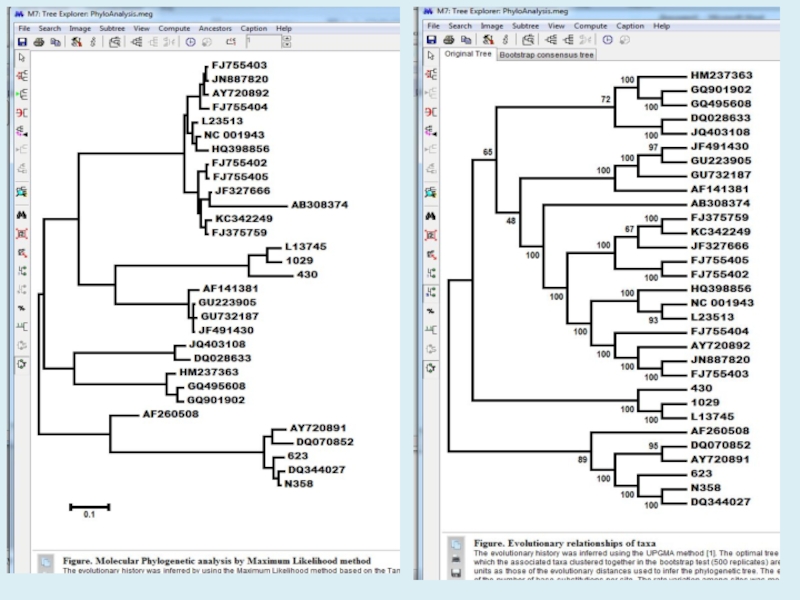
- 22. Figure. Evolutionary relationships of taxaThe evolutionary history
- 23. Слайд 23
- 24. Слайд 24
- 25. Слайд 25
- 26. Слайд 26
- 27. Слайд 27
- 28. Слайд 28
- 29. Слайд 29
- 30. Слайд 30
- 31. Слайд 31
- 32. Слайд 32
- 33. Слайд 33
- 34. Слайд 34
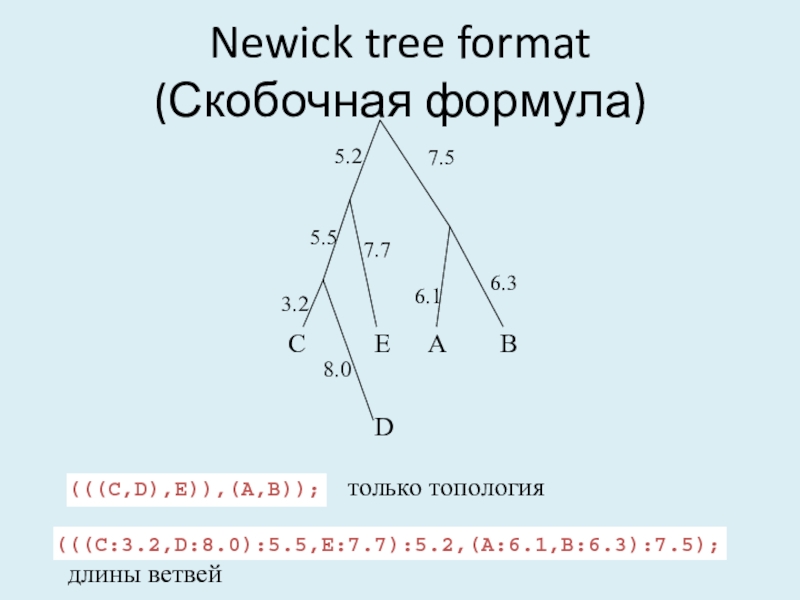
- 35. (((C:3.2,D:8.0):5.5,E:7.7):5.2,(A:6.1,B:6.3):7.5); длины ветвей (((C,D),E)),(A,B)); только топологияNewick tree format (Скобочная формула)ABCDE5.27.56.36.17.78.03.25.5
- 36. Слайд 36
- 37. http://www.iqtree.org/
- 38. Слайд 38
- 39. Слайд 39
- 40. Слайд 40
- 41. Слайд 41
- 42. Скачать презентанцию
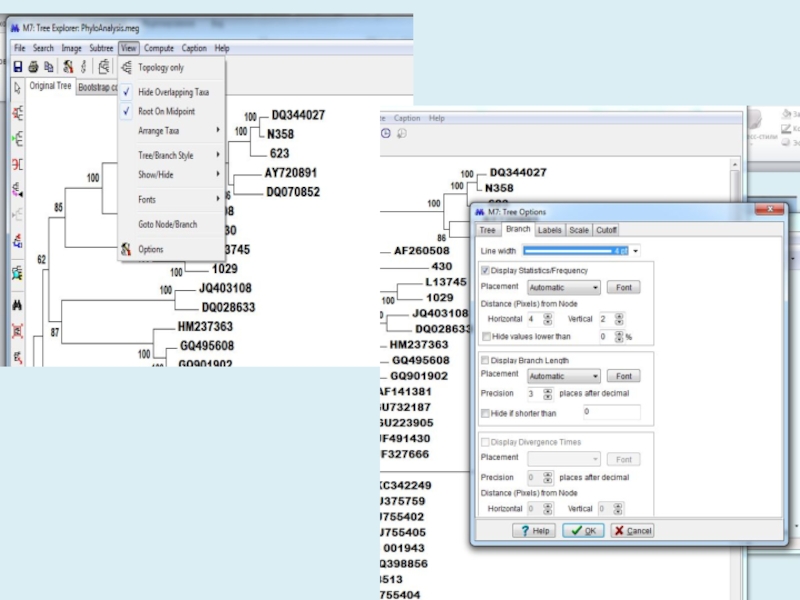
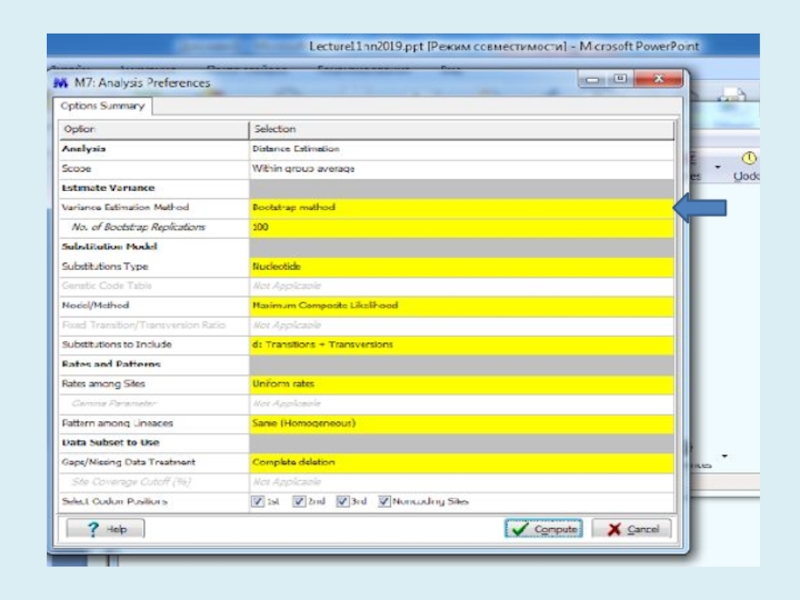
Для анализа с использованием метода максимального правдоподобия, параметр «branch swap filter» используется для установки строгости оптимизации относительно длины ветви. Более слабый фильтр приведет к более исчерпывающей оптимизации, однако поиск может занять
Слайды и текст этой презентации
Слайд 1Эволюционные модели и дистанции между последовательностями биополимеров
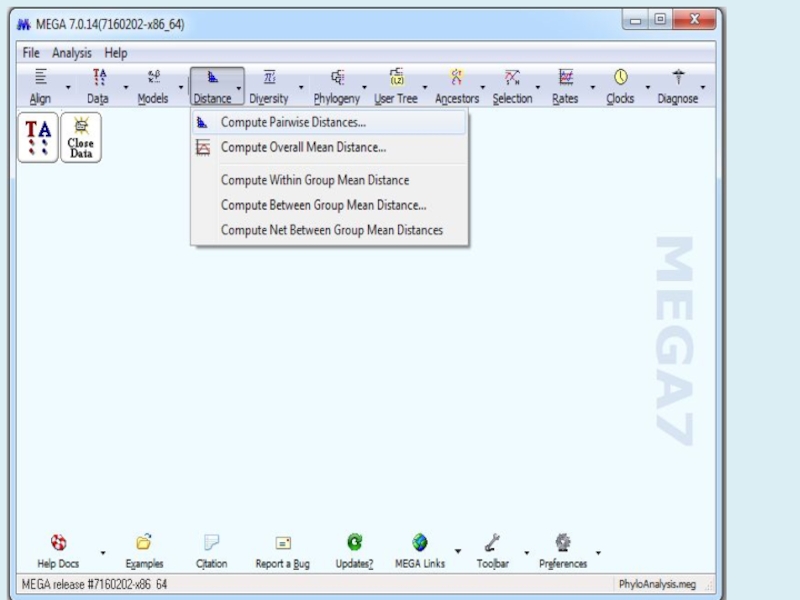
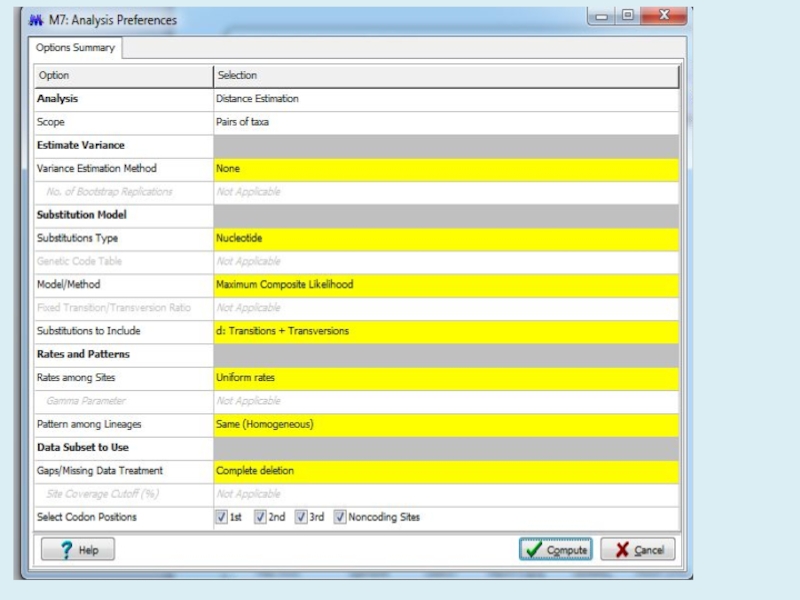
Молекулярная филогения
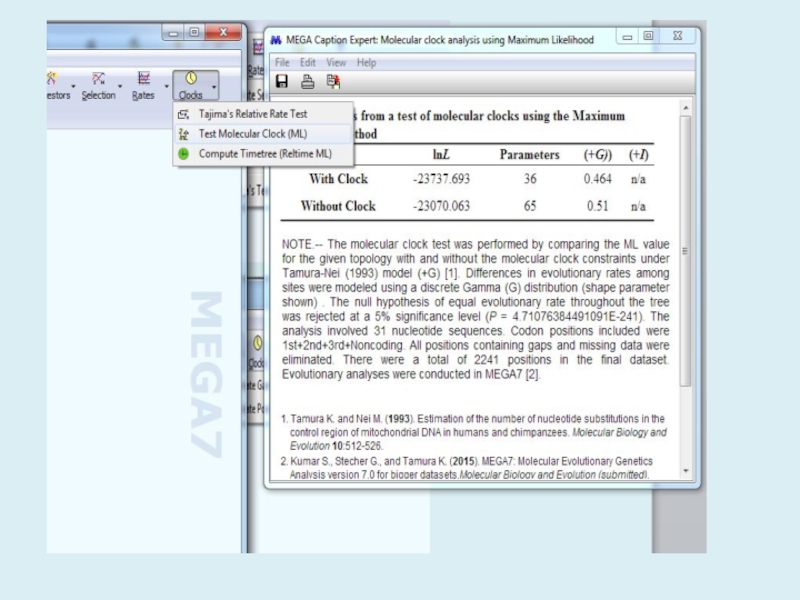
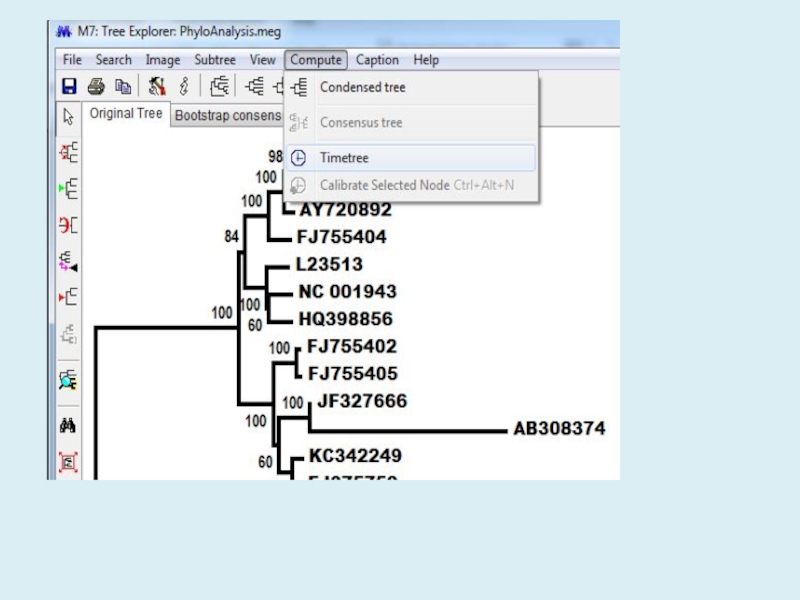
Статистические методы
оценки деревьев. Анализ молекулярных часов.
Слайд 5Для анализа с использованием метода максимального правдоподобия, параметр «branch swap
filter» используется для установки строгости оптимизации относительно длины ветви. Более
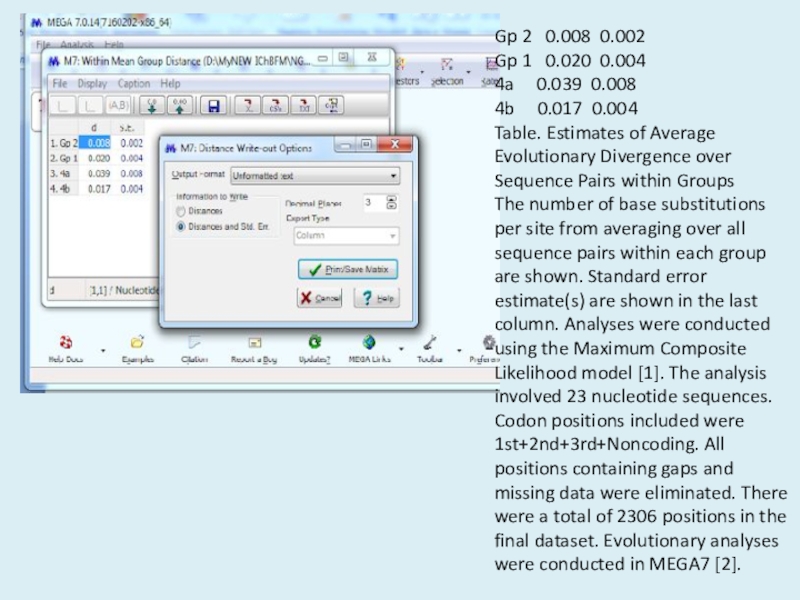
слабый фильтр приведет к более исчерпывающей оптимизации, однако поиск может занять больше времени, но потенциально, большее пространство будет изучено.Слайд 16Gp 2 0.008 0.002
Gp 1 0.020 0.004
4a
0.039 0.008
4b 0.017 0.004
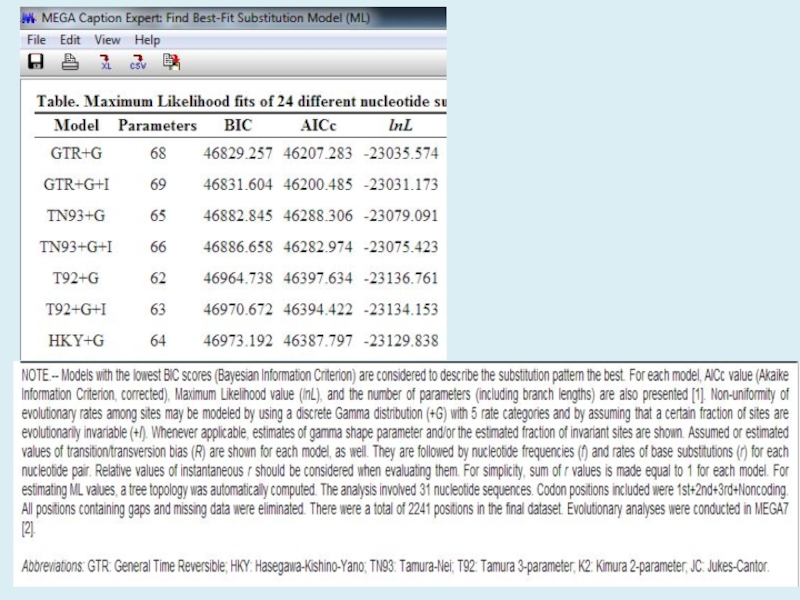
Table. Estimates of Average
Evolutionary Divergence over Sequence Pairs within Groups The number of base substitutions per site from averaging over all sequence pairs within each group are shown. Standard error estimate(s) are shown in the last column. Analyses were conducted using the Maximum Composite Likelihood model [1]. The analysis involved 23 nucleotide sequences. Codon positions included were 1st+2nd+3rd+Noncoding. All positions containing gaps and missing data were eliminated. There were a total of 2306 positions in the final dataset. Evolutionary analyses were conducted in MEGA7 [2].
Слайд 17
[1] #Gp_2
[2] #Gp_1
[3] #4a
[4] #4b
[ 1
2 3
4 ][1] [ 0.008 ][ 0.017 ][ 0.019 ]
[2] 0.042 [ 0.018 ][ 0.020 ]
[3] 0.091 0.099 [ 0.015 ]
[4] 0.104 0.111 0.082
Table. Estimates of Evolutionary Divergence over Sequence Pairs between Groups
The number of base substitutions per site from averaging over all sequence pairs between groups are shown. Standard error estimate(s) are shown above the diagonal. Analyses were conducted using the Maximum Composite Likelihood model [1]. The analysis involved 23 nucleotide sequences. Codon positions included were 1st+2nd+3rd+Noncoding. All positions containing gaps and missing data were eliminated. There were a total of 2306 positions in the final dataset. Evolutionary analyses were conducted in MEGA7 [2].
Слайд 18Таблица 1. Матрица средних значений генетических расстояний (число нуклеотидных замен
на сайт) между генотипами HAstV. В квадратных скобках приведено стандартное
отклонение, рассчитанное по 1000 реплик.1 2 3 4 5 6 8
[1]
[2] 0.259 [ 0.008]
[3] 0.228 [ 0.007] 0.241 [ 0.007]
[4] 0.236 [ 0.007] 0.271 [ 0.007] 0.269 [ 0.009]
[5] 0.232 [ 0.006] 0.276 [ 0.008] 0.236 [ 0.008] 0.251 [ 0.007]
[6] 0.329 [ 0.010] 0.335 [ 0.009] 0.318 [ 0.010] 0.362 [ 0.010] 0.309 [ 0.010]
[8] 0.201 [ 0.007] 0.241 [ 0.008] 0.230 [ 0.009] 0.164 [ 0.005] 0.207 [ 0.006] 0.322 [ 0.009]
Таблица 2. Средние значения генетических расстояний (число нуклеотидных замен на сайт) внутри генотипов HAstV. В квадратных скобках приведено стандартное отклонение, рассчитанное по 1000 реплик.
HastV-1 0.085 [0.002]
HastV-2 0.174 [0.004]
HastV-3 0.025 [0.001]
HastV-4 0.085 [0.003]
HastV-5 0.081 [0.004]
HAstV-6 0.055 [0.002]


![Эволюционные модели и дистанции между последовательностями [1] #Gp_2[2] #Gp_1[3] #4a[4] #4b[ 1 2 [1] #Gp_2[2] #Gp_1[3] #4a[4] #4b[ 1 2](/img/tmb/7/643776/b8c09b7fbab996142834ebbcbc79f7a9-800x.jpg)

![Эволюционные модели и дистанции между последовательностями Figure. Evolutionary relationships of taxaThe evolutionary history was inferred using the Figure. Evolutionary relationships of taxaThe evolutionary history was inferred using the Neighbor-Joining method [1]. The optimal tree](/img/tmb/7/643776/12f405462ac2d8f8eca62c08310d51d8-800x.jpg)