Разделы презентаций
- Разное
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Геометрия
- Детские презентации
- Информатика
- История
- Литература
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
Компьютерные методы анализа нуклеотидных последовательностей
Содержание
- 1. Компьютерные методы анализа нуклеотидных последовательностей
- 2. ПЦРМетод основан на многократном избирательном копировании определённого
- 3. ПЦР
- 4. Компоненты реакции:• ДНК-матрица, содержащая участок ДНК, который
- 5. TmВажнейшая характеристика праймеров — температура плавления (Tm)
- 6. Tmхорошие программы требуют для определения Tm задания двух
- 7. Tа (annealing)Более низкая температура отжига - появление
- 8. При выборе праймеров также желательно придерживаться следующих
- 9. Весьма опасным может быть формирование шпилечных структур
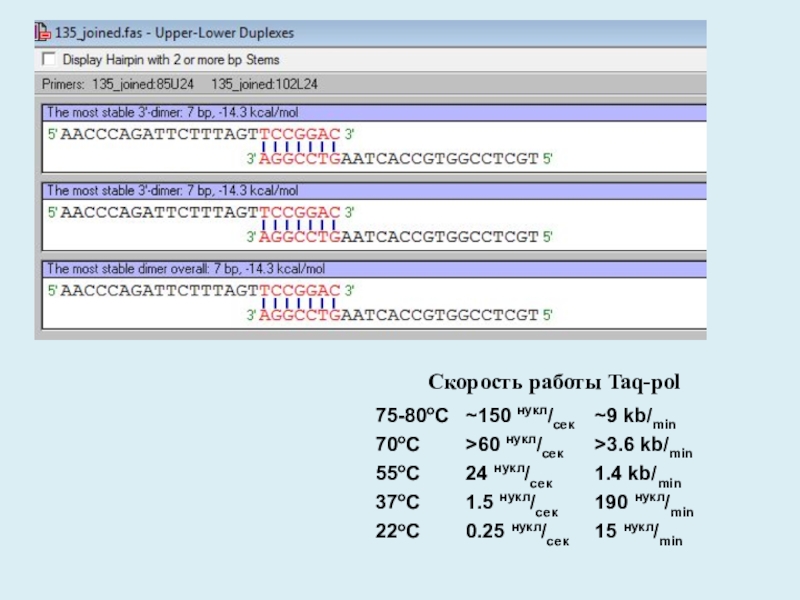
- 10. Cкорость работы Taq-pol
- 11. Слайд 11
- 12. Разновидности ПЦР
- 13. Разновидности ПЦР для рещения проблемы «плохих» праймеровПЦР
- 14. Vector NTI Oligo BioEdit Oligo Calc: Oligonucleotide Properties Calculator http://biotools.nubic.northwestern.edu/OligoCalc.html и т.д.
- 15. Задача 1. Поиск известной последовательности в образцеVector
- 16. Задача 1. Поиск известной последовательности в образцеVector
- 17. Диалоговое окно позволяет сделать следующее:Автоматически подобрать соответствующие
- 18. Слайд 18
- 19. Слайд 19
- 20. Слайд 20
- 21. Слайд 21
- 22. Слайд 22
- 23. Слайд 23
- 24. Слайд 24
- 25. Слайд 25
- 26. Слайд 26
- 27. Заявка на синтез праймеровПоследовательности от 5’ к
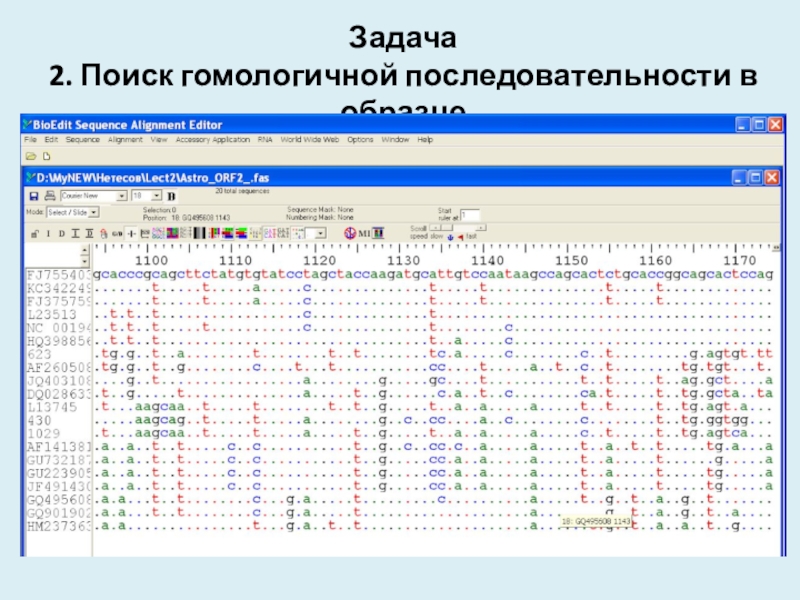
- 28. Задача 2. Поиск гомологичной последовательности в образце
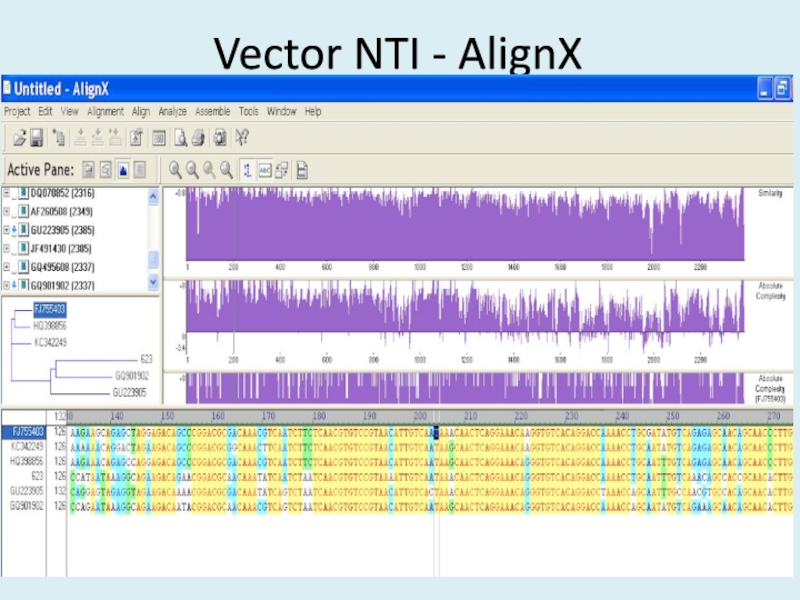
- 29. Vector NTI - AlignX
- 30. Отдельную группу затравочных молекул представляют собой так
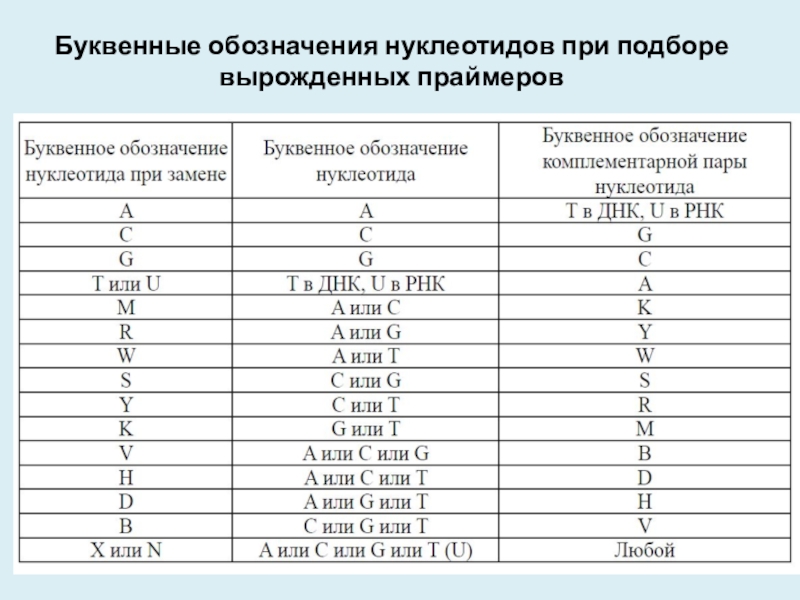
- 31. Буквенные обозначения нуклеотидов при подборе вырожденных праймеров
- 32. Задача 3. Праймеры для целевого ампликона (например, для его клонирования)
- 33. Выбор вектора.Подбор сайтов рестрикции в векторе.Анализ этих
- 34. Слайд 34
- 35. Oligo Calc: Oligonucleotide Properties Calculator http://biotools.nubic.northwestern.edu/OligoCalc.html
- 36. Oligo Calc: Oligonucleotide Properties Calculator http://biotools.nubic.northwestern.edu/OligoCalc.html
- 37. Oligo Calc: Oligonucleotide Properties Calculator http://biotools.nubic.northwestern.edu/OligoCalc.html
- 38. Слайд 38
- 39. Праймеры для секвенированияОптимальная температура плавления 50-600СРасположение праймеров на обеих цепях изучаемой последовательности (перекрывание, длина чтения)
- 40. • Primers should be at least 18
- 41. ABI recommended guidelines when designing primers/probes for
- 42. Слайд 42
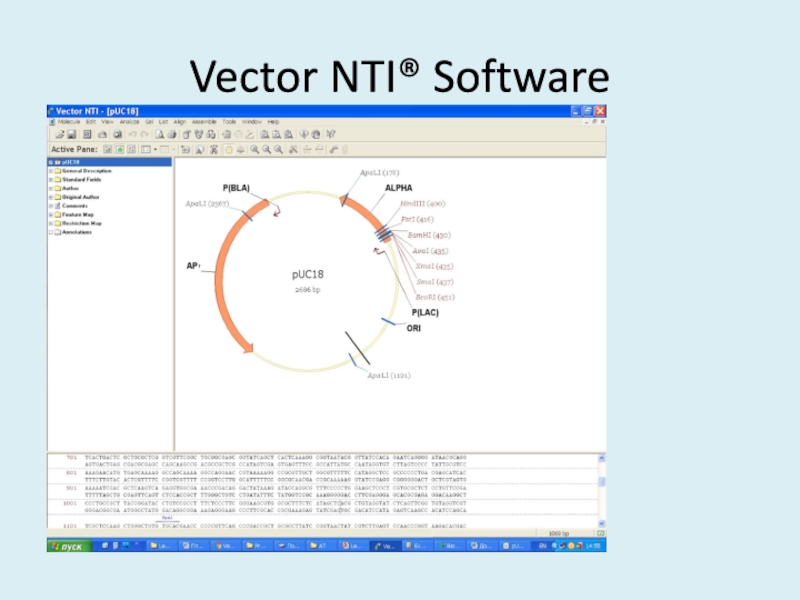
- 43. Vector NTI® Software
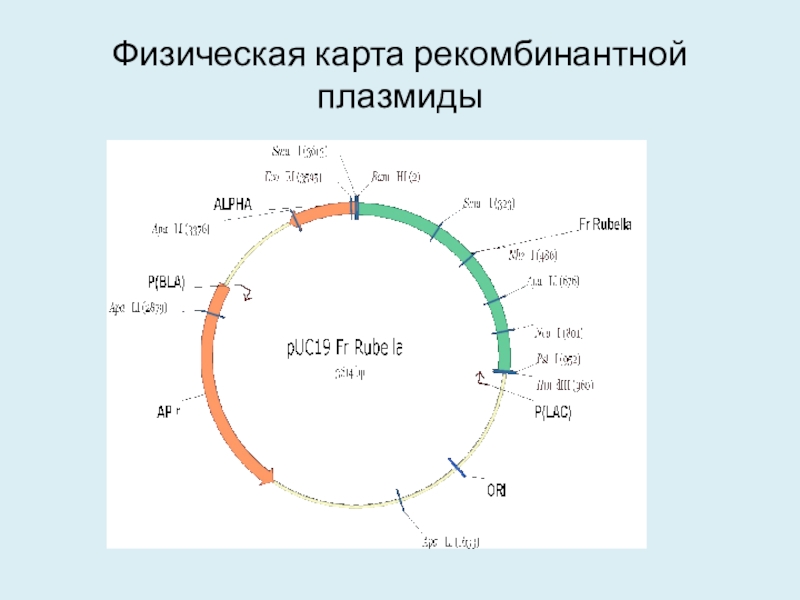
- 44. Физическая карта рекомбинантной плазмиды
- 45. Полезные Интернет-сайты:1. http://www.ncbi.nlm.nih.gov/2. http://www.ebi.ac.uk/3. http://web.expasy.org/4. http://evolution.genetics.washington.edu/phylip/software.html#methods5. http://blast.ncbi.nlm.nih.gov/Blast.cgi6. http://mafft.cbrc.jp/alignment/server/index.html7. http://unafold.rna.albany.edu/?q=mfold/DNA-Folding-Form8. http://molbiol.ru/
- 46. Скачать презентанцию
Слайды и текст этой презентации
Слайд 1Компьютерные методы анализа нуклеотидных последовательностей
Биоинформационные подходы, позволяющие планировать эксперименты.
ПЦР.
Дизайн праймеров для ПЦР и для секвенирования ДНК.
Слайд 2ПЦР
Метод основан на многократном избирательном копировании определённого участка ДНК in
vitro. C помощью ПЦР амплифицируются относительно короткие участки ДНК. В
обычном ПЦР-процессе длина продукта составляет не более 3 т.п.н. С помощью смеси различных полимераз и при определённых условиях длина ПЦР-фрагмента может достигать 20—40 тысяч пар нуклеотидов.Слайд 4Компоненты реакции:
• ДНК-матрица, содержащая участок ДНК, который требуется амплифицировать.
• Два
праймера, комплементарные противоположным концам разных цепей требуемого фрагмента ДНК (Forward-Reverse,
Sense Antisense, Upper-Lower).• Термостабильная ДНК-полимераза
• Дезоксирибонуклеозидтрифосфаты (dATP, dGTP, dCTP, dTTP).
• Ионы Mg2+, необходимые для работы полимеразы.
• Буферный раствор, обеспечивающий необходимые условия реакции
ПЦР
Специфичность ПЦР основана на образовании комплементарных комплексов между матрицей и праймерами, короткими синтетическими олигонуклеотидами длиной 18—30 оснований. Каждый из праймеров комплементарен одной из цепей двуцепочечной матрицы и ограничивает начало и конец амплифицируемого участка.
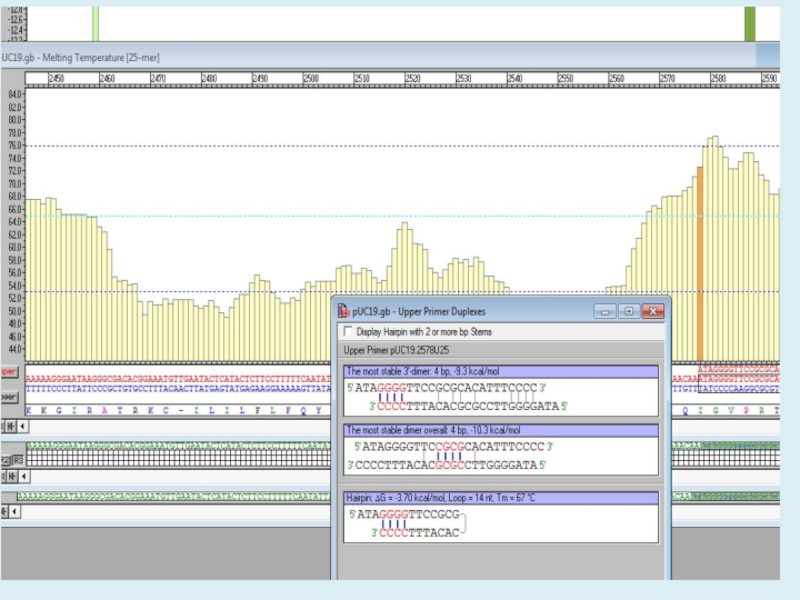
Слайд 5Tm
Важнейшая характеристика праймеров — температура плавления (Tm) комплекса праймер-матрица.
Tm —
температура, при которой половина ДНК-матриц образует комплекс с олигонуклеотидным праймером.
Простой расчет оптимальной температуры отжига праймера:
Tm = [(A+T) x 2 °C] + [(G+C) x 4 °C](если суммарная длина олигонуклеотида не превышает 20 оснований)
Tm = 22 + 1.46([2 x (G+C)] + (A+T))(если суммарная длина олигонуклеотида составляет 20-30 оснований) или
Tm= 64.9 +41 x (G+C-16.4)/(A+T+G+C)
Обычно мы выбираем температуру отжига на 3-5oС меньше, чем температура плавления олигонуклеотидов.
Слайд 6Tm
хорошие программы требуют для определения Tm задания двух параметров:
концентрации праймеров;
концентрации соли
L=G+C+A+T
Tm=
100.5 + (41 x (G+C)/(A+T+G+C)) - (820/(A+T+G+C)) + 16.6 x
log10([Na+]) или для длинных олигонуклеотидов:Tm= 81.5 + (41 x (G+C)/(A+T+G+C)) - (500/(A+T+G+C)) + 16.6 x log10([Na+])
Концентрация соли = [Na+]+[K+]+0.7[Tris].
Слайд 7Tа (annealing)
Более низкая температура отжига - появление неспецифических продуктов. Более
высокая температура отжига - более высокая специфичность. Но как только
она превышает некую критическую (для данной пары праймеров), количество продукта начинает резко снижаться.Слайд 8При выборе праймеров также желательно придерживаться следующих критериев:
GC-состав ~ 40—60
%;
близкие Tm праймеров (отличия не более, чем на 5 °C);
желательно,
чтобы на 3’-конце был гуанин или цитозин / последние несколько нуклеотидов 3' - конца праймера содержали GC-основанияотсутствие неспецифических вторичных структур — шпилек и димеров (особенно на 3’-конце);
Слайд 9Весьма опасным может быть формирование шпилечных структур внутри самого праймера
и образование димеров праймеров, 3'-концы которых образуют прочные связи.
Слайд 13Разновидности ПЦР для рещения проблемы «плохих» праймеров
ПЦР с использованием горячего
старта (Hot-start PCR) .
Ступенчатая ПЦР (Touchdown PCR )
Параметры обычной
PCR реакции.Td=95oC, 1';
25-30 циклов:
Td=94oC, 10'', Ta=55 oC, 30'', Te=72oC, 1';
T=72oC, 5';
T=4oC.
Слайд 14Vector NTI
Oligo
BioEdit
Oligo Calc: Oligonucleotide Properties Calculator
http://biotools.nubic.northwestern.edu/OligoCalc.html
и т.д.
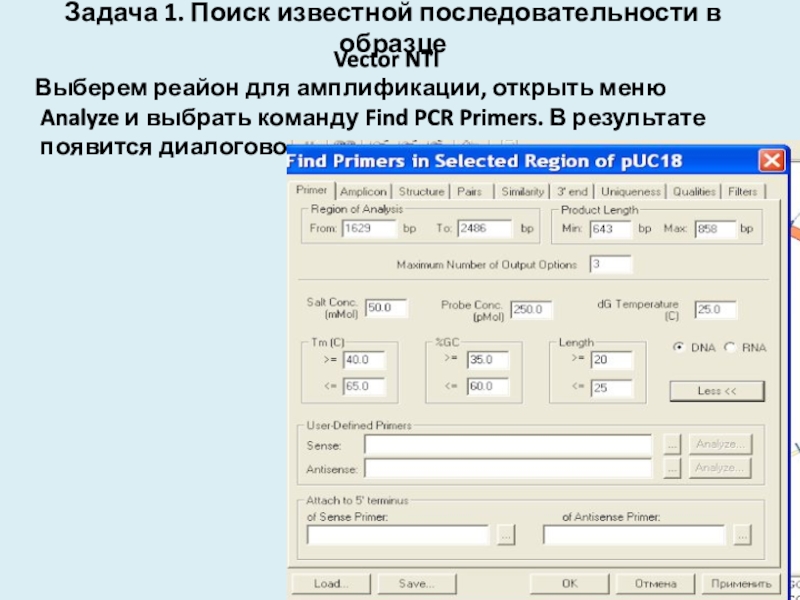
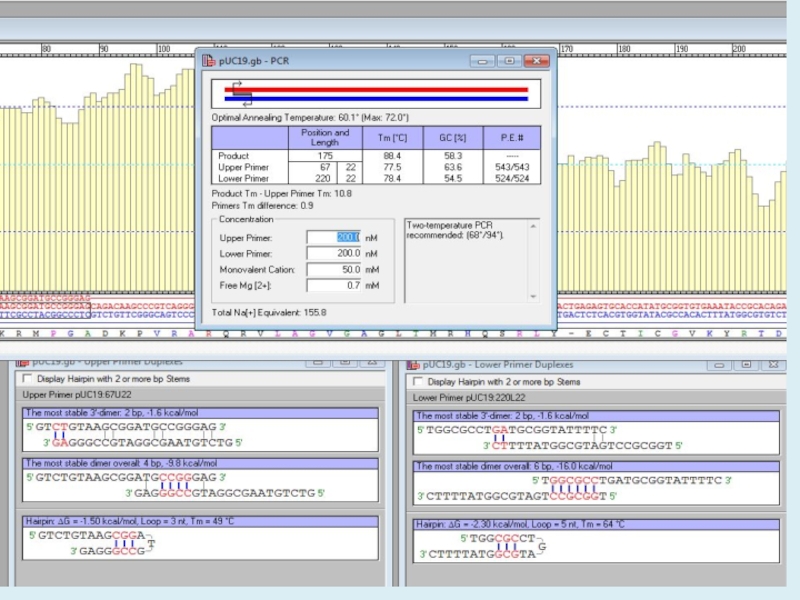
Слайд 15Задача 1. Поиск известной последовательности в образце
Vector NTI
Выберем реайон для
амплификации, открыть меню Analyze и выбрать команду Find PCR Primers.
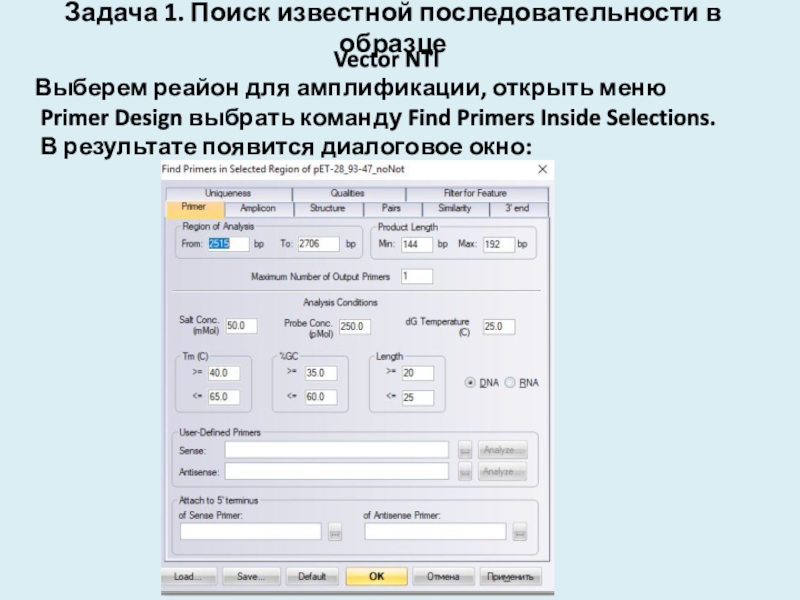
В результате появится диалоговое окно:Слайд 16Задача 1. Поиск известной последовательности в образце
Vector NTI
Выберем реайон для
амплификации, открыть меню Primer Design выбрать команду Find Primers Inside
Selections. В результате появится диалоговое окно:Слайд 17Диалоговое окно позволяет сделать следующее:
Автоматически подобрать соответствующие праймеры;
Задать праймеры собственноручно;
Добавить
к 5’ концу праймера короткую последовательность;
Сформулировать требования к гомологии праймера
(Primer Similarity);Задать биохимические и структурные параметры праймеров;
Определить требования к качеству праймеров.
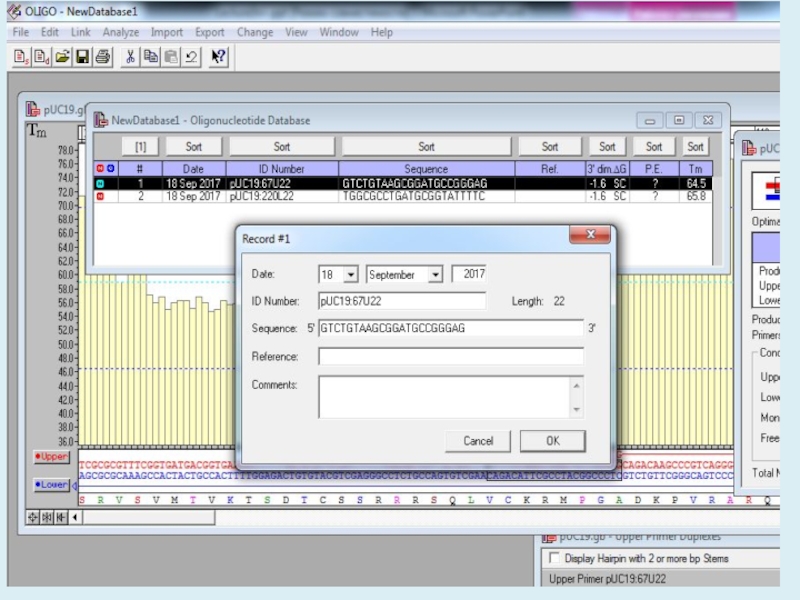
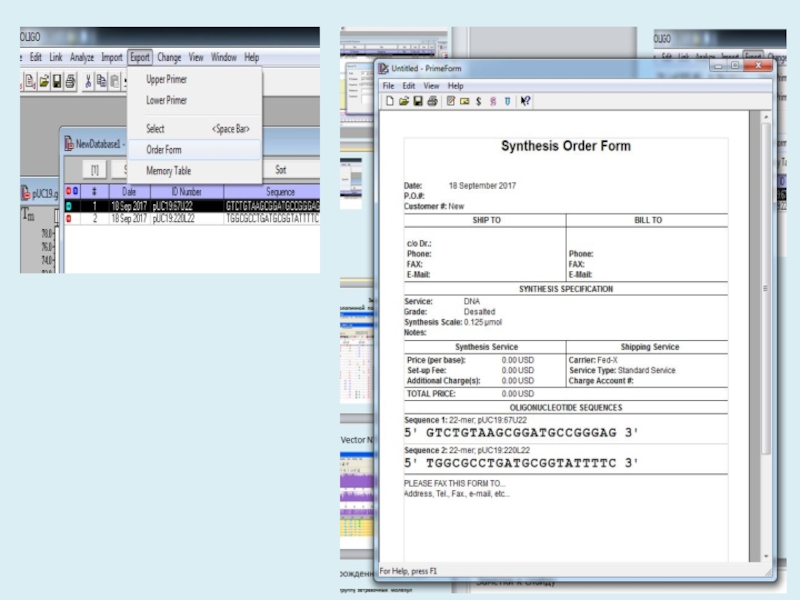
Слайд 27Заявка на синтез праймеров
Последовательности от 5’ к 3’ концу (Antisense
праймер необходимо записать как обратно комплиментарный)
«Буквы» только английские
Масштаб синтеза и
требуемый вид очистки Слайд 30Отдельную группу затравочных молекул представляют собой так называемые вырожденные праймеры,
составленные или на основе относительно консервативной последовательности нуклеотидов какого-либо гена
из мультигенного семейства. Зачастую такой праймер будет представлять собой весьма гетерогенную смесь олигонуклеотидов. Применение остатков инозина, образующего комплементарные пары с любым из четырех дезоксинуклеотидов, в вырожденных местах таких праймеров в значительной степени решает эту проблемуВырожденные праймеры
Слайд 33Выбор вектора.
Подбор сайтов рестрикции в векторе.
Анализ этих сайтов в целевом
фрагменте.
В случае слитых последовательностей – проверка непрерывности ОРТ.
Расчет праймеров и
введение последовательностей сайтов рестрикции на 5' концах праймеров (дополнительные нуклеотиды для работы ферментов.https://www.neb.com/
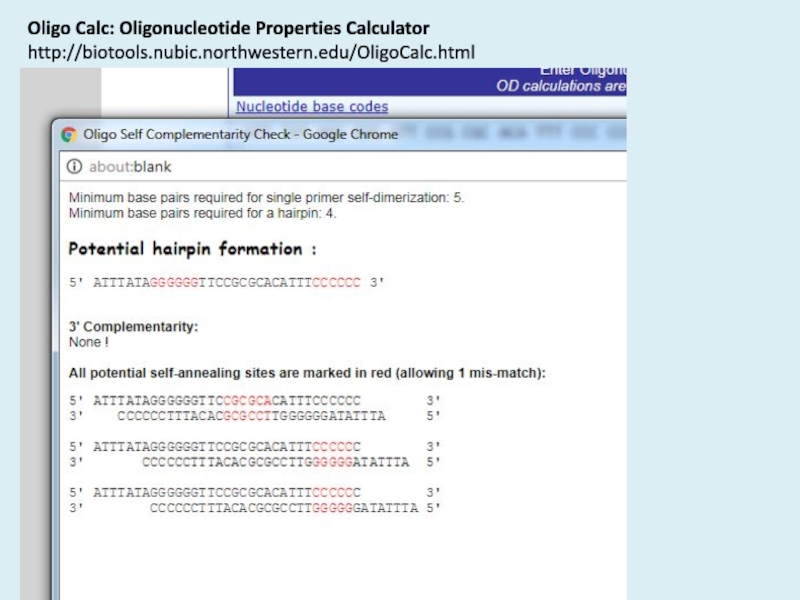
Слайд 35Oligo Calc: Oligonucleotide Properties Calculator http://biotools.nubic.northwestern.edu/OligoCalc.html
Слайд 36Oligo Calc: Oligonucleotide Properties Calculator http://biotools.nubic.northwestern.edu/OligoCalc.html
Слайд 37Oligo Calc: Oligonucleotide Properties Calculator http://biotools.nubic.northwestern.edu/OligoCalc.html
Слайд 39Праймеры для секвенирования
Оптимальная температура плавления 50-600С
Расположение праймеров на обеих цепях
изучаемой последовательности (перекрывание, длина чтения)
Слайд 40• Primers should be at least 18 bases long to
ensure good hybridization.
• Avoid runs of an identical nucleotide, especially
guanine, where runs of four or more Gs should be avoided. Избегать 4 и более G
• Keep the G-C content in the range 30–80%.
• For cycle sequencing, primers with melting temperatures (Tm) above 45 °C produce better results than primers with lower Tm.
• For primers with a G-C content less than 50%, it may be necessary to extend the primer sequence beyond 18 bases to keep the Tm>45 °C.
• Use of primers longer than 18 bases also minimizes the chance of having a secondary hybridization site on the target DNA.
• Avoid primers that have secondary structure or that can hybridize to form dimers.
• Several computer programs for primer selection are available.
Слайд 41ABI recommended guidelines when designing primers/probes for Quantitative assay
· select the
probe first and design the primers as close as possible
to the probe without overlapping it. Amplicons of 50 to 150 bp are strongly recommended. · keep primer/probe GC content within 30-80% · avoid runs of identical nucleotides, this is especially true for guanine, where runs of four or more Gs should be avoidedWhen designing primers: · Tm should be within 58°C to 60°C · the last 5 bases at the 3 prime end should have no more than two G's or C's · keep the annealing Ts of the primers as close as possible · select primer pairs with minimal number of potential primer dimers and primer hairpins as possible
When designing probes: · Tm should be within 68°C to 70°C · no Gs on the 5’ end · select the strand that gives the probe more C than G bases · make the TaqMan probes as short as possible, without being shorter than 13 nucleotides