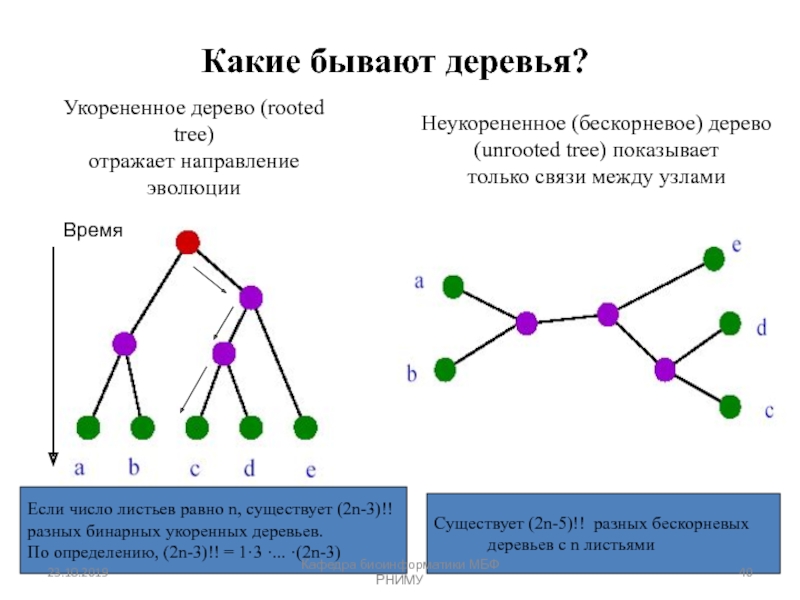
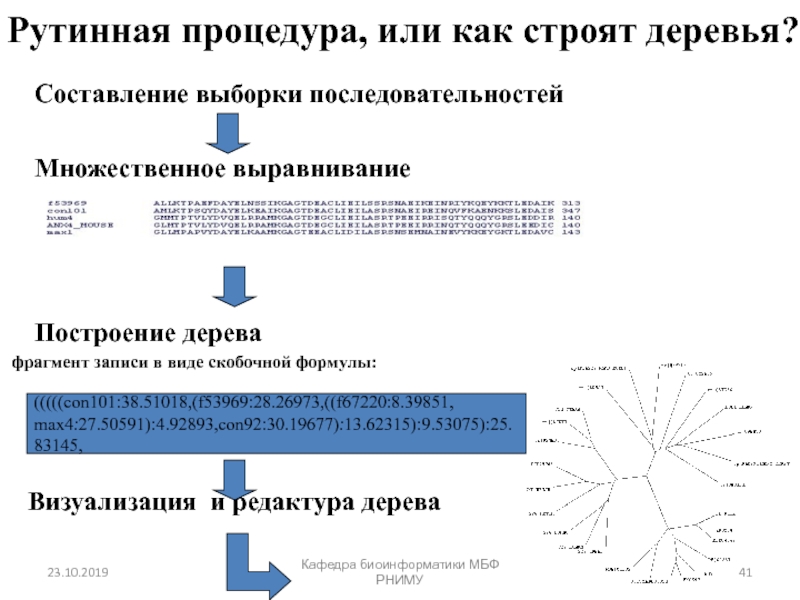
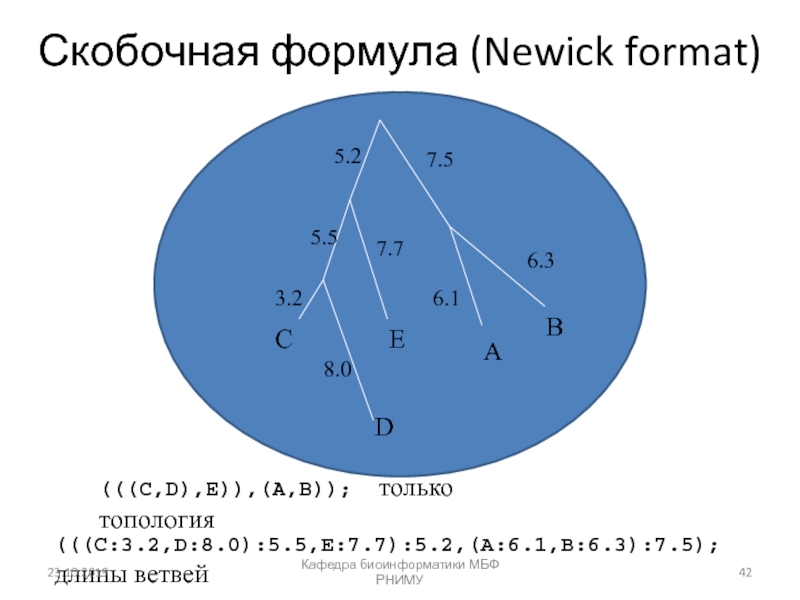
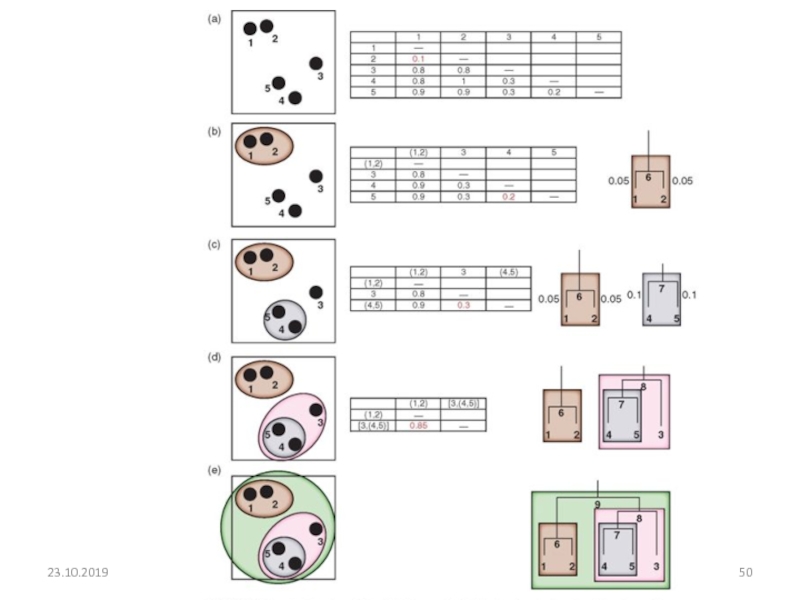
естественного отбора — эволюционным силам, факторам, направленным на отбор такой
мутации, ее преимущественное закрепление в популяции.
Мутации, ухудшающие приспособленность организма, подвергаются действию отрицательного естественного отбора — эволюционным силам, факторам, направленным на преимущественное удаление такой мутации из популяции.
Мутации, не изменяющие приспособленности организма к окружающей среде, называют селективно нейтральными.
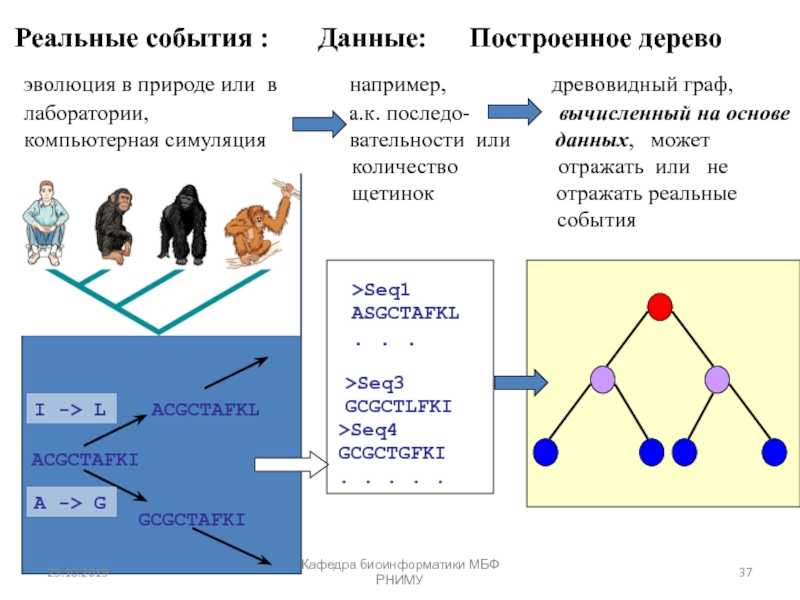
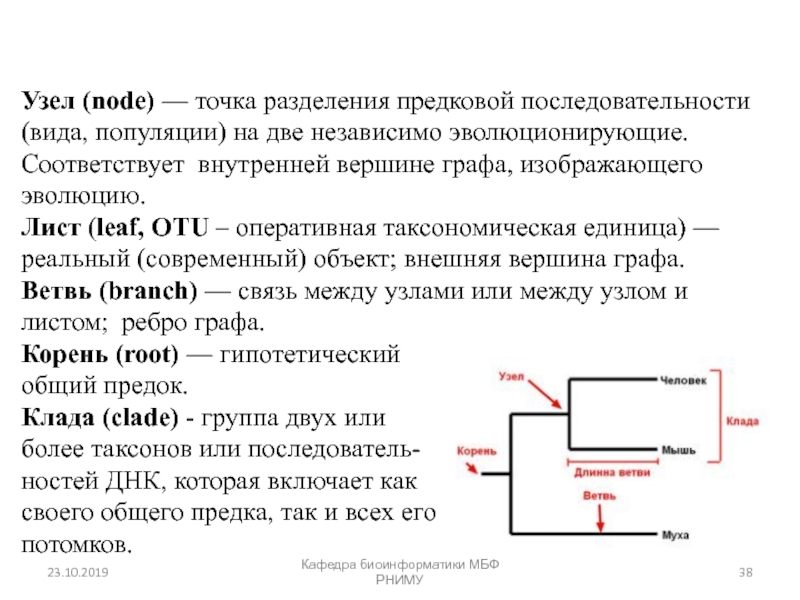
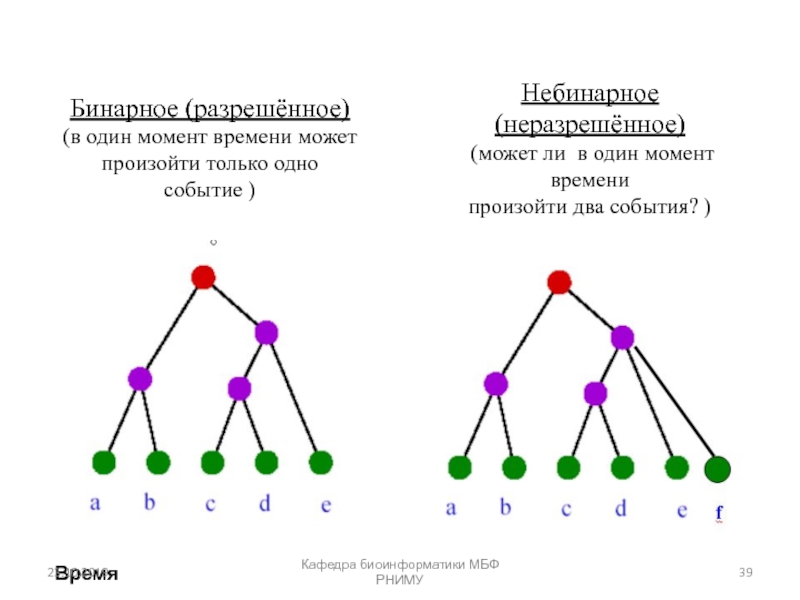
Процесс изменения частоты мутации в популяции под действием стохастических процессов называют случайным генетическим дрейфом (random genetic drift).
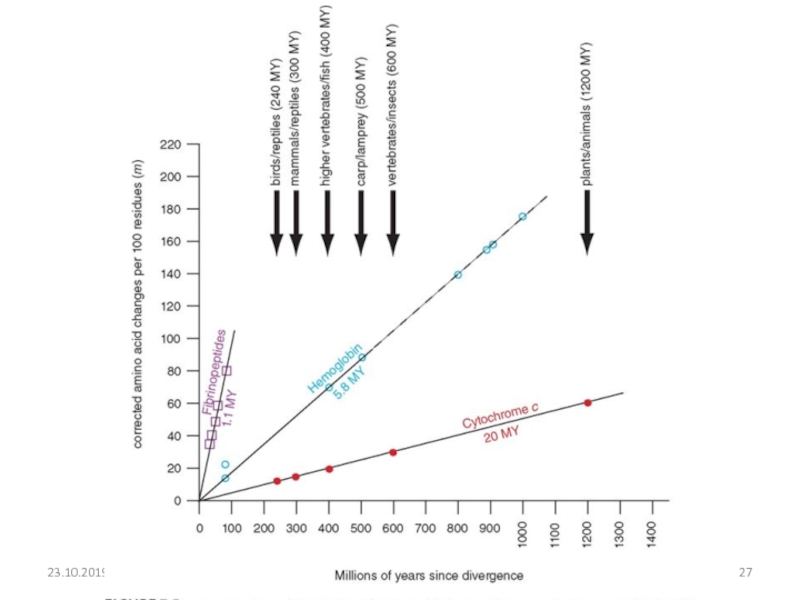
Для кодирующих нуклеотидных последовательностей основными факторами естественного отбора являются эволюционные факторы, действующие на уровне белка. Соответственно действию этих факторов подвергаются несинонимичные, изменяющие белок, а не синонимичные замены.
В целом синонимичные замены принято считать селективно нейтральными, или близкими к нейтральным.
Кафедра биоинформатики МБФ РНИМУ