Разделы презентаций
- Разное
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Геометрия
- Детские презентации
- Информатика
- История
- Литература
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
МОЛЕКУЛЯРНА БІОТЕХНОЛОГІЯ. ЛЕКЦІЯ №1. ВСТУП. ІНСТРУМЕНТИ МОЛЕКУЛЯРНОЇ
Содержание
- 1. МОЛЕКУЛЯРНА БІОТЕХНОЛОГІЯ. ЛЕКЦІЯ №1. ВСТУП. ІНСТРУМЕНТИ МОЛЕКУЛЯРНОЇ
- 2. ПЛАН ЛЕКЦІЇРестриктази – загальна характеристика. Фактори, які
- 3. ПЛАН ЛЕКЦІЇФрагмент Кльонова (Exo-).ДНК-полімерази фагів Т4 та
- 4. РЕСТРИКТАЗИ – ОДНІ З ОСНОВНИХ ФЕРМЕНТІВ МОЛЕКУЛЯРНОЇ БІОТЕХНОЛОГІЇ
- 5. ВІДКРИТТЯ РЕСТРИКТАЗHindII – перша рестриктаза, яка була
- 6. Система метилювання-рестрикції (МР-система) мікроорганізмів (а) – чужорідна
- 7. Система метилювання-рестрикції (МР-система) мікроорганізмів (b) – ДНК E. сoli метильована за сайтом EcoRI
- 8. Система метилювання-рестрикції (МР-система) мікроорганізмів (b) – ДНК E. coli не деградується рестриктазами
- 9. Система метилювання-рестрикції (МР) Захищає прокаріотичну клітину від
- 10. ЯК УТВОРЮЮТЬСЯ НАЗВИ РЕСТРИКТАЗ:Назва виду бактерій, в
- 11. ПРИКЛАДИ РЕСТРИКТАЗ0
- 12. ЯК УТВОРЮЮТЬСЯ НАЗВИ РЕСТРИКТАЗ:
- 13. ПРИКЛАДИ СЛІВ-ПАЛІНДРОМІВ (ДАЛЕКО НЕ УСІ РЕСТРИКТАЗИ ГІДРОЛІЗУЮТЬ ДНК В ПАЛІНДРОМАХ)ЗаразАннаПилипзараЗаннАпилиП
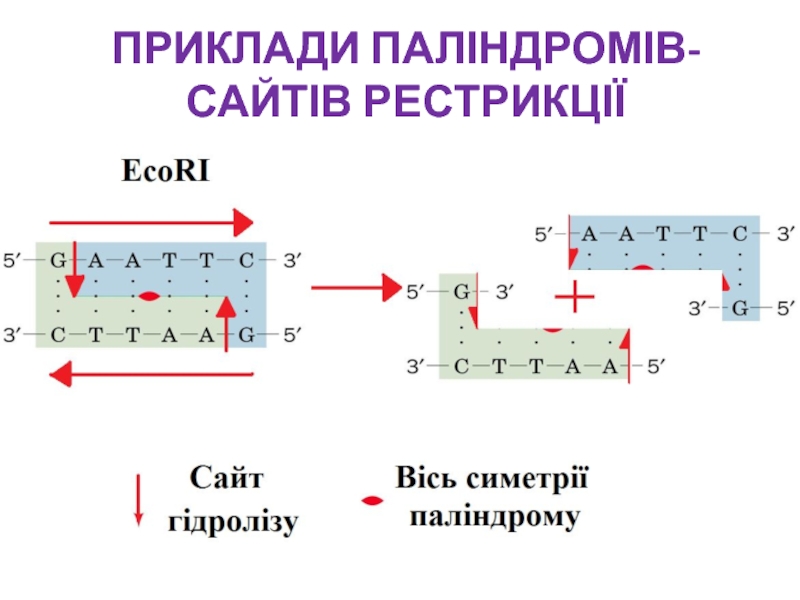
- 14. ПРИКЛАДИ ПАЛІНДРОМІВ-САЙТІВ РЕСТРИКЦІЇ
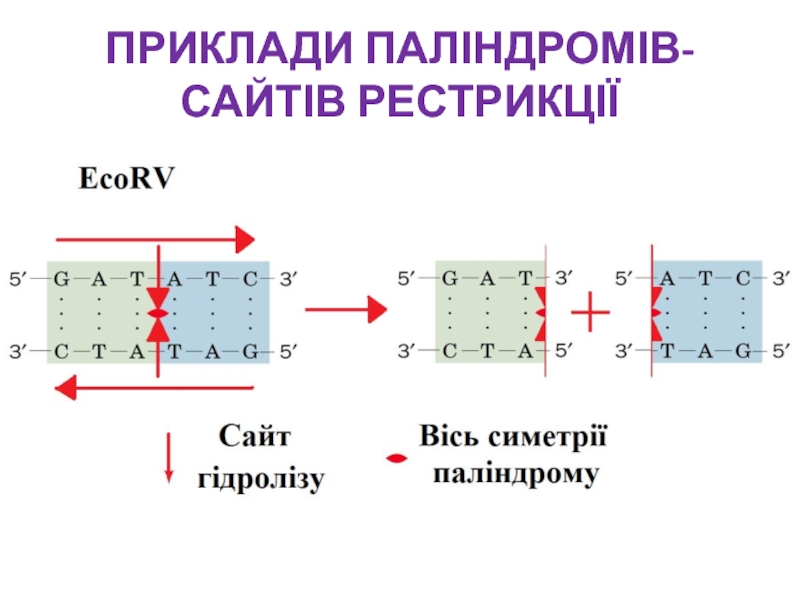
- 15. ПРИКЛАДИ ПАЛІНДРОМІВ-САЙТІВ РЕСТРИКЦІЇ
- 16. Сайт рестрикції більшості рестриктаз, окрім одномірної симетрії
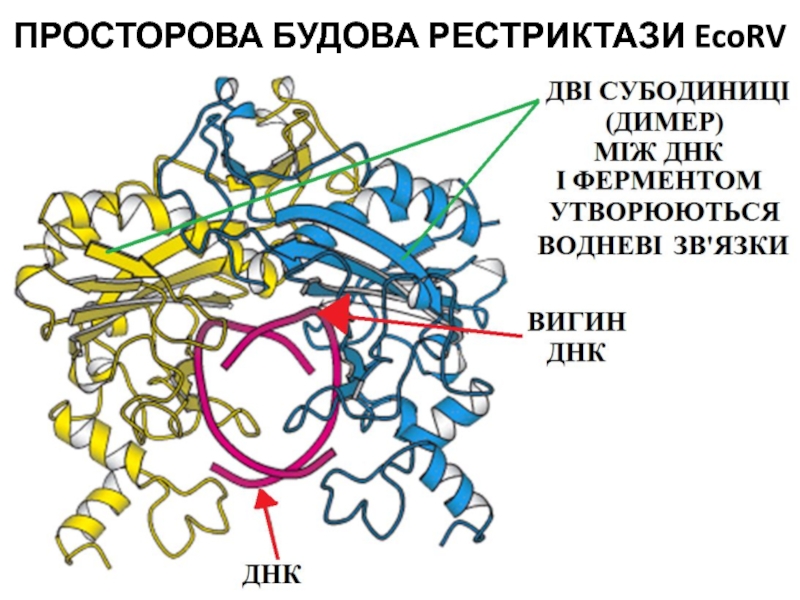
- 17. ПРОСТОРОВА БУДОВА РЕСТРИКТАЗИ EcoRV
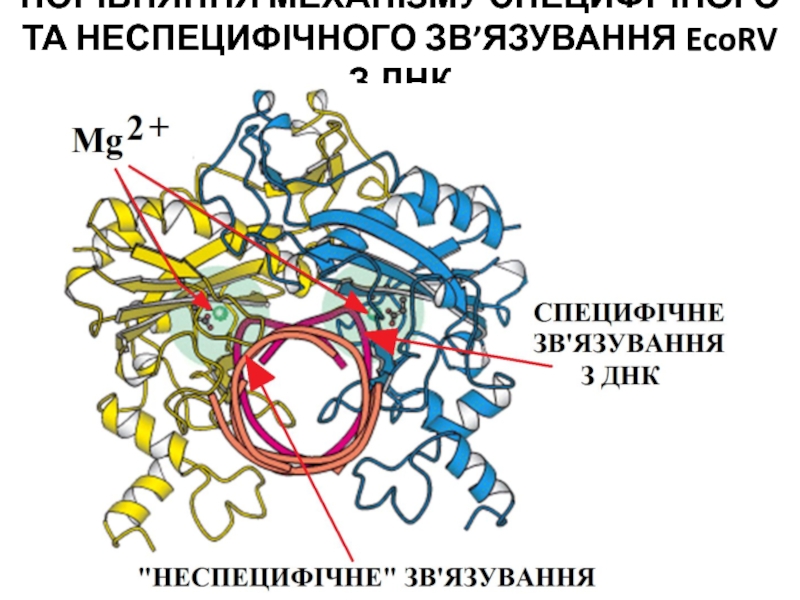
- 18. ПОРІВНЯННЯ МЕХАНІЗМУ СПЕЦИФІЧНОГО ТА НЕСПЕЦИФІЧНОГО ЗВ’ЯЗУВАННЯ EcoRV З ДНК
- 19. МЕХАНІЗМ ДІЇ РЕСТРИКТАЗ НА ДНК ,,Сканування” ДНК;
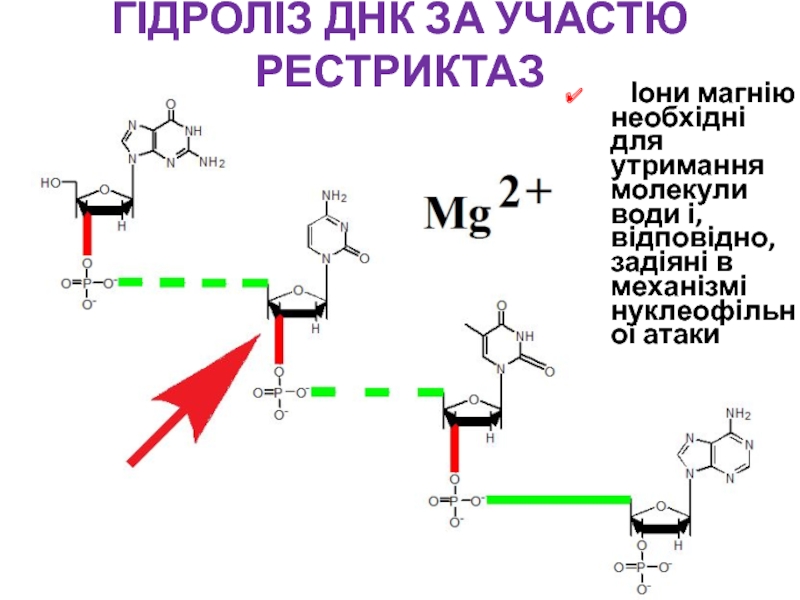
- 20. ГІДРОЛІЗ ДНК ЗА УЧАСТЮ РЕСТРИКТАЗ Іони
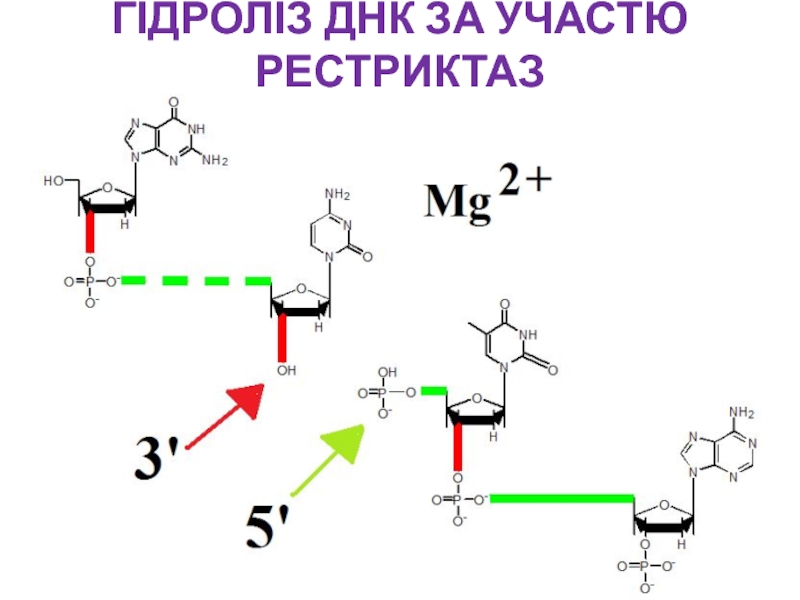
- 21. ГІДРОЛІЗ ДНК ЗА УЧАСТЮ РЕСТРИКТАЗ
- 22. Рестриктази I-го типу Являють собою олігомерний білок,
- 23. Рестриктази IІІ-го типу Являють собою олігомерний білок,
- 24. Рестриктази IV-го типуДосліджені недостатньо; Складаються з одного
- 25. Рестриктази IІ-го типуШироко використовуються в бтх:Розпізнають паліндромні
- 26. Рестриктази IІ-го типу підкласу ІІРБільше половини усіх
- 27. Рестриктази IІ-го типу підкласів ІІW і IINІІW
- 28. Рестриктази IІ-го типу підкласів ІІS і IITІІS
- 29. РЕСТРИКТАЗИ ІІ ТИПУ - УЗАГАЛЬНЕННЯ Найбільш широко
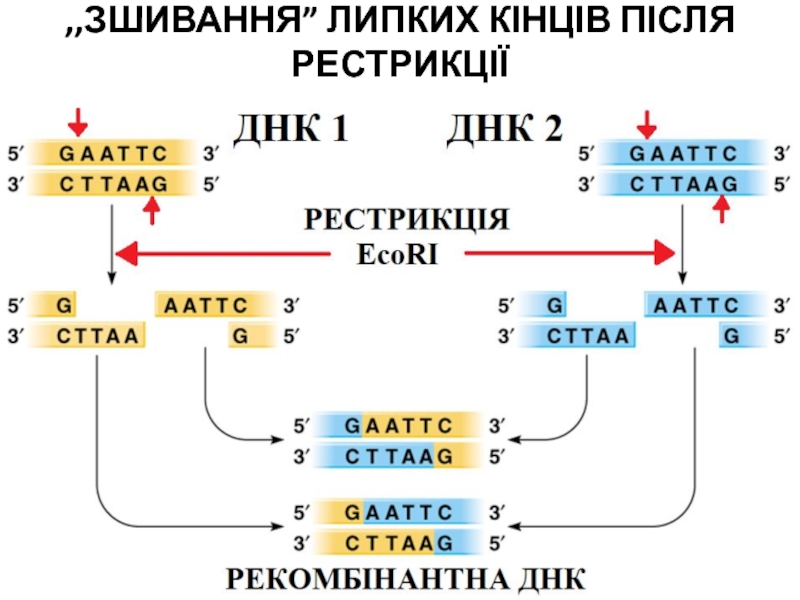
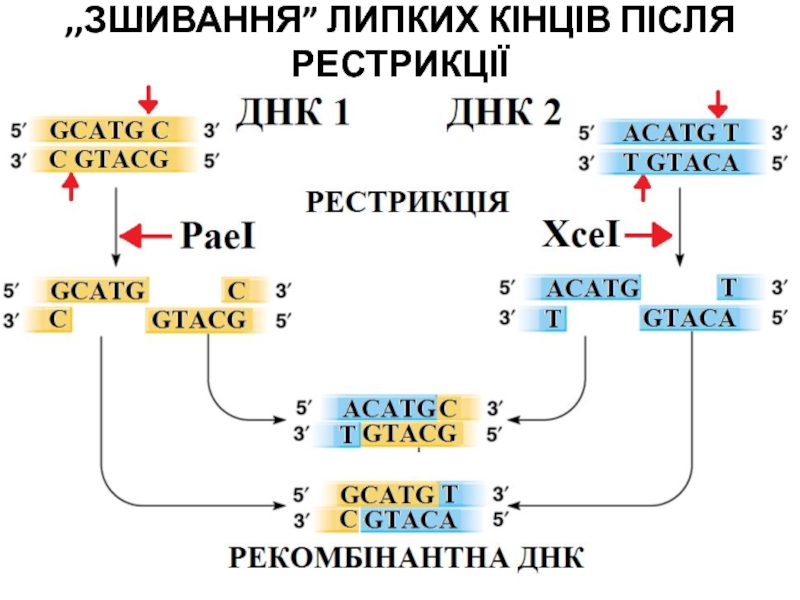
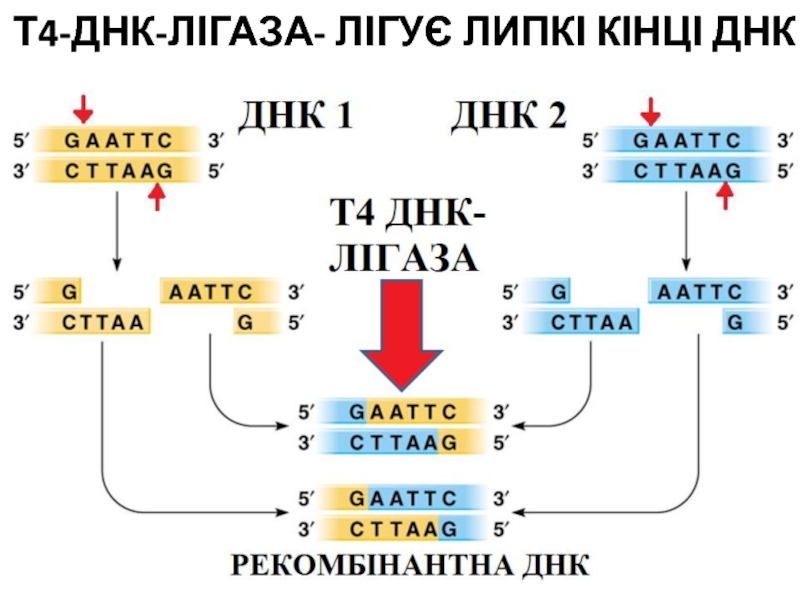
- 30. ,,ЗШИВАННЯ” ЛИПКИХ КІНЦІВ ПІСЛЯ РЕСТРИКЦІЇ
- 31. ,,ЗШИВАННЯ” ЛИПКИХ КІНЦІВ ПІСЛЯ РЕСТРИКЦІЇ
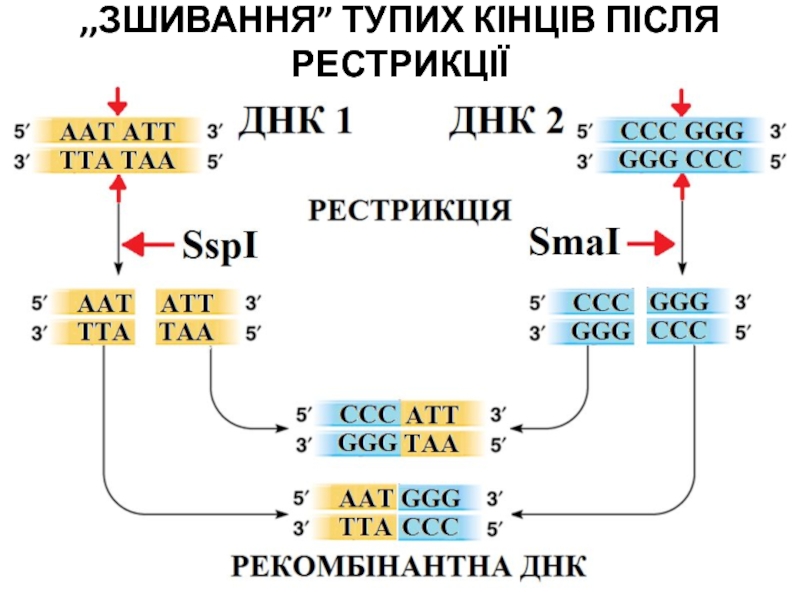
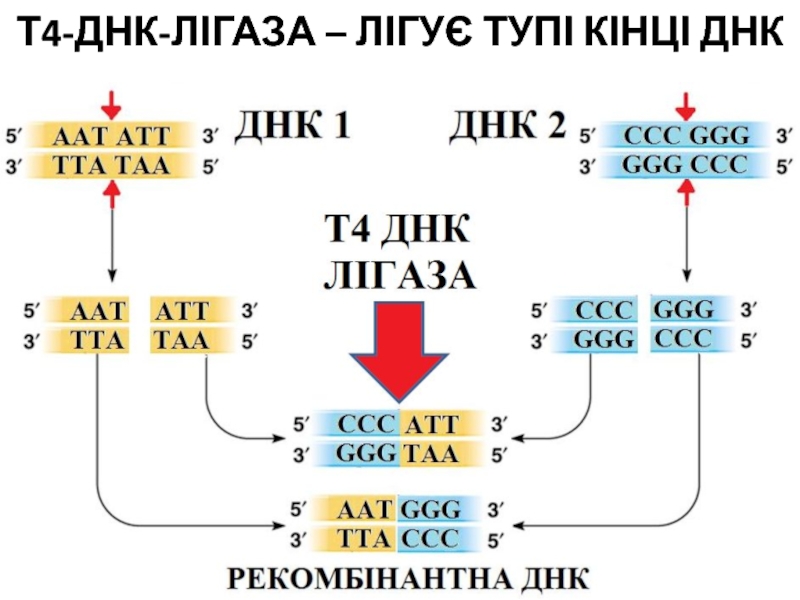
- 32. ,,ЗШИВАННЯ” ТУПИХ КІНЦІВ ПІСЛЯ РЕСТРИКЦІЇ
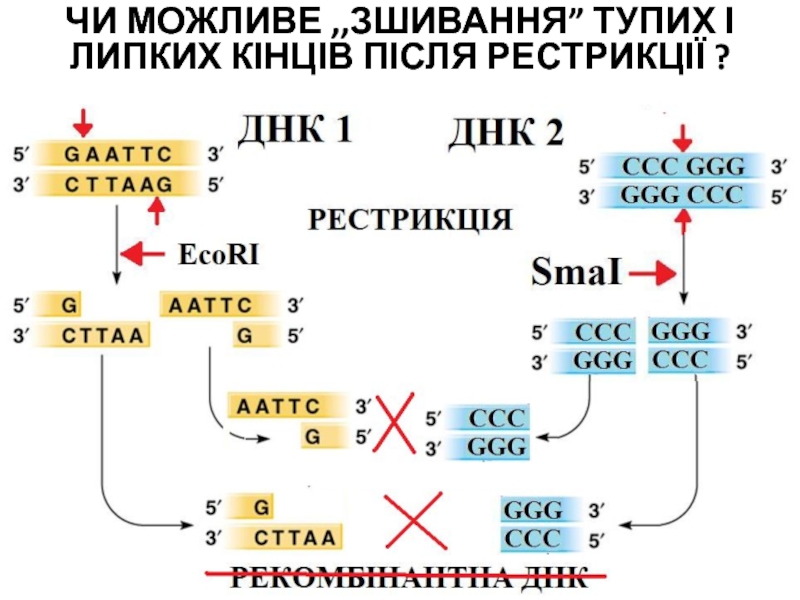
- 33. ЧИ МОЖЛИВЕ ,,ЗШИВАННЯ” ТУПИХ І ЛИПКИХ КІНЦІВ ПІСЛЯ РЕСТРИКЦІЇ ?
- 34. ФАКТОРИ, ЯКІ ВПЛИВАЮТЬ НА РОБОТУ РЕСТРИКТАЗ: DAM- ТА DCM-МЕТИЛЮВАННЯ
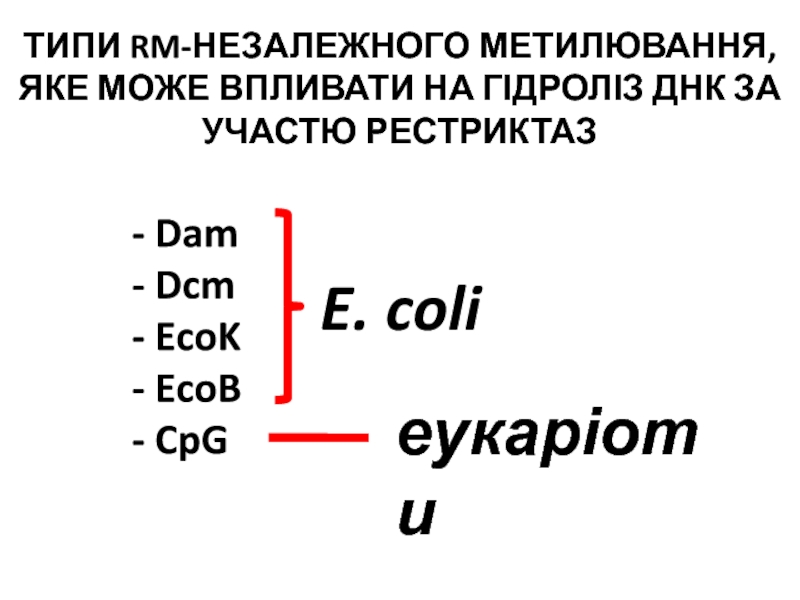
- 35. ТИПИ RM-НЕЗАЛЕЖНОГО МЕТИЛЮВАННЯ, ЯКЕ МОЖЕ ВПЛИВАТИ НА
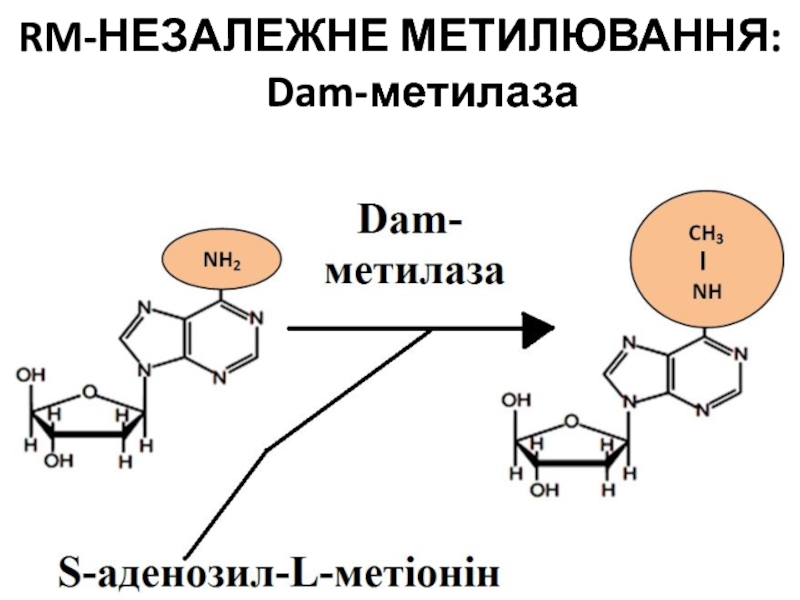
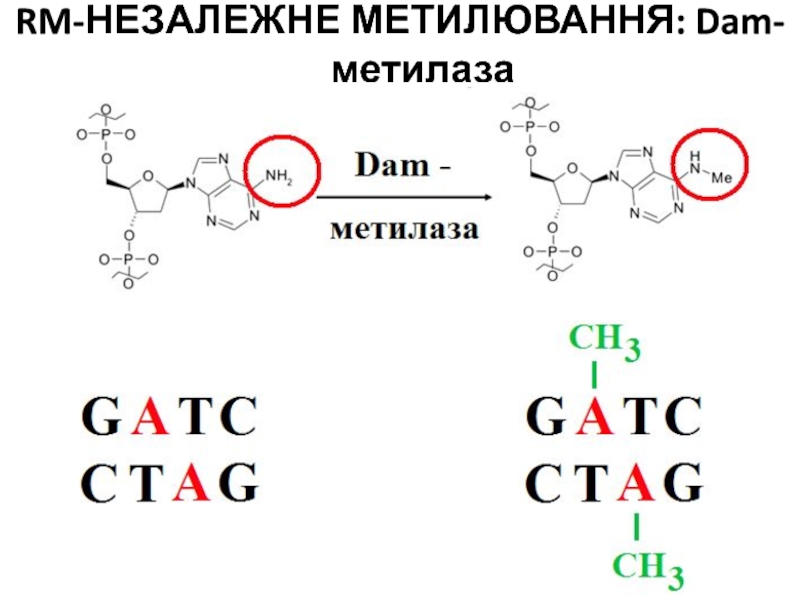
- 36. RM-НЕЗАЛЕЖНЕ МЕТИЛЮВАННЯ: Dam-метилаза
- 37. RM-НЕЗАЛЕЖНЕ МЕТИЛЮВАННЯ: Dam-метилаза
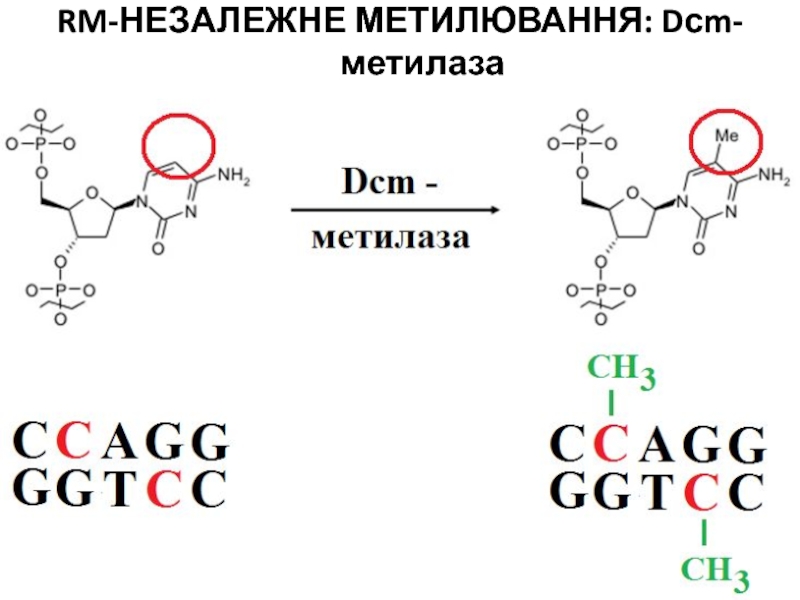
- 38. RM-НЕЗАЛЕЖНЕ МЕТИЛЮВАННЯ: Dсm-метилаза
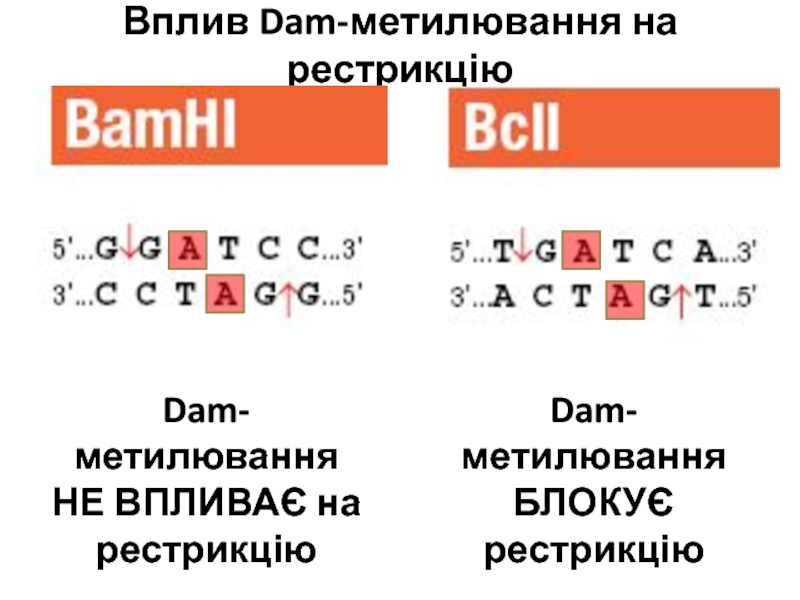
- 39. 0Вплив Dam-метилювання на рестрикціюDam-метилювання НЕ ВПЛИВАЄ на рестрикціюDam-метилювання БЛОКУЄ рестрикцію
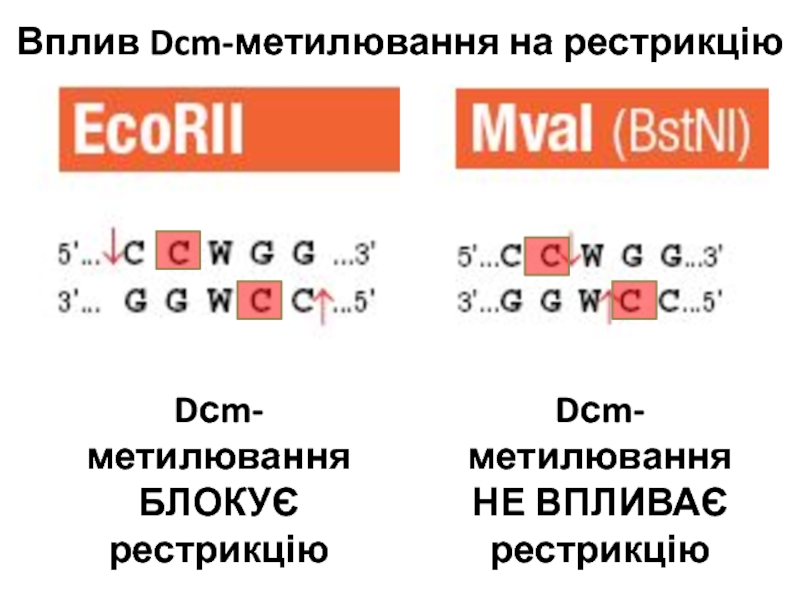
- 40. 0Вплив Dcm-метилювання на рестрикціюDсm-метилювання БЛОКУЄ рестрикціюDсm-метилювання НЕ ВПЛИВАЄ рестрикцію
- 41. ,,ІЗОМЕРІЯ” РЕСТРИКТАЗ
- 42. Ізошизомери і гетерошизомериІЗОМЕРИ – рестриктази різного походження,
- 43. Ізошизомери і гетерошизомери0
- 44. РЕСТРИКТАЗИ: ПОБУДОВА РЕСТРИКЦІЙНИХ КАРТ
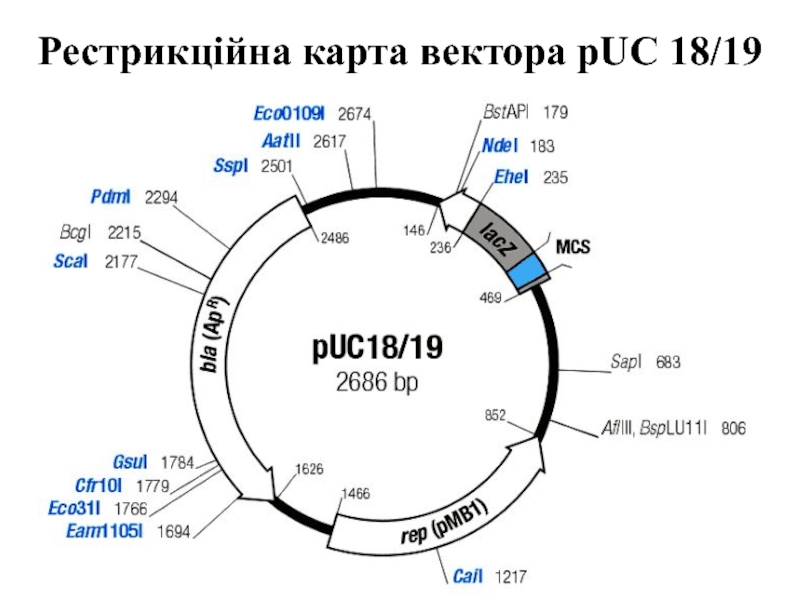
- 45. Рестрикційна карта вектора pUC 18/19
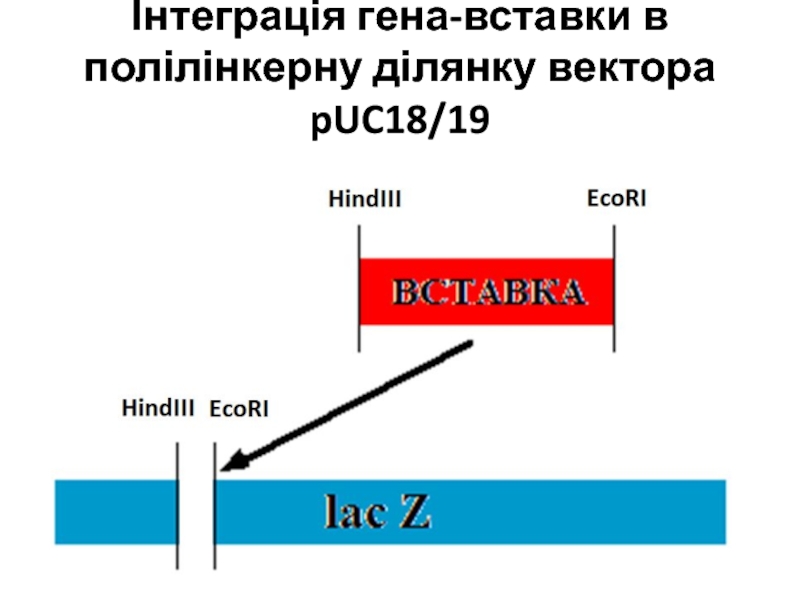
- 46. Інтеграція гена-вставки в полілінкерну ділянку вектора pUC18/19
- 47. ПОБУДОВА РЕСТРИКЦІЙНОЇ КАРТИ кДНК (мРНК) ІНСУЛІНУ ЛЮДИНИ (NCBI Reference Sequence: NM_000207.2)
- 48. РЕСУРС PubMed http://www.ncbi.nlm.nih.gov шукаємо послідовність мРНК інсуліну в форматі FASTA0
- 49. РЕСУРС PubMed http://www.ncbi.nlm.nih.gov шукаємо послідовність мРНК інсуліну в форматі FASTA0
- 50. РЕСУРС PubMed http://www.ncbi.nlm.nih.gov шукаємо послідовність мРНК інсуліну
- 51. РЕСУРС PubMed http://www.ncbi.nlm.nih.gov шукаємо послідовність мРНК інсуліну в форматі FASTA0ID ПОСЛІДОВНОСТІFASTA
- 52. РЕСУРС PubMed http://www.ncbi.nlm.nih.gov шукаємо послідовність мРНК інсуліну в форматі FASTA0FASTA
- 53. РЕСУРС NEB-CUTTER http://nc2.neb.com/NEBcutter2/ вводимо послідовність мРНК інсуліну в форматі FASTA0
- 54. РЕСУРС NEB-CUTTER http://nc2.neb.com/NEBcutter2/ вводимо послідовність мРНК інсуліну в форматі FASTA0
- 55. РЕСУРС NEB-CUTTER Унікальні сайти рестрикції мРНК інсуліну (NCBI Reference Sequence: NM_000207.2)0
- 56. РЕСУРС NEB-CUTTERСайти рестрикції мРНК інсуліну (NCBI Reference Sequence: NM_000207.2), які зустрічаються двічі 0
- 57. РЕСУРС NEB-CUTTERСайти рестрикції мРНК інсуліну (NCBI Reference Sequence: NM_000207.2), які зустрічаються тричі0
- 58. РЕСУРС NEB-CUTTER http://nc2.neb.com/NEBcutter2/ ДОДАТКОВІ МОЖЛИВОСТІ0
- 59. РЕСУРС NEB-CUTTER http://nc2.neb.com/NEBcutter2/ ДОДАТКОВІ МОЖЛИВОСТІ0
- 60. ЗАВДАННЯ ДЛЯ САМОСТІЙНОЇ РОБОТИ: ОТРИМАТИ РЕСТРИКЦІЙНІ КАРТИ
- 61. ЛІГАЗИ: ЗАГАЛЬНА ХАРАКТЕРИСТИКА, ВИДИ ТА МЕХАНІЗМ РЕАКЦІЇ
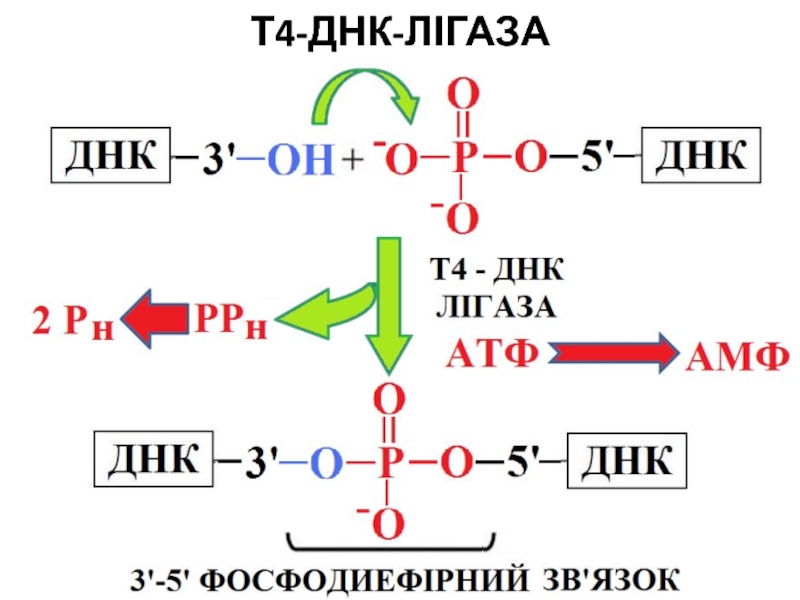
- 62. Т4-ДНК-ЛІГАЗА
- 63. Ферментативні активності Т4-ДНК лігазиКаталізує утворення фосфодиефірного зв’язку
- 64. Т4-ДНК-ЛІГАЗА- ЛІГУЄ ЛИПКІ КІНЦІ ДНК
- 65. Т4-ДНК-ЛІГАЗА – ЛІГУЄ ТУПІ КІНЦІ ДНК
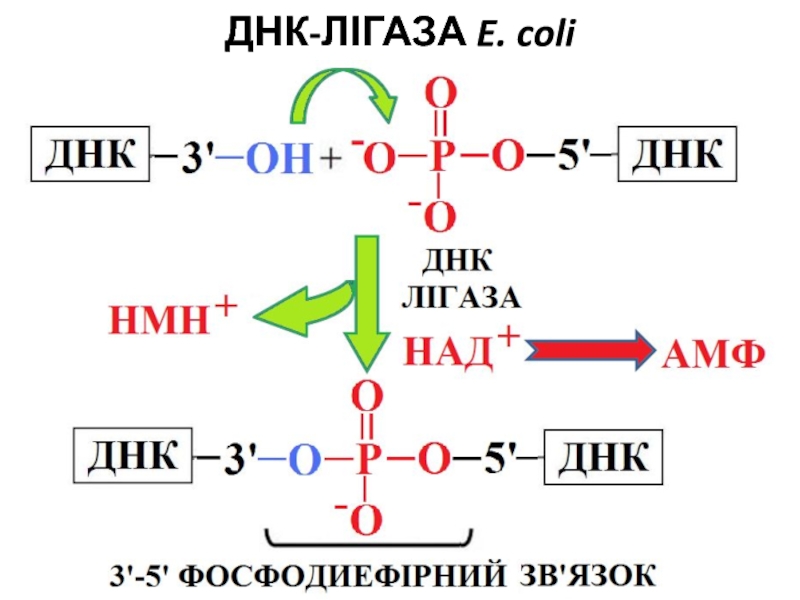
- 66. ДНК-ЛІГАЗА E. coliH O 2Мg2+
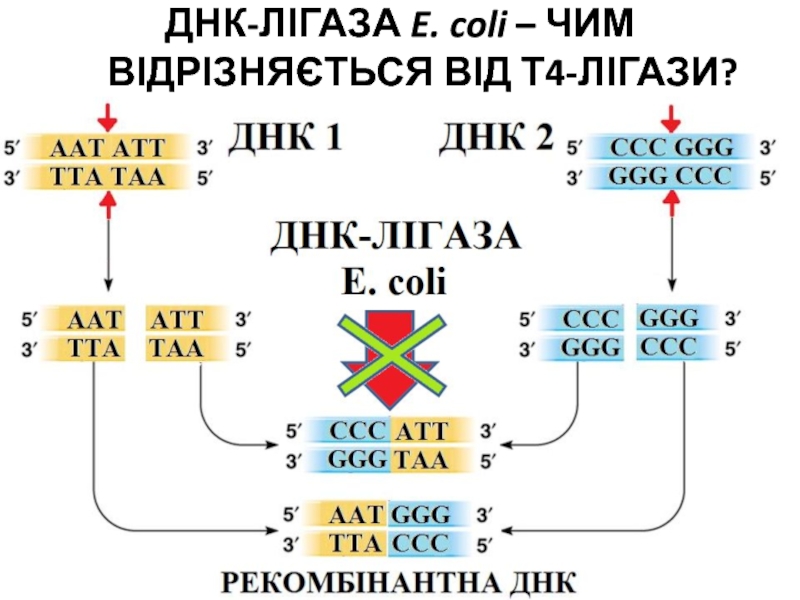
- 67. ДНК-ЛІГАЗА E. coli – ЧИМ ВІДРІЗНЯЄТЬСЯ ВІД Т4-ЛІГАЗИ?
- 68. ВИКОРИСТАННЯ ЛІГАЗ В МОЛЕКУЛЯРНІЙ БІОТЕХНОЛОГІЇКлонування фрагментів (зокрема,
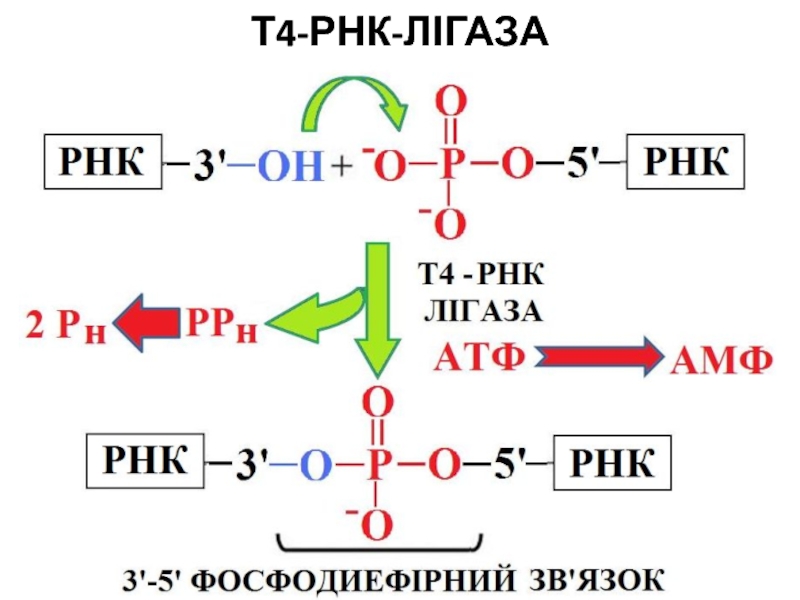
- 69. Т4 РНК-ЛІГАЗАТ4 РНК лігаза каталізує утворення фосфодиефірних
- 70. Т4-РНК-ЛІГАЗА
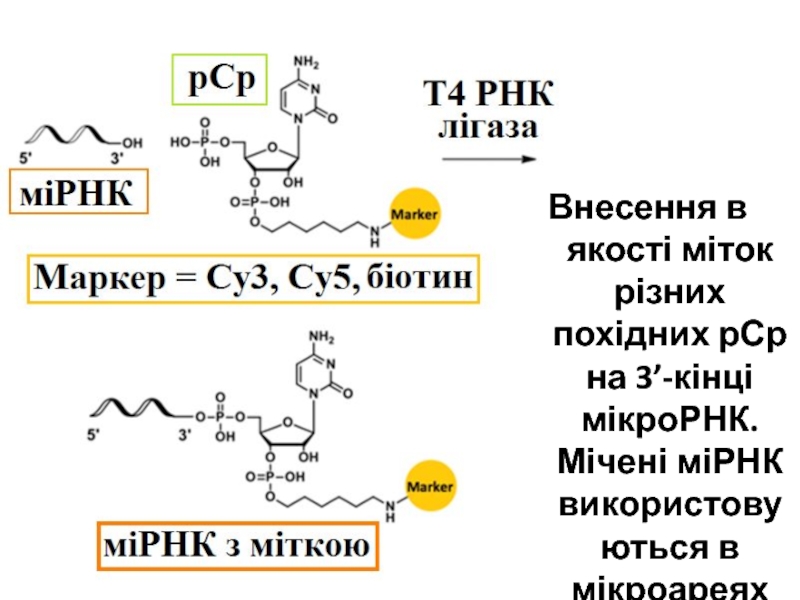
- 71. ВИКОРИСТАННЯ Т4 РНК-ЛІГАЗИВнесення міток на 3’-кінці РНК
- 72. Внесення в якості міток різних похідних рСр на 3’-кінці мікроРНК. Мічені міРНК використовуються в мікроареях
- 73. ФОСФАТАЗИ: ЗАГАЛЬНА ХАРАКТЕРИСТИКА, ВИКОРИСТАННЯ
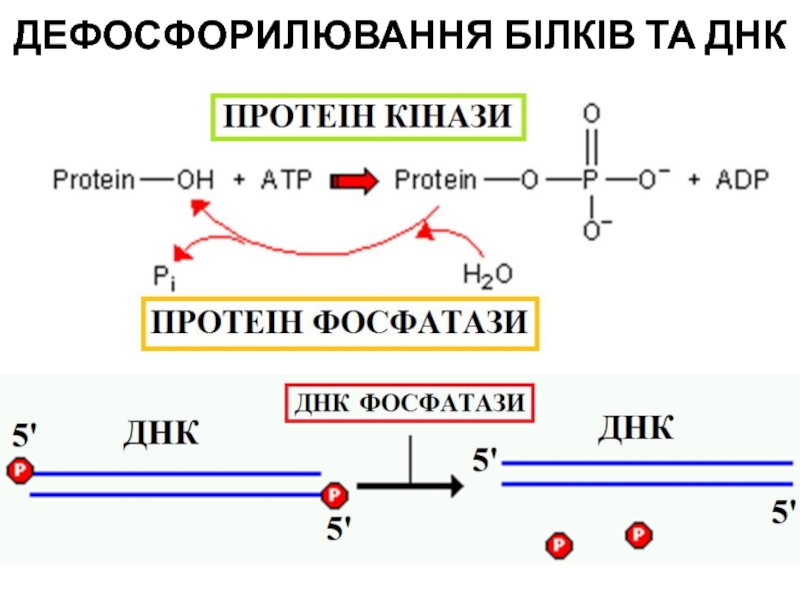
- 74. ДЕФОСФОРИЛЮВАННЯ БІЛКІВ ТА ДНК
- 75. ЛУЖНІ ФОСФАТАЗИ В МОЛЕКУЛЯРНІЙ БІОТЕХНОЛОГІЇЛужні фосфатази каталізують
- 76. ЛУЖНІ ФОСФАТАЗИ, ЯКІ НАЙЧАСТІШЕ ВИКОРИСТОВУЮТЬ В МОЛЕКУЛЯРНІЙ
- 77. ЛУЖНІ ФОСФАТАЗИ, ЯКІ НАЙЧАСТІШЕ ВИКОРИСТОВУЮТЬ В МОЛЕКУЛЯРНІЙ
- 78. НАПРЯМКИ ВИКОРИСТАННЯ ЛУЖНИХ ФОСФАТАЗЄ ДВА ОСНОВНИХ НАПРЯМКИ
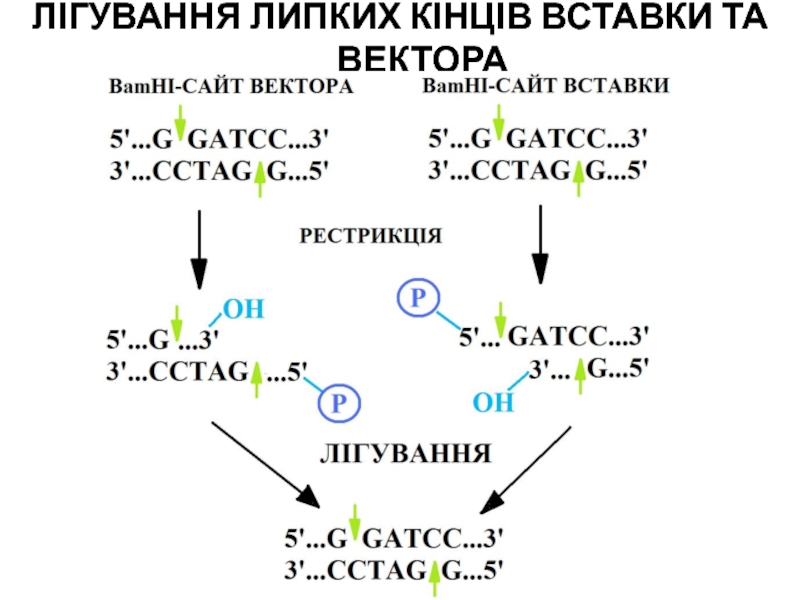
- 79. ЛІГУВАННЯ ЛИПКИХ КІНЦІВ ВСТАВКИ ТА ВЕКТОРА
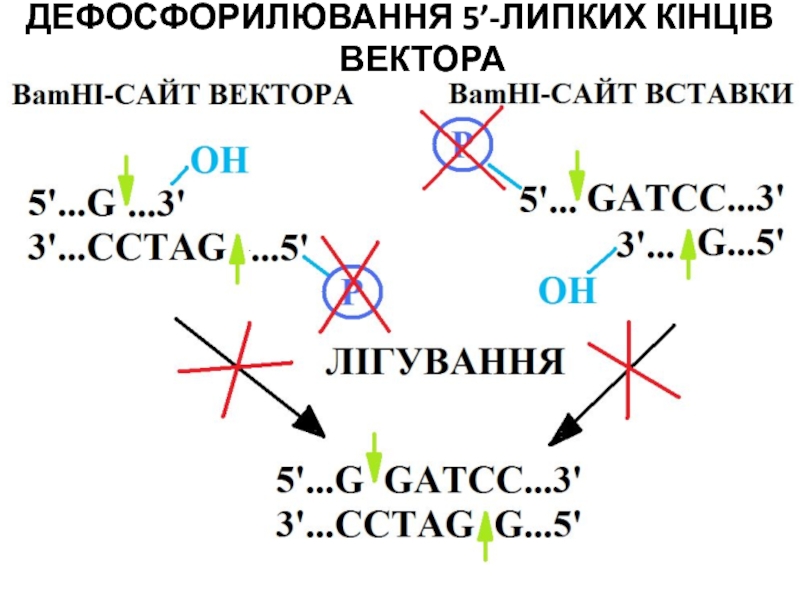
- 80. ДЕФОСФОРИЛЮВАННЯ 5’-ЛИПКИХ КІНЦІВ ВЕКТОРА
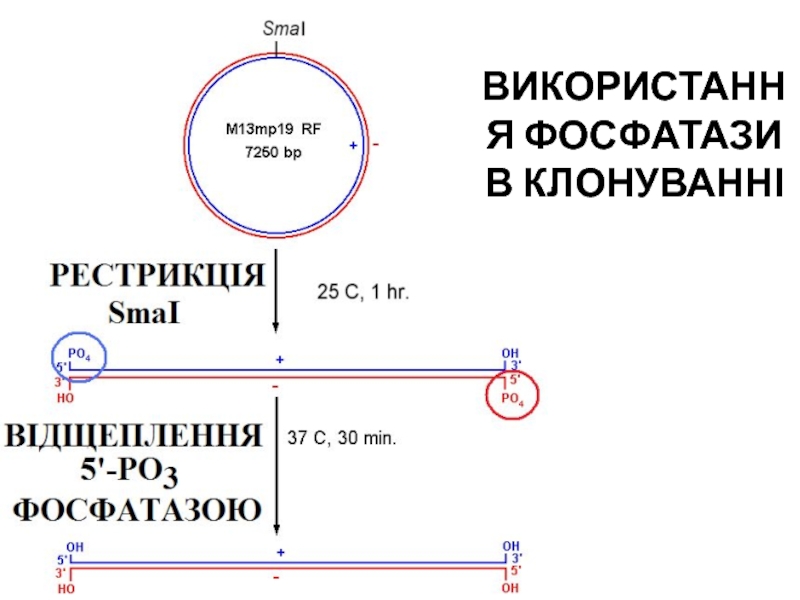
- 81. ВИКОРИСТАННЯ ФОСФАТАЗИ В КЛОНУВАННІ
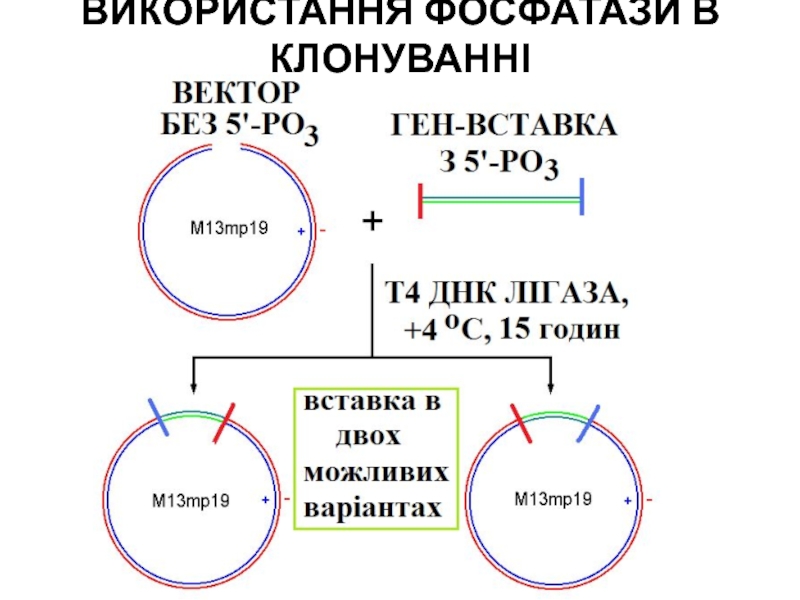
- 82. ВИКОРИСТАННЯ ФОСФАТАЗИ В КЛОНУВАННІ
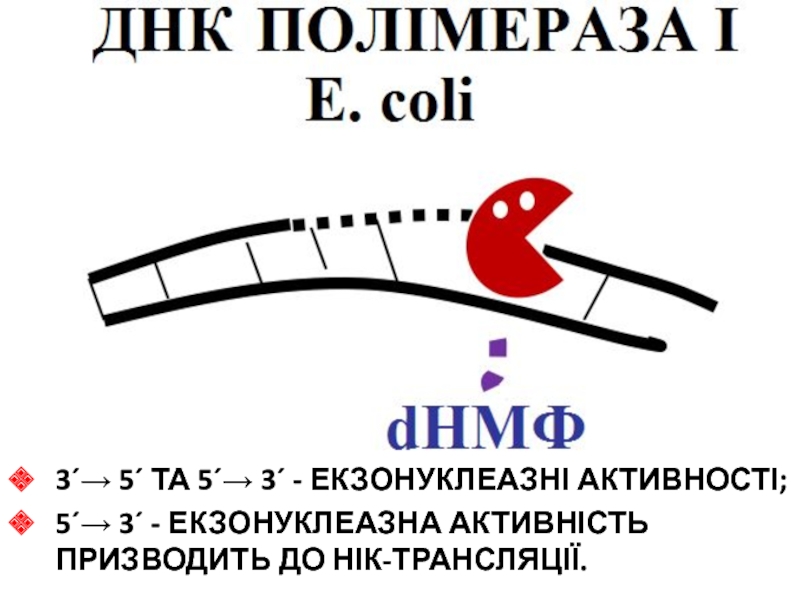
- 83. 3´→ 5´ ТА 5´→ 3´ - ЕКЗОНУКЛЕАЗНІ АКТИВНОСТІ;5´→ 3´ - ЕКЗОНУКЛЕАЗНА АКТИВНІСТЬ ПРИЗВОДИТЬ ДО НІК-ТРАНСЛЯЦІЇ.
- 84. ДНК-ПОЛІМЕРАЗА І E. coliФермент складається з одного
- 85. ДНК-ПОЛІМЕРАЗА І E. coli5’-3’-ЕКЗОНУКЛЕАЗНА АКТИВНІСТЬ – відщеплення
- 86. ДНК-ПОЛІМЕРАЗА І E. coli3’-5’-ЕКЗОНУКЛЕАЗНА АКТИВНІСТЬ – відщеплення
- 87. ДНК-ПОЛІМЕРАЗА І E. coliНІК-ТРАНСЛЯЦІЯ – переміщення ніка
- 88. НА СЬОГОДНІШНІЙ ДЕНЬ МАЙЖЕ НЕ ВИКОРИСТОВУЄТЬСЯ В МОЛЕКУЛЯРНІЙ БІОТЕХНОЛОГІЇ, АЛЕ …..
- 89. На відміну від ДНК-полімерази І – НЕ ЗДІЙСНЮЄ НІК-ТРАНСЛЯЦІЮ
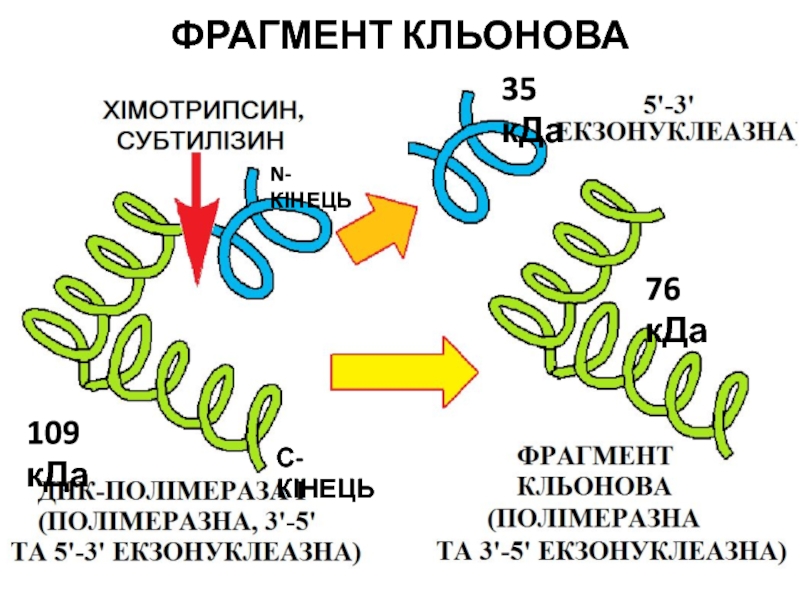
- 90. ФРАГМЕНТ КЛЬОНОВА109 кДа76 кДа35 кДаС-КІНЕЦЬN-КІНЕЦЬ
- 91. ФРАГМЕНТ КЛЬОНОВАВикористовується для ,,заповнення” виступаючих одноланцюгових 5’-кінців, які, наприклад, утворюються при розрізанні ДНК рестриктазами:
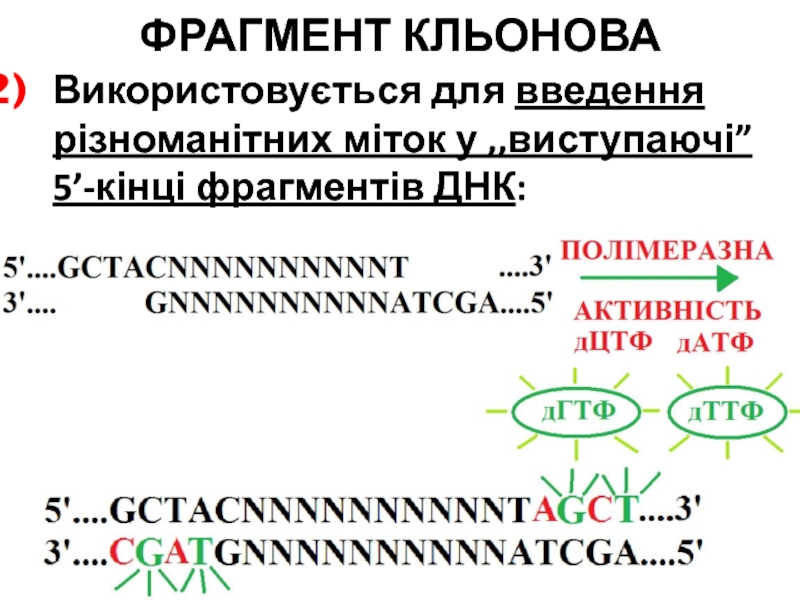
- 92. ФРАГМЕНТ КЛЬОНОВАВикористовується для введення різноманітних міток у ,,виступаючі” 5’-кінці фрагментів ДНК:
- 93. ФРАГМЕНТ КЛЬОНОВАВикористовується для синтезу другого комплементарного ланцюга
- 94. Відсутні екзонуклеазні активності
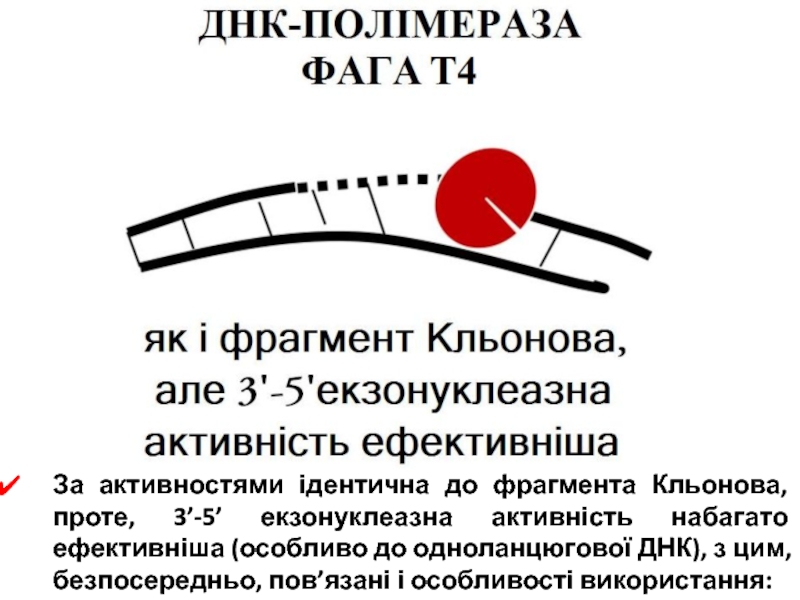
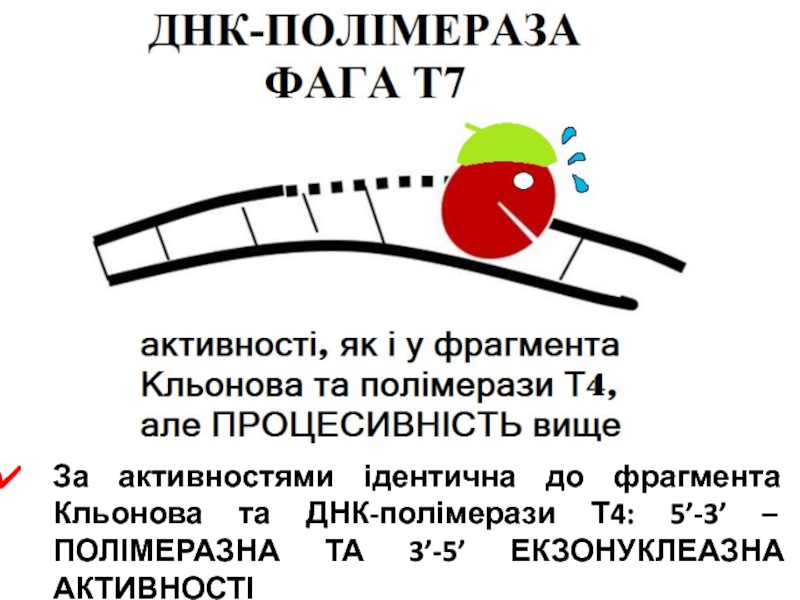
- 95. За активностями ідентична до фрагмента Кльонова, проте,
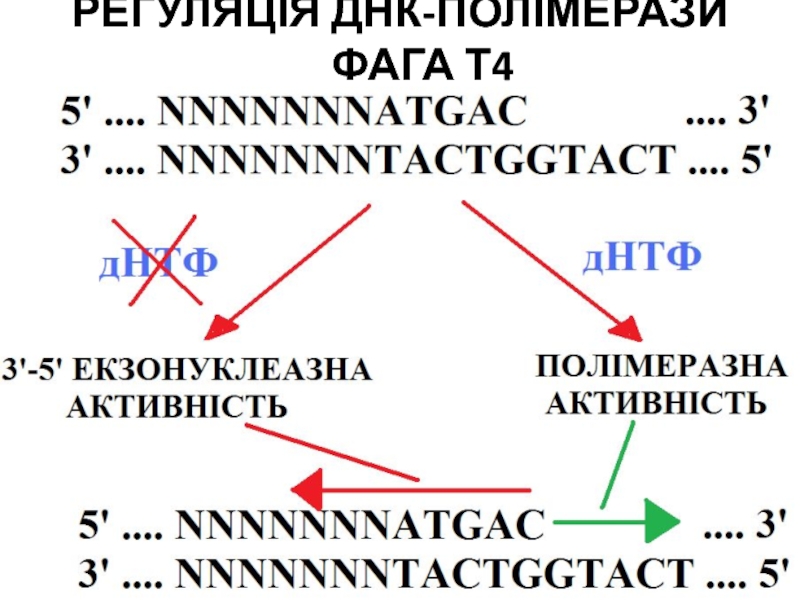
- 96. РЕГУЛЯЦІЯ ДНК-ПОЛІМЕРАЗИ ФАГА Т4
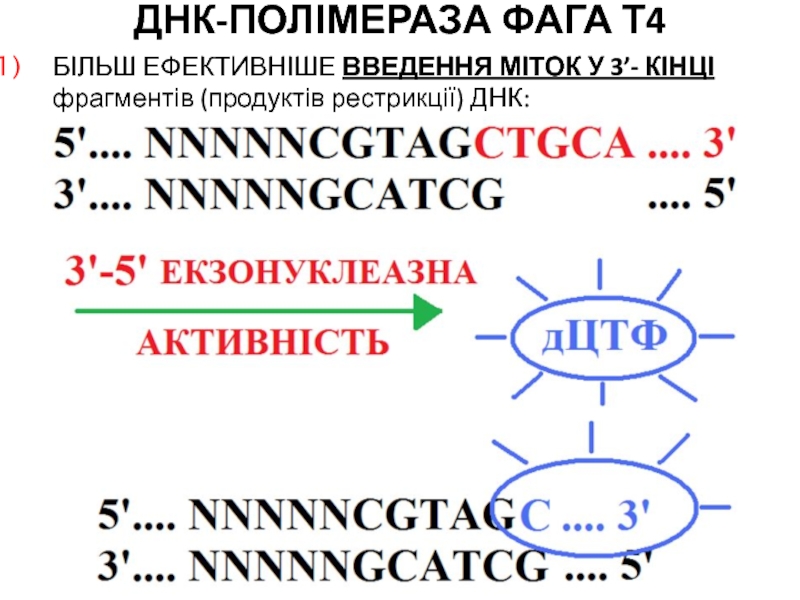
- 97. ДНК-ПОЛІМЕРАЗА ФАГА Т4БІЛЬШ ЕФЕКТИВНІШЕ ВВЕДЕННЯ МІТОК У 3’- КІНЦІ фрагментів (продуктів рестрикції) ДНК:
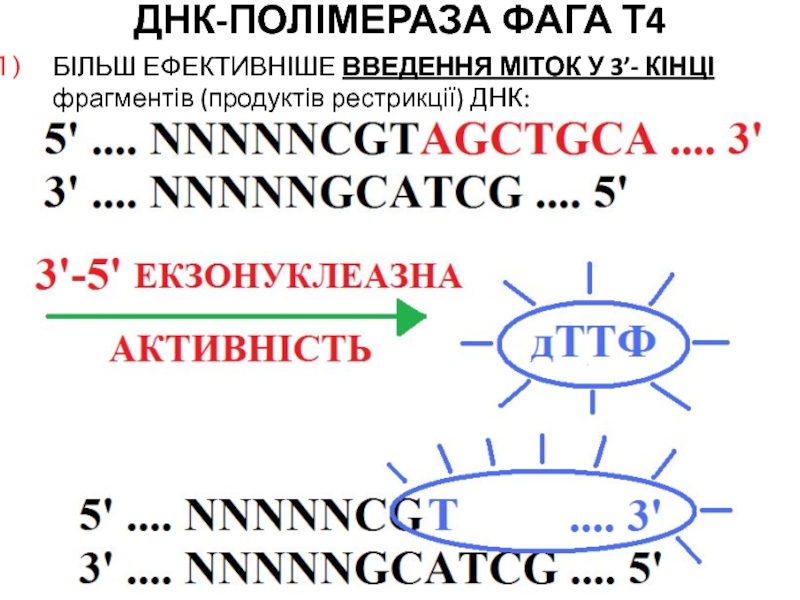
- 98. ДНК-ПОЛІМЕРАЗА ФАГА Т4БІЛЬШ ЕФЕКТИВНІШЕ ВВЕДЕННЯ МІТОК У 3’- КІНЦІ фрагментів (продуктів рестрикції) ДНК:
- 99. ДНК-ПОЛІМЕРАЗА ФАГА Т4ВВЕДЕННЯ МІТОК У ВИСТУПАЮЧІ 5’- КІНЦІ фрагментів (продуктів рестрикції) ДНК:
- 100. ДНК-ПОЛІМЕРАЗА ФАГА Т4ВВЕДЕННЯ МІТОК В ОБШИРНІ ОБЛАСТІ ДВОХЛАНЦЮГОВИХ ДНК використовуючи по-черзі екзонуклеазну і полімеразну активності
- 101. За активностями ідентична до фрагмента Кльонова та ДНК-полімерази Т4: 5’-3’ – ПОЛІМЕРАЗНА ТА 3’-5’ ЕКЗОНУКЛЕАЗНА АКТИВНОСТІ
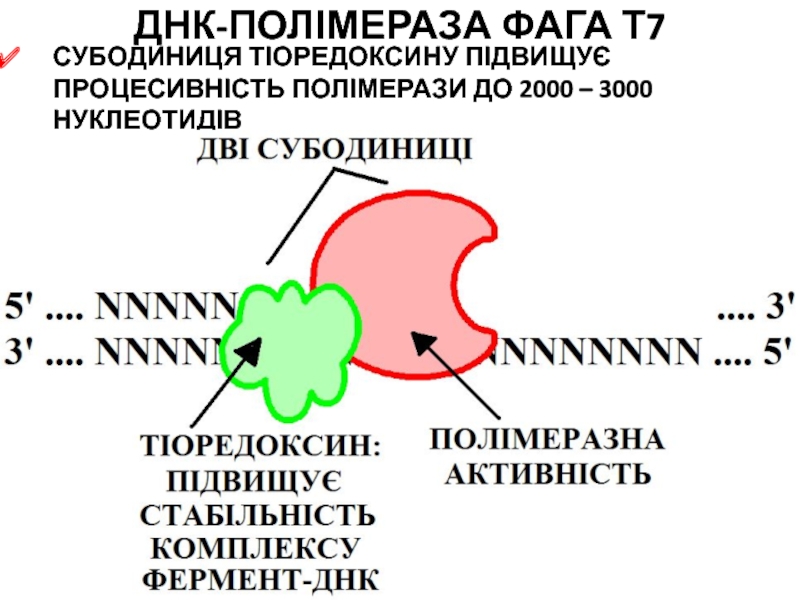
- 102. ДНК-ПОЛІМЕРАЗА ФАГА Т7СУБОДИНИЦЯ ТІОРЕДОКСИНУ ПІДВИЩУЄ ПРОЦЕСИВНІСТЬ ПОЛІМЕРАЗИ ДО 2000 – 3000 НУКЛЕОТИДІВ
- 103. ДНК-ПОЛІМЕРАЗА ФАГА Т7Завдяки високій процесивності фермент використовується
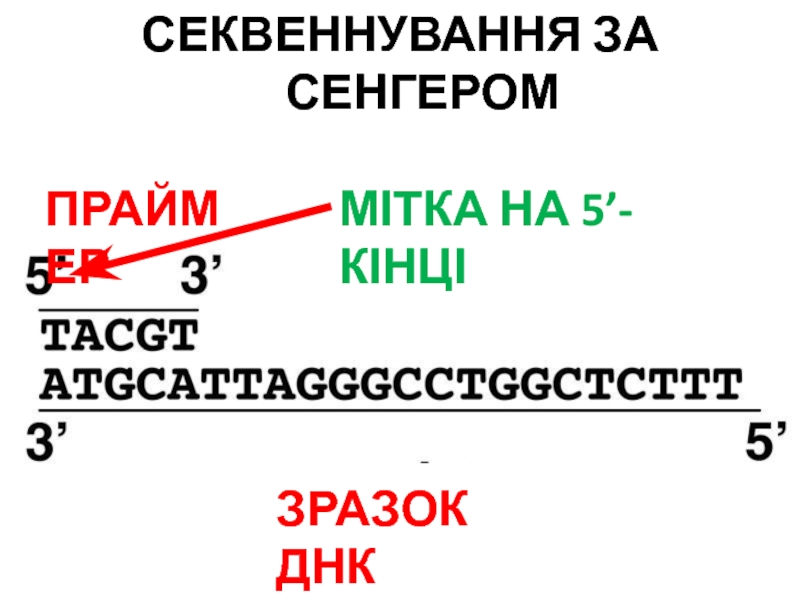
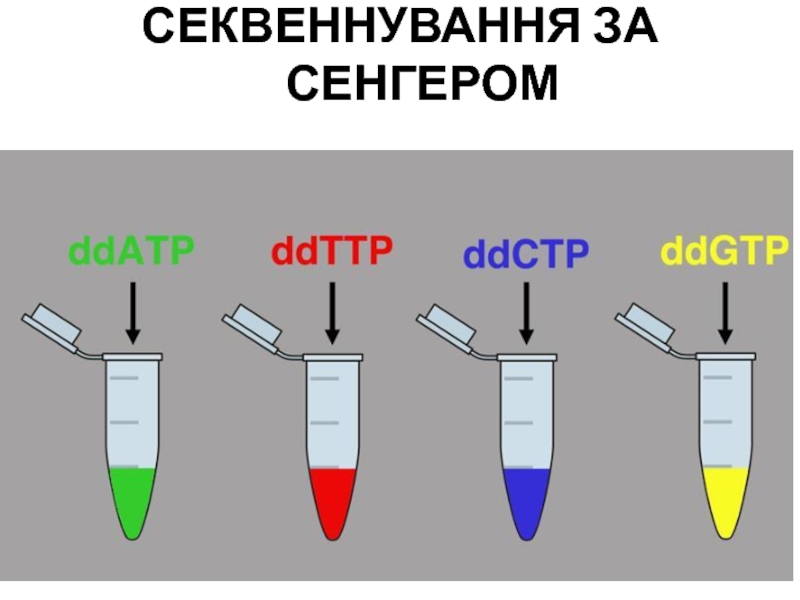
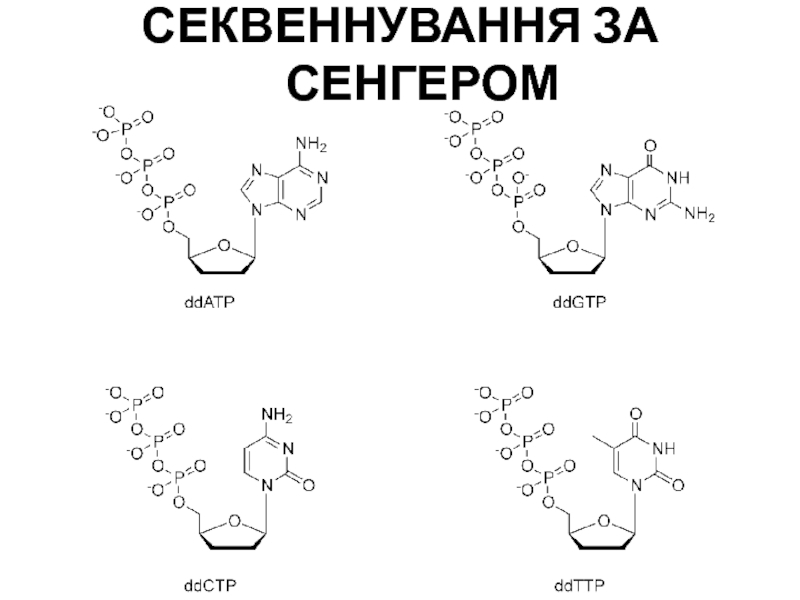
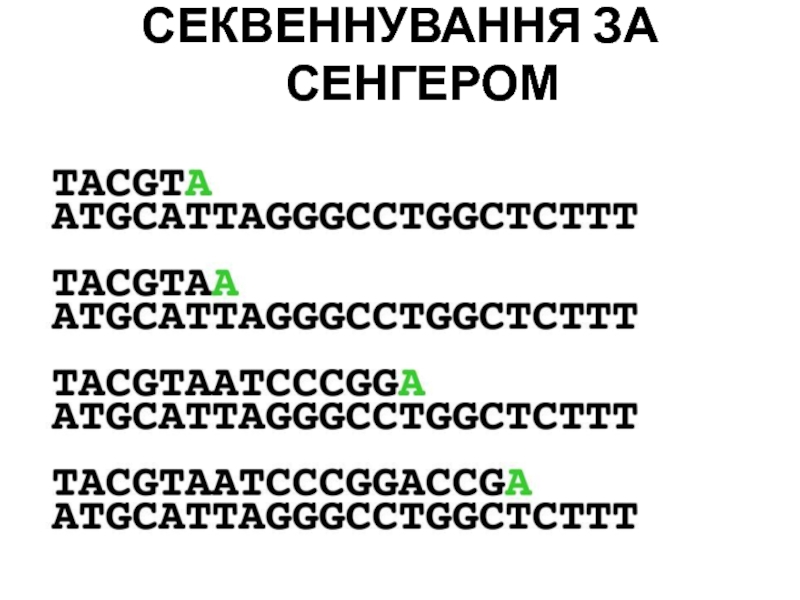
- 104. СЕКВЕННУВАННЯ ЗА СЕНГЕРОМ ПРАЙМЕРЗРАЗОК ДНКМІТКА НА 5’-КІНЦІ
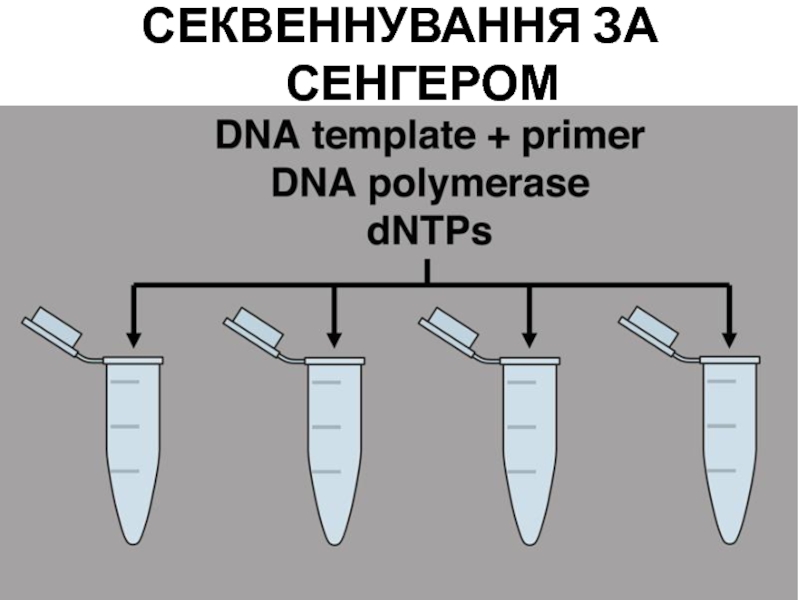
- 105. СЕКВЕННУВАННЯ ЗА СЕНГЕРОМ
- 106. СЕКВЕННУВАННЯ ЗА СЕНГЕРОМ
- 107. СЕКВЕННУВАННЯ ЗА СЕНГЕРОМ
- 108. СЕКВЕННУВАННЯ ЗА СЕНГЕРОМ
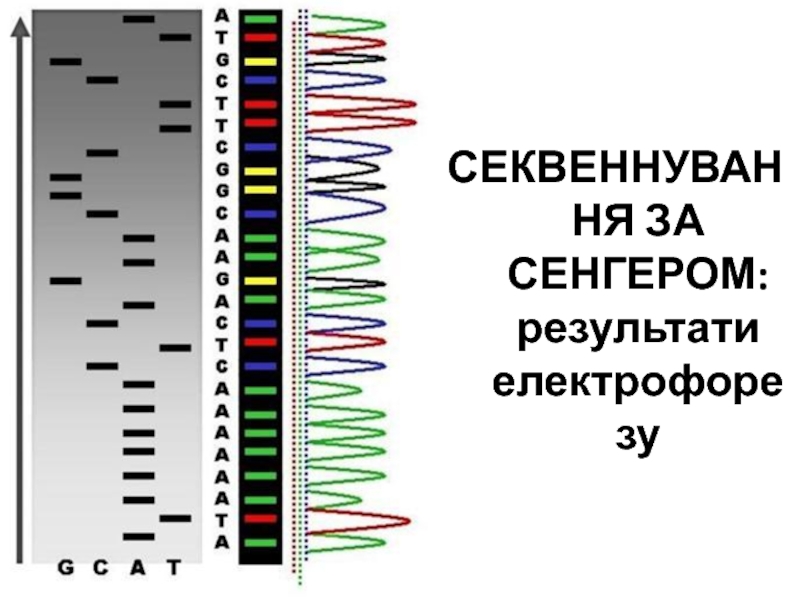
- 109. СЕКВЕННУВАННЯ ЗА СЕНГЕРОМ: результати електрофорезу
- 110. ТЕРМОСТАБІЛЬНІ ДНК-ПОЛІМЕРАЗИ ПЛР
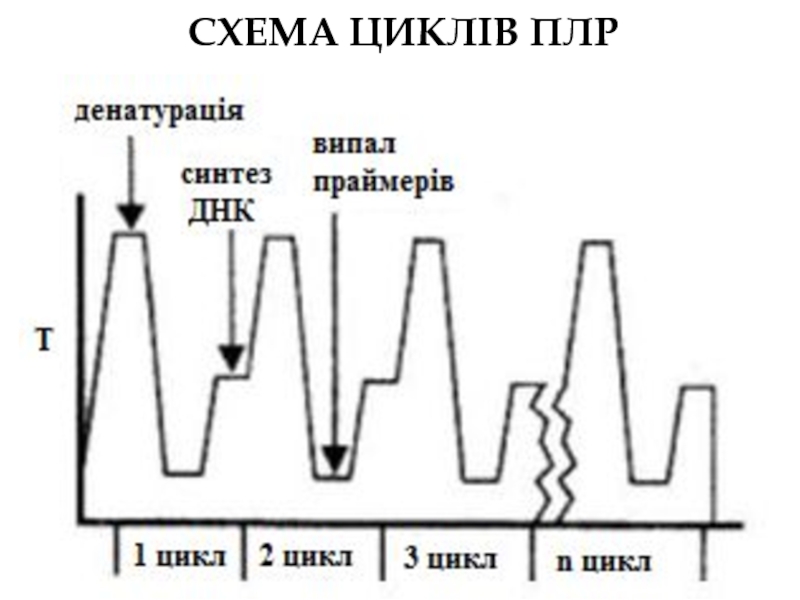
- 111. СХЕМА ЦИКЛІВ ПЛР
- 112. ТЕРМОСТАБІЛЬНІ ДНК-ПОЛІМЕРАЗИ 0 Taq Tth Pfu Vent,
- 113. ЗВОРОТНЯ ТРАНСКРИПТАЗА
- 114. 1975 рік – Нобелівська премія - за
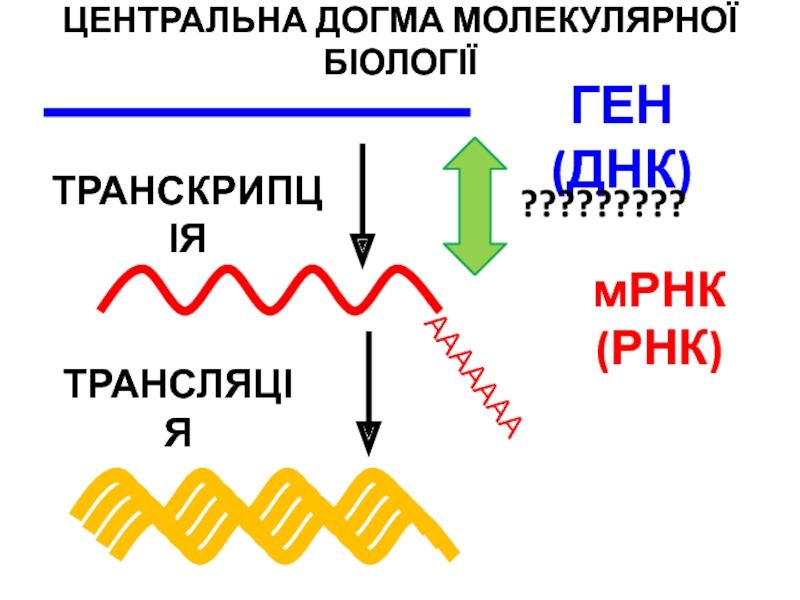
- 115. ЦЕНТРАЛЬНА ДОГМА МОЛЕКУЛЯРНОЇ БІОЛОГІЇмРНК (РНК)ГЕН (ДНК)ТРАНСЛЯЦІЯТРАНСКРИПЦІЯ?????????
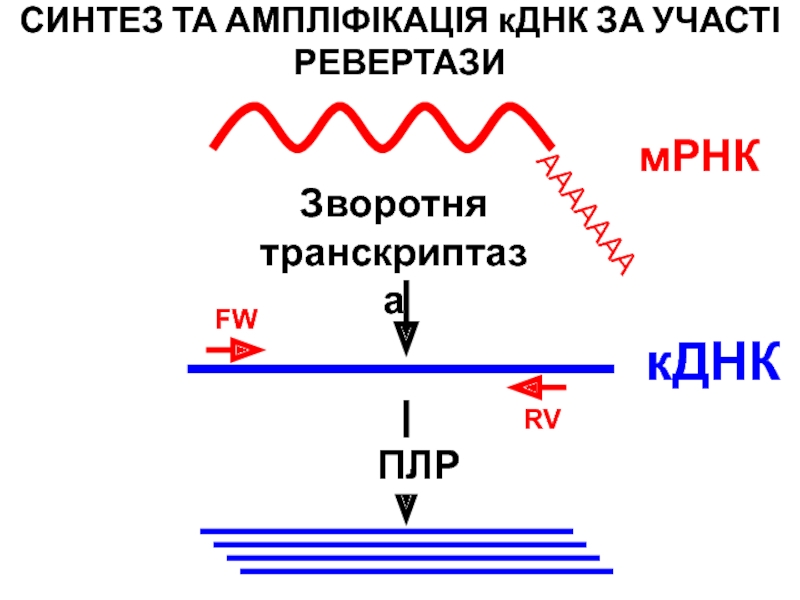
- 116. СИНТЕЗ ТА АМПЛІФІКАЦІЯ кДНК ЗА УЧАСТІ РЕВЕРТАЗИмРНКкДНКFWRVПЛРЗворотня транскриптаза
- 117. ЗАГАЛЬНА СХЕМА КЛОНУВАННЯ З ВИКОРИСТАННЯМ РЕСТРИКТАЗ, ЛІГАЗ ТА ФОСФАТАЗ
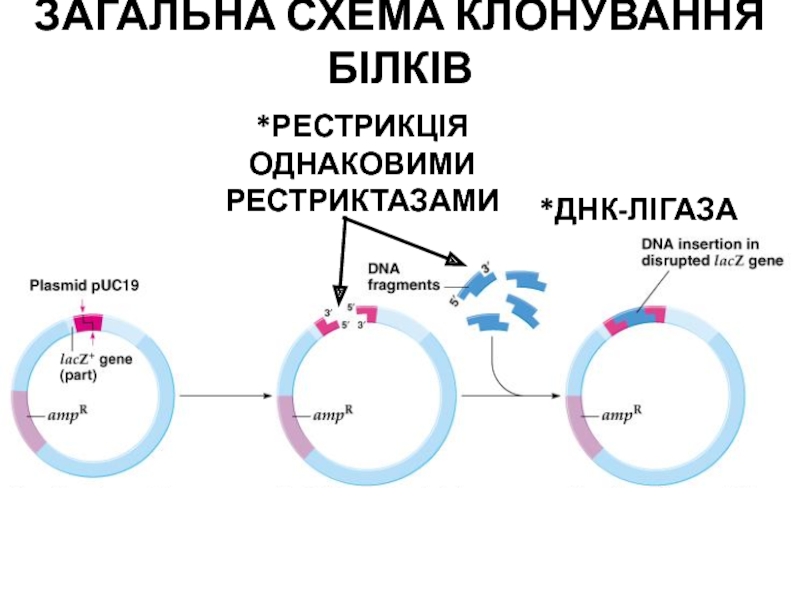
- 118. *РЕСТРИКЦІЯ ОДНАКОВИМИ РЕСТРИКТАЗАМИ*ДНК-ЛІГАЗАЗАГАЛЬНА СХЕМА КЛОНУВАННЯ БІЛКІВ
- 119. Скачать презентанцию
Слайды и текст этой презентации
Слайд 1МОЛЕКУЛЯРНА БІОТЕХНОЛОГІЯ.
ЛЕКЦІЯ №1.
ВСТУП. ІНСТРУМЕНТИ МОЛЕКУЛЯРНОЇ БІОТЕХНОЛОГІЇ: ФЕРМЕНТИ ТА ВЕКТОРИ
АСИСТЕНТ КАФЕДРИ
БІОХІМІЇ, К.Б.Н. МАНДРИК С.Я.
Слайд 2ПЛАН ЛЕКЦІЇ
Рестриктази – загальна характеристика. Фактори, які впливають на роботу
рестриктаз. Побудова рестрикційних карт послідовностей ДНК.
Лігази.
Фосфатази.
ДНК-полімераза І з клітин E.
coli.Фрагмент Кльонова ДНК-полімерази І E. coli.
Слайд 3ПЛАН ЛЕКЦІЇ
Фрагмент Кльонова (Exo-).
ДНК-полімерази фагів Т4 та Т7.
Термостабільні ДНК-полімерази в
ПЛР.
Зворотня транскриптаза.
Загальна схема клонування білків з використанням окремих ферментів.
Слайд 5ВІДКРИТТЯ РЕСТРИКТАЗ
HindII – перша рестриктаза, яка була випадково відкрита в
1970 році в дослідженнях процесів потрапляння ДНК вірусів в клітини
бактерій Haemophilus influenzae;HindII розпізнає та гідролізує ДНК в сайтах:
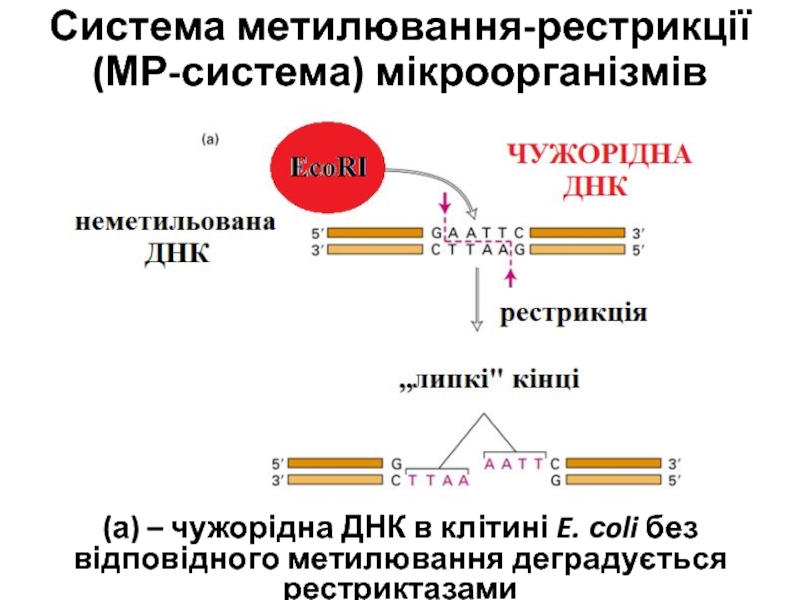
Слайд 6Система метилювання-рестрикції (МР-система) мікроорганізмів
(а) – чужорідна ДНК в клітині
E. сoli без відповідного метилювання деградується рестриктазами
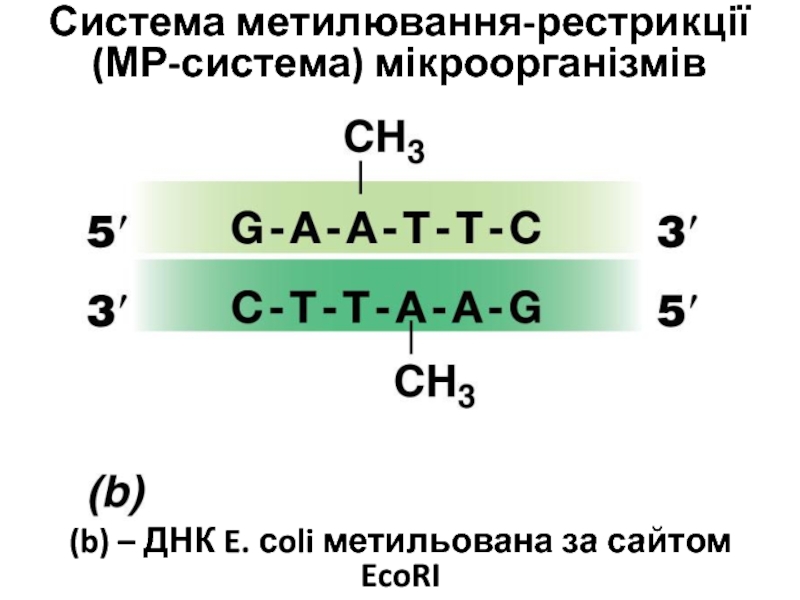
Слайд 7Система метилювання-рестрикції (МР-система) мікроорганізмів
(b) – ДНК E. сoli метильована
за сайтом EcoRI
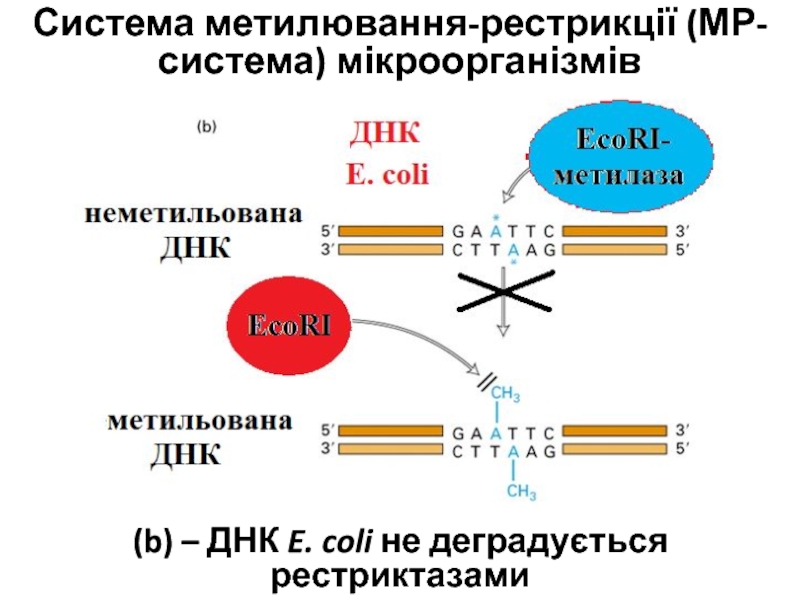
Слайд 8Система метилювання-рестрикції (МР-система) мікроорганізмів
(b) – ДНК E. coli не
деградується рестриктазами
Слайд 9Система метилювання-рестрикції (МР)
Захищає прокаріотичну клітину від чужорідної ДНК (бактеріофаги);
Поширена
в прокаріот;
Складається з метилази і відповідної рестриктази;
Кожен ВИД І ШТАМ
прокаріот містить свою унікальну систему МР;Фаги, які розмножувались в клітинах одного штаму бактерій обмежені в здатності інфікувати інші штами.
Є незамінною і широко використовується в in vitro маніпуляціях з ДНК;
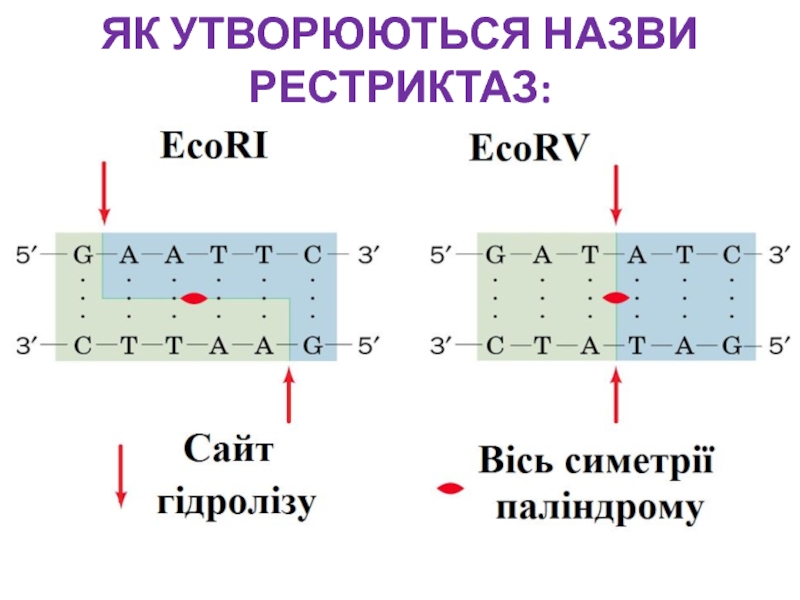
Слайд 10ЯК УТВОРЮЮТЬСЯ НАЗВИ РЕСТРИКТАЗ:
Назва виду бактерій, в клітинах яких було
виявлено рестриктазу;
В якому порядку з клітин даного виду рестриктаза була
ідентифікована, очищена та охарактеризована.ПРИКЛАД: EcoRI
R – ШТАМ E.coli
I – ПЕРША РЕСТРИКТАЗА, ЯКУ БУЛО ІДЕНТИФІКОВАНО І ОХАРАКТЕРИЗОВАНО В КЛІТИНАХ ДАНОГО ШТАМУ E.coli.
Слайд 13ПРИКЛАДИ СЛІВ-ПАЛІНДРОМІВ (ДАЛЕКО НЕ УСІ РЕСТРИКТАЗИ ГІДРОЛІЗУЮТЬ ДНК В ПАЛІНДРОМАХ)
Зараз
Анна
Пилип
зараЗ
аннА
пилиП
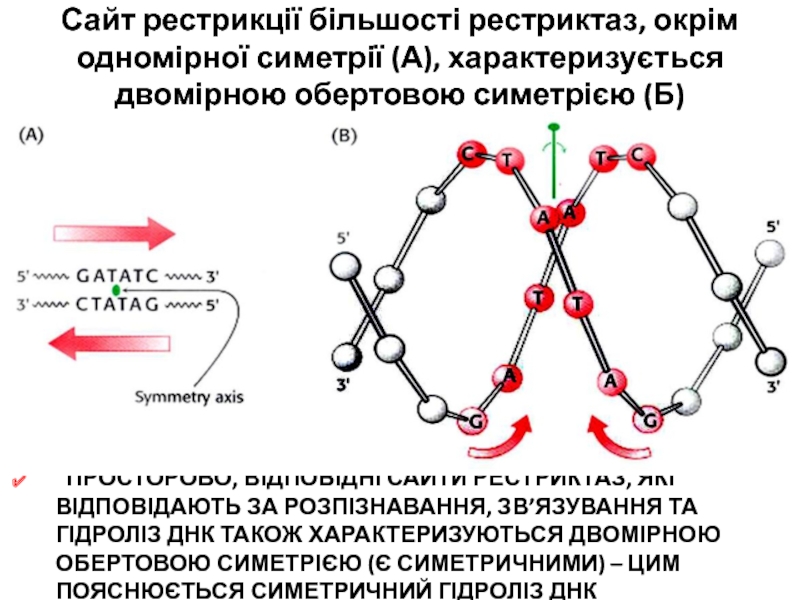
Слайд 16Сайт рестрикції більшості рестриктаз, окрім одномірної симетрії (А), характеризується двомірною
обертовою симетрією (Б)
ПРОСТОРОВО, ВІДПОВІДНІ САЙТИ РЕСТРИКТАЗ, ЯКІ ВІДПОВІДАЮТЬ ЗА
РОЗПІЗНАВАННЯ, ЗВ’ЯЗУВАННЯ ТА ГІДРОЛІЗ ДНК ТАКОЖ ХАРАКТЕРИЗУЮТЬСЯ ДВОМІРНОЮ ОБЕРТОВОЮ СИМЕТРІЄЮ (Є СИМЕТРИЧНИМИ) – ЦИМ ПОЯСНЮЄТЬСЯ СИМЕТРИЧНИЙ ГІДРОЛІЗ ДНКСлайд 19МЕХАНІЗМ ДІЇ РЕСТРИКТАЗ НА ДНК
,,Сканування” ДНК;
Розпізнавання певного сайту
на ДНК;
Внесення одинарного розриву на кожному дезоксирибозофосфатному ланцюзі;
Гідроліз ДНК
відбувається між 3’- атомом Оксигену та атомом Фосфору фосфодиефірного зв’язку.Слайд 20ГІДРОЛІЗ ДНК ЗА УЧАСТЮ РЕСТРИКТАЗ
Іони магнію необхідні для
утримання молекули води і, відповідно, задіяні в механізмі нуклеофільної атаки
Слайд 22Рестриктази I-го типу
Являють собою олігомерний білок, який складається з
декількох поліпептидних субодиниць;
Комплекс містить:
Дві R (restriction, рестрикція)
субодиниці;Дві M (methylation, метилювання) субодиниці;
Одну S (specificity, специфічність) субодиницю;
Сайт гідролізу ДНК міститься на довільній відстані від сайту розпізнавання;
Приклад – EcoK з E. coli: сайт розпізнавання несиметричний
0
5’ - AACNNNNNNGTGC - 3’
3’ – TTGNNNNNNCACG - 5’
Слайд 23Рестриктази IІІ-го типу
Являють собою олігомерний білок, який складається з
декількох поліпептидних субодиниць;
Сайт гідролізу ДНК міститься на ТОЧНО ДЕТЕРМІНОВАНІЙ
відстані від сайту розпізнавання;Приклад – EcoРІ з E. coli: сайт розпізнавання несиметричний
AGACC (27/25)
0
Слайд 24Рестриктази IV-го типу
Досліджені недостатньо;
Складаються з одного поліпептидного ланцюга, який
володіє і метилазною і рестриктазною активністю;
Сайт гідролізу ДНК міститься
на відстані до 30 нуклеотидів від сайту розпізнавання;Гідролізують тільки модифіковані (метильовані, гідроксиметильовані, глікозил-гідроксиметильовані) послідовності;
Приклад – Eco571 з E. coli: сайт розпізнавання несиметричний
CTGAAG (16/14)
0
Слайд 25Рестриктази IІ-го типу
Широко використовуються в бтх:
Розпізнають паліндромні (4-8 нуклеотидів) і
непаліндромні послідовності;
Сайт гідролізу розміщений в сайті розпізнавання і чітко детермінований
(або розміщений на чітко детермінованій відстані);Здебільшого потребують Mg 2+ в якості кофактора;
В РМ-системі метилаза і рестриктаза є незалежними окремими білками;
Не потребують АТФ;
При гідролізі ДНК на 5’-кінці залишається залишок фосфорної кислоти.
0
Слайд 26Рестриктази IІ-го типу підкласу ІІР
Більше половини усіх рестриктаз ІІ типу
відносяться до ПІДКЛАСУ ІІР: розпізнають тетра- гекса- і октануклеотидні паліндроми:
Або
- для HaeI i HaeII, відповідно, сайти, в яких можливі альтернативні симетричні варіанти:(де Pu – пуриновий нуклеотид, Py – піримідиновий, N –будь-який)
0
5’ - TСGА - 3’
3’ – AGCT - 5’
5’ - TGGСCА - 3’
3’ – ACCGGT - 5’
TaqI
BalI
5’ – (A/T)GGCC(А/T) - 3’
3’ – (T/A)CCGG(T/A) - 5’
5’ – PuGCGCPy - 3’
3’ – PyCGCGPu - 5’
Слайд 27Рестриктази IІ-го типу підкласів ІІW і IIN
ІІW - pозпізнають пента-
та гептануклеотидні (непарна к-ть) паліндроми з різними варіантами центрального і
фланкуючих нуклеотидів:ІІN – pозпізнають паліндроми, які можуть перериватись декількома довільними нуклеотидами: SfiI - сайти
(де Pu – пуриновий нуклеотид, Py – піримідиновий, N –будь-який)
0
5’ - GANTС - 3’
3’ – CTNAG - 5’
5’ - PuGGNСCPy - 3’
3’ – PyCCNGGPu - 5’
HinfI
DraI
5’ – GGCCN5GGCС - 3’
3’ – CCGGN5CCGG - 5’
SfiI
Слайд 28Рестриктази IІ-го типу підкласів ІІS і IIT
ІІS – pозпізнають НЕПАЛІНДРОМНІ
послідовності, які складаються з 4-6 і більшої кількості нуклеотидів і
гідролізують ДНК на чітко детермінованій відстані від сайту розпізнавання (1-20 нуклеотидів): MnlIІІT – pозпізнають НЕПАЛІНДРОМНІ послідовності, які складаються з 4-6 і більшої кількості нуклеотидів і гідролізують ДНК в цих сайтах: SgrAI
(де Pu – пуриновий нуклеотид, Py – піримідиновий, N –будь-який)
0
5’ – CCTCN7 – 3’
5’ – CAG CCGGTCG – 3’
MnlI
SgrAI
Слайд 29РЕСТРИКТАЗИ ІІ ТИПУ - УЗАГАЛЬНЕННЯ
Найбільш широко використовуються в клонуванні
і, загалом, аналізі геному;
Більш ніж 3500 представників;
Розпізнають 4-8
нуклеотидні послідовності (не усі з них паліндроми);Гідроліз ДНК на чітко детермінованій відстані від сайту розпізнавання;
Гомодимери.
0
Слайд 35ТИПИ RM-НЕЗАЛЕЖНОГО МЕТИЛЮВАННЯ, ЯКЕ МОЖЕ ВПЛИВАТИ НА ГІДРОЛІЗ ДНК ЗА
УЧАСТЮ РЕСТРИКТАЗ
0
Dam
Dcm
EcoK
EcoB
CpG
E. coli
еукаріоти
Слайд 390
Вплив Dam-метилювання на рестрикцію
Dam-метилювання НЕ ВПЛИВАЄ на рестрикцію
Dam-метилювання БЛОКУЄ рестрикцію
Слайд 400
Вплив Dcm-метилювання на рестрикцію
Dсm-метилювання БЛОКУЄ рестрикцію
Dсm-метилювання НЕ ВПЛИВАЄ рестрикцію
Слайд 42Ізошизомери і гетерошизомери
ІЗОМЕРИ – рестриктази різного походження, які розпізнають однакові
сайти на ДНК;
ІЗОШИЗОМЕРИ - рестриктази різного походження, які розпізнають однакові
сайти і гідролізують ДНК за однаковими положеннями;0
ІЗОШИЗОМЕРИ:
Bsp1431 (Sau3AI) – рестрикція НЕ БЛОКУЄТЬСЯ Dam-метилюванням
MboI – рестрикція БЛОКУЄТЬСЯ Dam-метилюванням
Слайд 43Ізошизомери і гетерошизомери
0
ГЕТЕРОШИЗОМЕРИ:
Acc651 (Asp718I) – Dсm-метилювання МОЖЕ ВПЛИВАТИ
НА РЕСТРИКЦІЮ; KpnI – Dсm-метилювання НЕ ВПЛИВАЄ НА РЕСТРИКЦІЮ;
ГЕТЕРОШИЗОМЕРИ (НЕОШИЗОМЕРИ) - рестриктази різного походження, які розпізнають однакові сайти, але гідролізують ДНК за різними положеннями;
Слайд 47ПОБУДОВА РЕСТРИКЦІЙНОЇ КАРТИ кДНК (мРНК) ІНСУЛІНУ ЛЮДИНИ
(NCBI Reference Sequence:
NM_000207.2)
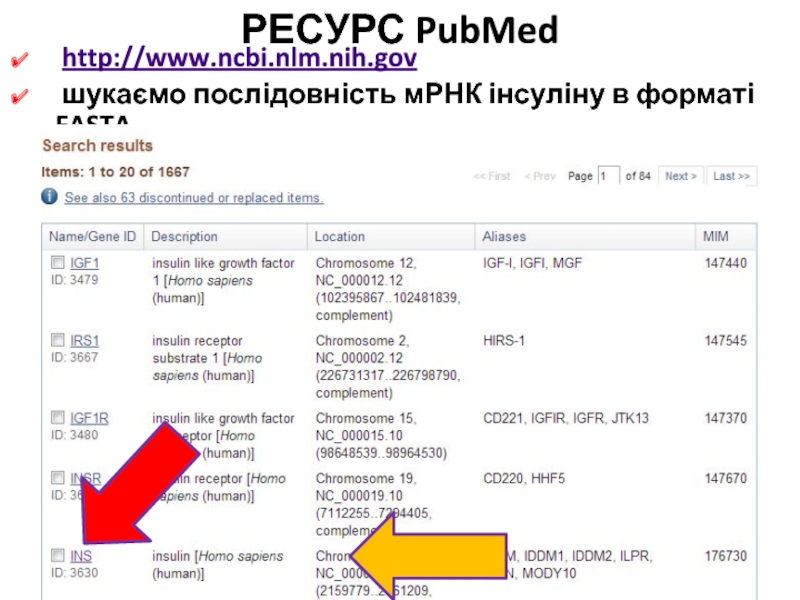
Слайд 48РЕСУРС PubMed
http://www.ncbi.nlm.nih.gov
шукаємо послідовність мРНК інсуліну в форматі
FASTA
0
Слайд 49РЕСУРС PubMed
http://www.ncbi.nlm.nih.gov
шукаємо послідовність мРНК інсуліну в форматі
FASTA
0
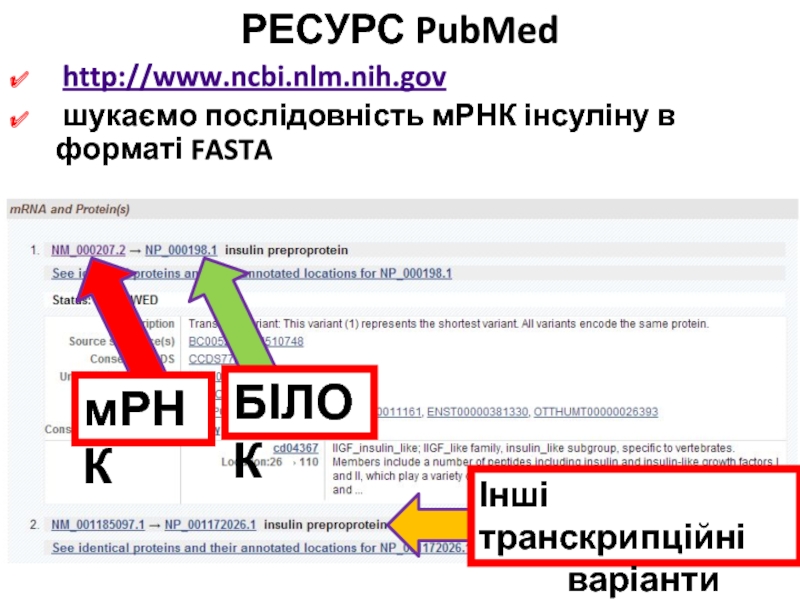
Слайд 50РЕСУРС PubMed
http://www.ncbi.nlm.nih.gov
шукаємо послідовність мРНК інсуліну в форматі
FASTA
0
мРНК
БІЛОК
Інші транскрипційні
варіанти
Слайд 51РЕСУРС PubMed
http://www.ncbi.nlm.nih.gov
шукаємо послідовність мРНК інсуліну в форматі
FASTA
0
ID ПОСЛІДОВНОСТІ
FASTA
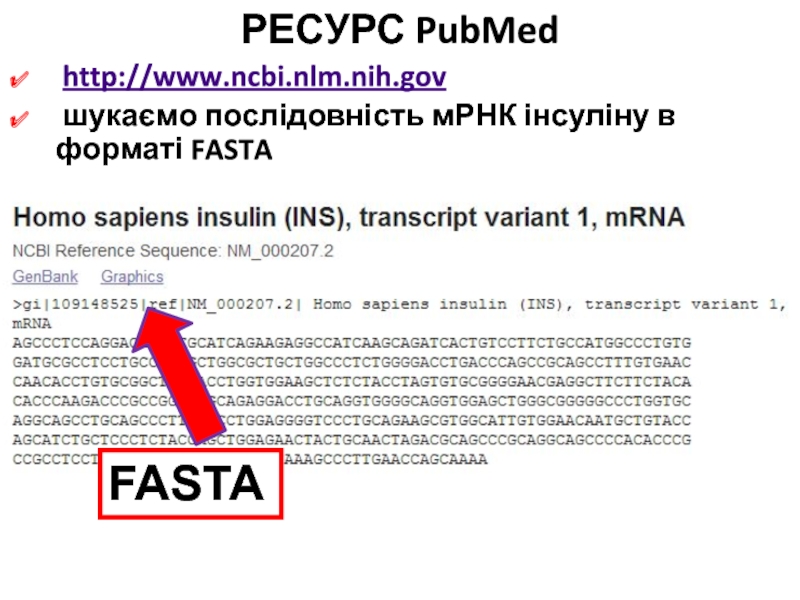
Слайд 52РЕСУРС PubMed
http://www.ncbi.nlm.nih.gov
шукаємо послідовність мРНК інсуліну в форматі
FASTA
0
FASTA
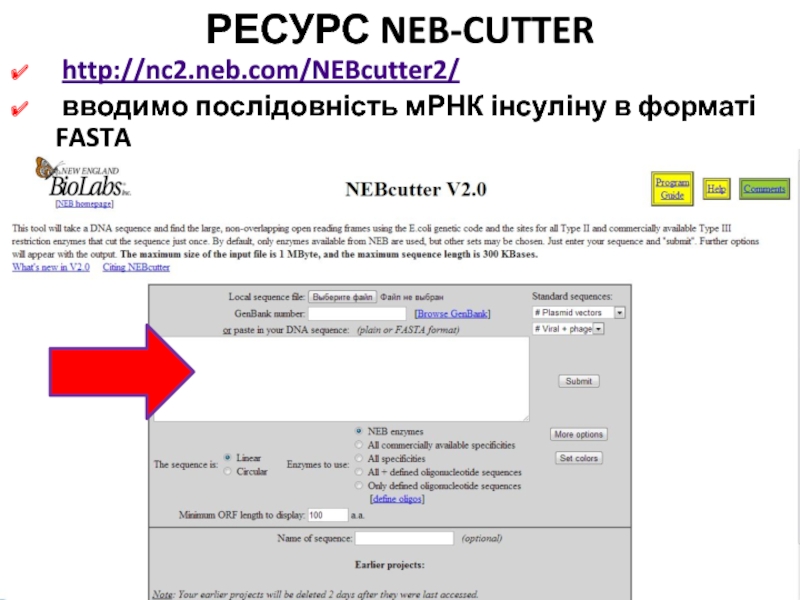
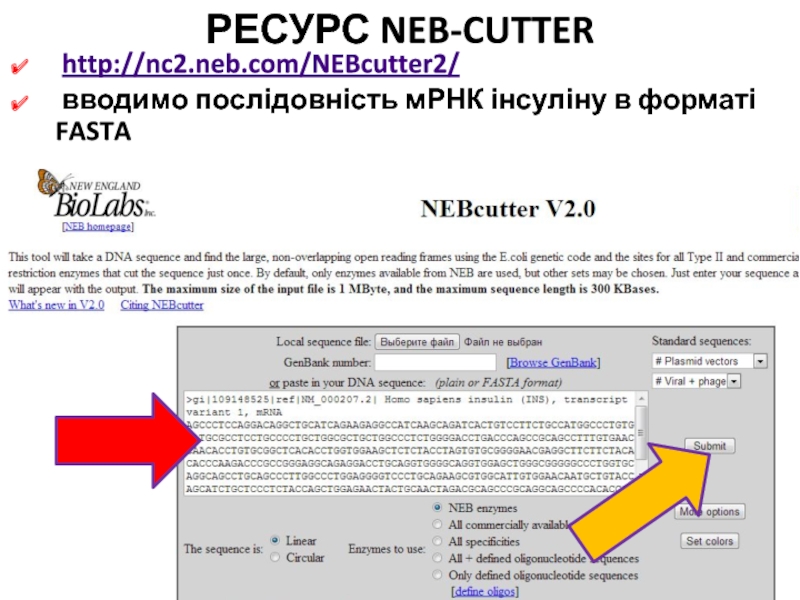
Слайд 53РЕСУРС NEB-CUTTER
http://nc2.neb.com/NEBcutter2/
вводимо послідовність мРНК інсуліну в форматі FASTA
0
Слайд 54РЕСУРС NEB-CUTTER
http://nc2.neb.com/NEBcutter2/
вводимо послідовність мРНК інсуліну в форматі FASTA
0
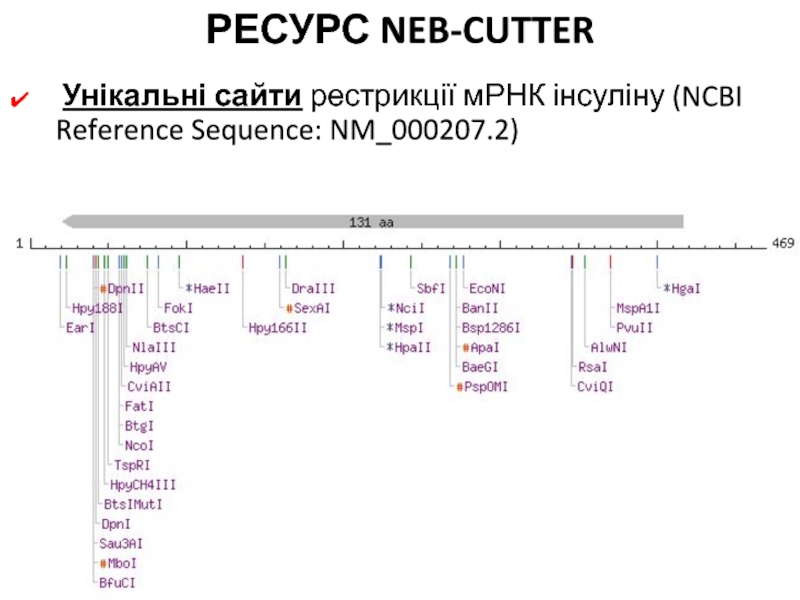
Слайд 55РЕСУРС NEB-CUTTER
Унікальні сайти рестрикції мРНК інсуліну (NCBI Reference Sequence:
NM_000207.2)
0
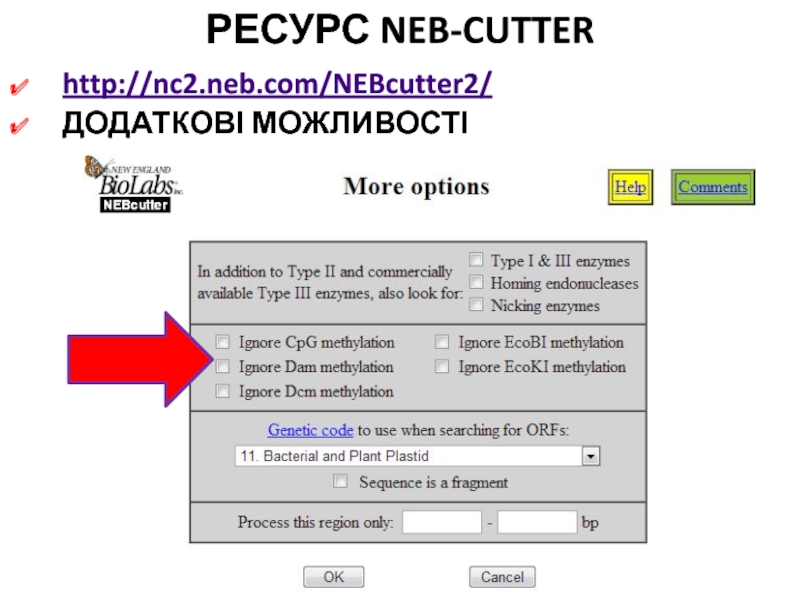
Слайд 56РЕСУРС NEB-CUTTER
Сайти рестрикції мРНК інсуліну (NCBI Reference Sequence: NM_000207.2), які
зустрічаються двічі
0
Слайд 57РЕСУРС NEB-CUTTER
Сайти рестрикції мРНК інсуліну (NCBI Reference Sequence: NM_000207.2), які
зустрічаються тричі
0
Слайд 60ЗАВДАННЯ ДЛЯ САМОСТІЙНОЇ РОБОТИ: ОТРИМАТИ РЕСТРИКЦІЙНІ КАРТИ кДНК (мРНК) ІНТЕРФЕРОНІВ,
АЛЬБУМІНУ ЛЮДИНИ ТА ДНК-ЗАЛЕЖНОЇ ДНК ПОЛІМЕРАЗИ І Escherichia coli
Слайд 63Ферментативні активності Т4-ДНК лігази
Каталізує утворення фосфодиефірного зв’язку між 5’‑фосфатом та
3’-гідроксильною групою:
Репарує одноланцюгові розриви (ніки) в дуплексах ДНК, РНК, чи
ДНК/РНК-гібридах;“Зшиває” фрагменти ДНК з липкими чи тупими кінцями;
Для каталізу T4-ДНК лігазі необхідний АТФ в якості кофактора.
Слайд 68ВИКОРИСТАННЯ ЛІГАЗ В МОЛЕКУЛЯРНІЙ БІОТЕХНОЛОГІЇ
Клонування фрагментів (зокрема, ПЛР) ДНК після
рестрикції;
Приєднання двохланцюгових ДНК-лінкерів/адапторів до цільової ДНК;
Сайт-спрямований мутагенез (Site-directed mutagenesis);
Поліморфізм довжин
ампліфікованих фрагментів (Amplified fragment length polymorphism (AFLP));Лігазна ПЛР (Ligase-mediated RNA detection);
Репарація ніків в дуплексах ДНК, РНК чи ДНК/РНК-гібридах;
Слайд 69Т4 РНК-ЛІГАЗА
Т4 РНК лігаза каталізує утворення фосфодиефірних зв’язків між 3’-ОН
– групою та 5’-залишком фосфорної кислоти в:
Олігонуклеотидах;
Одноланцюгових молекулах РНК та
ДНК;Реакція є АТФ-залежною.
Слайд 71ВИКОРИСТАННЯ Т4 РНК-ЛІГАЗИ
Внесення міток на 3’-кінці РНК (зокрема, різних похідних
рСр (цитидин 3’,5’‑ біс – фосфат));
,,Зшивання” одноланцюгових молекул РНК;
Специфічні модифікації
тРНК;Лігування олігодезоксинуклеотидів з одноланцюговими кДНК для 5’ RACE (Rapid Amplification of cDNA Ends).
Слайд 72Внесення в якості міток різних похідних рСр на 3’-кінці мікроРНК.
Мічені міРНК використовуються в мікроареях
Слайд 75ЛУЖНІ ФОСФАТАЗИ В МОЛЕКУЛЯРНІЙ БІОТЕХНОЛОГІЇ
Лужні фосфатази каталізують відщеплення 5‘-фосфатного залишку
від ДНК та РНК.
Ферменти також володіють здатністю каталізувати відщеплення фосфатних
залишків в нуклеотидах та білках. Найвищу активність проявляють в лужному діапазоні pH.
Слайд 76ЛУЖНІ ФОСФАТАЗИ, ЯКІ НАЙЧАСТІШЕ ВИКОРИСТОВУЮТЬ В МОЛЕКУЛЯРНІЙ БІОТЕХНОЛОГІЇ
Три ізоформи фермента
найчастіше використовують в маніпуляціях з ДНК та РНК. Основною відмінністю
цих ферментів є здатність інактивуватись при певній температурі:Бактеріальна (E. coli) лужна фосфатаза (Bacterial alkaline phosphatase (BAP)) - найбільш активний фермент – як наслідок – рідко використовуюється на практиці, оскільки навіть при високих температурах інактивується неповністю і реакцію дефосфорилювання не вдається зупинити.
Слайд 77ЛУЖНІ ФОСФАТАЗИ, ЯКІ НАЙЧАСТІШЕ ВИКОРИСТОВУЮТЬ В МОЛЕКУЛЯРНІЙ БІОТЕХНОЛОГІЇ
Лужна фосфатаза кишківника
ВРХ (Calf intestinal alkaline phosphatase (CIP)). Найбільш широко використовується, оскільки
відносно легко інактивується протеазами або під впливом високих температур (75°C/10 хв у присутності 5 мМ ЕДTA). Активність нижча ніж у бактеріальної ізоформи.Лужна фосфатаза креветок (Shrimp alkaline phosphatase (SAP)). Також досить часто використовується. За кінетичними характеристиками схожа до ізоформи ВРХ. Отримана з антарктичних креветок. Інактивація за 65°C протягом 15 хв.
Слайд 78НАПРЯМКИ ВИКОРИСТАННЯ ЛУЖНИХ ФОСФАТАЗ
Є ДВА ОСНОВНИХ НАПРЯМКИ ВИКОРИСТАННЯ ЦИХ ФЕРМЕНТІВ
В МАНІПУЛЯЦІЯХ З ДНК:
Відщеплення 5‘-залишку фосфату фагових та плазмідних векторів
після їх рестрикції. Таке дефосфорилювання попереджує самолігування векторів і, як наслідок, полегшує лігування ДНК цільових генів з дефосфорильованими векторами.Відщеплення 5‘-залишку фосфату перед внесенням міченого фосфату у послідовності ДНК. Полінуклеотид кіназа ефективніше фосфорилює ДНК у випадку якщо 5‘-фосфат був попередньо видалений.
Слайд 833´→ 5´ ТА 5´→ 3´ - ЕКЗОНУКЛЕАЗНІ АКТИВНОСТІ;
5´→ 3´ -
ЕКЗОНУКЛЕАЗНА АКТИВНІСТЬ ПРИЗВОДИТЬ ДО НІК-ТРАНСЛЯЦІЇ.
Слайд 84ДНК-ПОЛІМЕРАЗА І E. coli
Фермент складається з одного поліпептидного ланцюга (близько
109 кДа), який володіє 3-ма активностями:
Полімеразною – синтез ланцюга в
5’-3’ напрямку. До 3’-ОН-кінця ,,праймера” комплементарно приєднуються дНТФ. Процесивність (здатність приєднувати певну кількість залишків нуклеотидів за один акт посадки на ДНК-матрицю) невисока – в середньому 10-50 нуклеотидів. Слайд 85ДНК-ПОЛІМЕРАЗА І E. coli
5’-3’-ЕКЗОНУКЛЕАЗНА АКТИВНІСТЬ – відщеплення нуклеотидів з 5’-кінця
двохланцюгових молекул ДНК, що може призвести до їх деградації.
Слайд 86ДНК-ПОЛІМЕРАЗА І E. coli
3’-5’-ЕКЗОНУКЛЕАЗНА АКТИВНІСТЬ – відщеплення нуклеотидів з 3’-кінця
одно- та двохланцюгових молекул ДНК, що може призвести до їх
деградації. На двохланцюговій ДНК екзонуклеазна активність інгібується полімеразноюСлайд 87ДНК-ПОЛІМЕРАЗА І E. coli
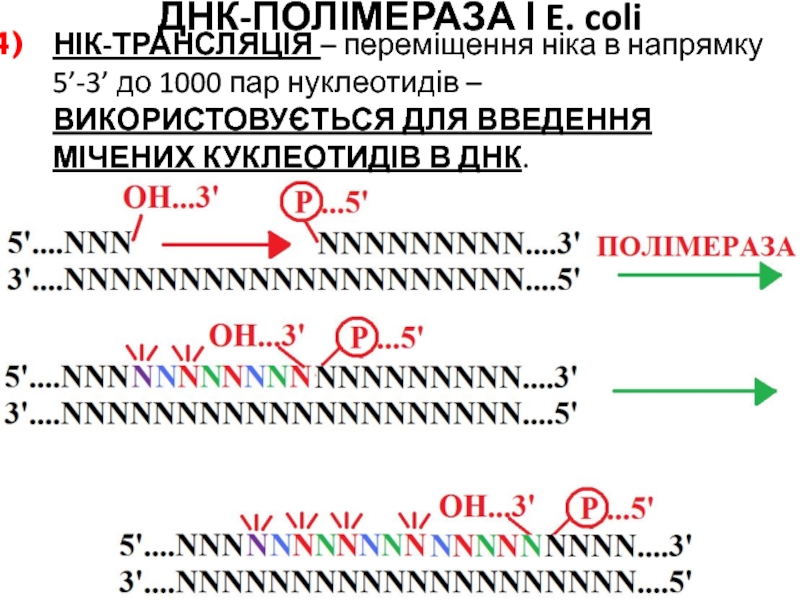
НІК-ТРАНСЛЯЦІЯ – переміщення ніка в напрямку 5’-3’
до 1000 пар нуклеотидів – ВИКОРИСТОВУЄТЬСЯ ДЛЯ ВВЕДЕННЯ МІЧЕНИХ КУКЛЕОТИДІВ
В ДНК.Слайд 91ФРАГМЕНТ КЛЬОНОВА
Використовується для ,,заповнення” виступаючих одноланцюгових 5’-кінців, які, наприклад, утворюються
при розрізанні ДНК рестриктазами:
Слайд 92ФРАГМЕНТ КЛЬОНОВА
Використовується для введення різноманітних міток у ,,виступаючі” 5’-кінці фрагментів
ДНК:
Слайд 93ФРАГМЕНТ КЛЬОНОВА
Використовується для синтезу другого комплементарного ланцюга кДНК в ПЛР
зі зворотньою транскрибцією.
Для секвенування ДНК методом Сенгера.
Для синтезу
олігонуклеотидів.ШВИДКІСТЬ РОБОТИ – БЛИЗЬКО 30 НУКЛЕОТИДІВ ЗА СЕКУНДУ
Слайд 95За активностями ідентична до фрагмента Кльонова, проте, 3’-5’ екзонуклеазна активність
набагато ефективніша (особливо до одноланцюгової ДНК), з цим, безпосередньо, пов’язані
і особливості використання:Слайд 97ДНК-ПОЛІМЕРАЗА ФАГА Т4
БІЛЬШ ЕФЕКТИВНІШЕ ВВЕДЕННЯ МІТОК У 3’- КІНЦІ фрагментів
(продуктів рестрикції) ДНК:
Слайд 98ДНК-ПОЛІМЕРАЗА ФАГА Т4
БІЛЬШ ЕФЕКТИВНІШЕ ВВЕДЕННЯ МІТОК У 3’- КІНЦІ фрагментів
(продуктів рестрикції) ДНК:
Слайд 99ДНК-ПОЛІМЕРАЗА ФАГА Т4
ВВЕДЕННЯ МІТОК У ВИСТУПАЮЧІ 5’- КІНЦІ фрагментів (продуктів
рестрикції) ДНК:
Слайд 100ДНК-ПОЛІМЕРАЗА ФАГА Т4
ВВЕДЕННЯ МІТОК В ОБШИРНІ ОБЛАСТІ ДВОХЛАНЦЮГОВИХ ДНК використовуючи
по-черзі екзонуклеазну і полімеразну активності
Слайд 101За активностями ідентична до фрагмента Кльонова та ДНК-полімерази Т4: 5’-3’
– ПОЛІМЕРАЗНА ТА 3’-5’ ЕКЗОНУКЛЕАЗНА АКТИВНОСТІ
Слайд 102ДНК-ПОЛІМЕРАЗА ФАГА Т7
СУБОДИНИЦЯ ТІОРЕДОКСИНУ ПІДВИЩУЄ ПРОЦЕСИВНІСТЬ ПОЛІМЕРАЗИ ДО 2000 –
3000 НУКЛЕОТИДІВ
Слайд 103ДНК-ПОЛІМЕРАЗА ФАГА Т7
Завдяки високій процесивності фермент використовується для копіювання довгих
фрагментів ДНК.
Для секвенування методом Сенгера використовують генно модифіковану полімеразу
Т7 – без 3’-5’- екзонуклеазної активності – такі ферменти називають СЕКВЕНАЗАМИ. Їх процесивність – до 320 нуклеотидів за секунду.Слайд 112ТЕРМОСТАБІЛЬНІ ДНК-ПОЛІМЕРАЗИ
0
Taq
Tth
Pfu
Vent, Deep Vent
тощо
Вперше
відкрита Томасом Броком і Хадсоном Фрізом в районі Великих Фонтанів
Єллоустонского національного парку.Thermus aquaticus
Слайд 1141975 рік – Нобелівська премія - за відкриття, які стосуються
взаємодії між онкогенними вірусами і генетичним матеріалом клітини.
0
Ховард
Темін
Девід
Балтімор
Ренато
Дульбекко