Слайд 120 лет назад потерял руку. Врач посоветовал биоинженерию. Рука выросла
за 3 года. Чтобы проверить, действительно ли ДНК помогла или
я такой регенерирующий, я похитил трёх жителей нашего города, отрубил одному руку, второму - ногу, а третьему - голову. И начал кормить их ДНК. С третьим возникли проблемы, потому что орального отверстия не было.
Сергей Л., Малые Вяземы.
ПЫТЛИВЫЙ УМ – ДЛЯ БИОИНЖЕНЕРА ПЕРВОЕ ДЕЛО!
БИОИНЖЕНЕРИЯ №7
Слайд 2Секвенирование
Сегодня, ребятки, у нас будет переходная лекция! Мы переходим из
одной биоинженерной эпохи в другую.
Первая биоинженерная эпоха: работа с генами
Что
научились делать биоинженеры за время первой эпохи?
«вынимать» ДНК из клетки – выделение и очистка нуклеиновых кислот,
видеть ДНК глазами – электрофорез,
«вынимать» нужные участки из ДНК - рестрикция,
соединять несколько участков ДНК в один - лигирование, клонирование,
«засовывать» ДНК в клетку - трансформация,
многократно увеличивать количество нужного участка ДНК - ПЦР,
определять нуклеотидную последовательность нужного участка ДНК – секвенирование.
Слайд 3Вторая биоинженерная эпоха: работа с геномами
(продолжается по настоящее время)
Что к
сегодняшнему дню научились делать биоинженеры за время второй эпохи?
определять нуклеотидную
последовательность целых геномов – высокопроизводительное секвенирование,
вносить изменения в гены прямо в живой клетке, на уровне целого генома – редактирование генома.
Мало, скажете вы? Да ничего подобного, это просто-таки революция в науке. На следующих лекциях поймете, почему.
Ну а пока останемся на время в первой эпохе и поговорим о секвенировании генов, а не геномов.
Слайд 4Секвенирование по Максаму-Гилберту
Первый метод определения последовательности нуклеотидов ДНК, 1970-е годы
Под
действием различных химических агентов ДНК можно расщепить по G, A/G,
C и C/T.
Образец нужно разделить на 4 части и подвергнуть каждую из частей действию только одного агента.
Концентрации химических агентов подбираются так, чтобы каждая реакция приводила только к одному разрыву цепочки ДНК.
ДНК должна быть мечена по одному концу.
Продукты реакции наносятся на ПААГ, гель подвергается авторадиографии.
Слайд 5Как пометить радиоактивностью двуцепочечную ДНК?
Выступающий 5’-конец – можно пометить ДНК-полимеразой,
добавив меченные нуклеотиды (матричный синтез).
Выступающий 3’-конец – можно пометить терминальной
трансферазой, опять же в присутствии меченных нуклеотидов (нематричный синтез).
Тупые концы – тоже годится терминальная трансфераза.
Проблему мечения обоих концов ДНК обычно решали рестрикцией:
Слайд 6Метод Сэнгера
Появился в конце 1970-х годов и мгновенно вытеснил метод
Максама-Гилберта.
Основа данного метода – дидезоксинуклеотиды.
Если дидезоксинуклеотидтрифосфат (ddNTP) включился в цепочку
ДНК в ходе матричного синтеза, этот самый синтез на нем и закончится – у ddNTP нет 3’-концевой гидроксильной группы, необходимой для формирования фосфодиэфирной связи в ходе полимеризации ДНК.
Если организовать достройку второй цепи ДНК с меченного праймера в присутствии полного набора dNTPs и одного ddNTP с правильно подобранной концентрацией, вы получите набор меченных фрагментов ДНК, заканчивающихся на все имевшиеся в матричной цепи нуклеотиды, комплементарные данному ddNTP.
Слайд 7Пометить олигонуклеотид – ваще не вопрос, пацаны!
1. Обработайте олигонуклеотид щелочной
фосфатазой (АР). Она откусит пирофосфат у 5’-концевого нуклеотида.
2. Обработайте дефосфорилированный
олигонуклеотид Т4-полинуклеотидкиназой (T4 PNK) в присутствии меченного по гамма-положению дАТФ. Меченный фосфат перейдет на 5’-конец олигонуклеотида.
3. Очистите олигонуклеотид от низкомолекулярных соединений, содержащих метку (продукты деградации, сам дАТФ).
4. …
5. PROFIT!!!
Слайд 8Метод Сэнгера
меченный!!!
Проведите 4 независимых реакции удлинения праймера с каждым из
ddNTP, нанесите результат на ПААГ, радиоавтографируйте гель и…
Слайд 9Результат метода Сэнгера
За один прогон на геле можно было прочесть
не больше 100 нуклеотидов.
Слайд 10Сравнение методов секвенирования по Максаму-Гилберту и по Сэнгеру
Неудивительно, что метод
Максама-Гилберта был очень быстро вытеснен методом Сэнгера. Максам-Гилберт остался только
лишь для тех случаев, когда ДНК-полимераза, используемая в процессе по Сэнгеру, не могла пройти сложные вторичные структуры ДНК (псевдоузлы и т.п.).
А теперь прошу обратить внимание на столбец «возможность автоматизации».
Слайд 11Автоматические секвенаторы, использующие метод Сэнгера
Четыре разных флюоресцентных красителя => одна
реакция в одной пробирке
Капиллярный электрофорез => более 1000 нуклеотидов за
одно прочтение
Слайд 12Набор флюоресцентных красителей BigDye для мечения ddNTPs
Слайд 13Автоматический секвенатор дает на выходе набор пиков и сам же
пишет соответствующую нуклеотидную последовательность.
Очень удобно!
Секвенирование по Сэнгеру применяется до
сих пор для решения всех задач, не связанных с анализами геномного уровня.
Слайд 14Высокопроизводительное секвенирование
(секвенирование следующего поколения,
Next Generation Sequencing, NGS)
Общее название нескольких
методов секвенирования, объединенных следующими свойствами (в сравнении с методом Сэнгера):
Упрощение
приборной базы (для конечного пользователя, если говорить о принципах работы – то, безусловно, усложнение)
Удешевление процесса,
Увеличение эффективности чтения ДНК (возможность читать более длинные фрагменты или же читать ДНК быстрее),
Более высокая степень автоматизации процесса.
И мы наконец переходим во вторую биоинженерную эпоху!
Слайд 15Пиросеквенирование (Sequencing By Synthesis)
Метод основан на детекции пирофосфата, высвобождающегося при
присоединении каждого нового нуклеотида к растущей цепи ДНК в ходе
ПЦР.
Длина одного рида: до 500 нуклеотидов, в современных приборах чаще до 100.
Используется, в основном, для ресеквенирования, то есть для повторного секвенирования геномов, прочтенных другими методами. Помимо этого, применяется для генотипирования и для определения статуса метилирования ДНК.
Являлось первой по времени технологией NGS, первоначально использовалось и для первичного секвенирования. По сравнению с методом Сэнгера (поскольку тогда сравнивать больше было не с чем) метод был фееричен.
Основное достоинство: высокая точность.
Основной недостаток: высокая длительность анализа по сравнению с другими технологиями NGS.
Первый прибор: компания 454 Life Sciences, 2005 год.
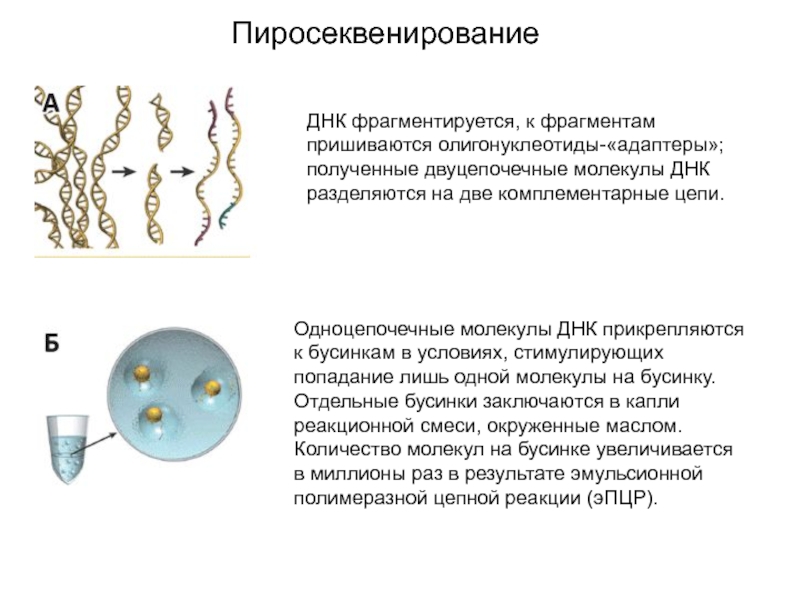
Слайд 16Пиросеквенирование
ДНК фрагментируется, к фрагментам пришиваются олигонуклеотиды-«адаптеры»; полученные двуцепочечные молекулы ДНК разделяются
на две комплементарные цепи.
Одноцепочечные молекулы ДНК прикрепляются к бусинкам в условиях, стимулирующих попадание
лишь одной молекулы на бусинку. Отдельные бусинки заключаются в капли реакционной смеси, окруженные маслом. Количество молекул на бусинке увеличивается в миллионы раз в результате эмульсионной полимеразной цепной реакции (эПЦР).
Слайд 17Пиросеквенирование. Схема эПЦР.
Бусинка покрыта адаптерными олигонуклеотидами.
В каплю попадает один фрагмент
ДНК…
и амплифицируется!
Слайд 18Пиросеквенирование
Эмульсия разбивается, и цепи ДНК-фрагментов, образовавшиеся в результате эПЦР, разделяются. Бусинки, несущие
на своей поверхности миллионы одноцепочечных копий первоначального фрагмента ДНК, помещаются в лунки
оптико-волоконного слайда, по одной в каждую лунку.
В каждую лунку добавляются бусинки поменьше, несущие на своей поверхности ферменты и другие молекулы, необходимые для пиросеквенирования.
Слайд 19Пиросеквенирование
ДНК– полимераза катализирует добавление dNTP к праймеру для секвенирования, если
он комплементарен к последовательности матрицы ДНК. Каждое присоединение к цепи
сопровождается выделением пирофосфата (PPi) в количестве, эквимолярном количеству встроившихся нуклеотидов.
АТФ-сульфурилаза превращает пирофосфат в АТФ в присутствии аденозин-5’- фосфосульфата.
Люцифераза запускает АТФ-опосредованную реакцию окисления люциферина в оксилюциферин, в результате чего происходит образование видимого света в количестве, пропорциональном количеству АТФ.
В течение всей реакции апираза деградирует невстроившиеся нуклеотиды и избыток АТФ. После завершения деградации нуклеотидов, не встроившихся в цепь ДНК, добавляется новый нуклеотид.
Добавление нуклеотидов осуществляется последовательно.
Слайд 20Результат пиросеквенирования
Отсутствие пика – что-то пошло не так.
Пик в два
раза выше обычного – два одинаковых нуклеотида подряд.
Отсюда вытекает
еще один существенный недостаток данной технологии: неспособность определять последовательности, состоящие из много раз повторенных одинаковых нуклеотидов (гомоолигомерные участки ДНК). Таких участков в геномах полно.
Слайд 21Ионное полупроводниковое секвенирование
(ion semiconductor sequencing)
В целом, этот метод тоже можно
назвать Sequencing by Synthesis. Его принцип очень похож на принцип
пиросеквенирования, только детектируется не свет, а протоны, то есть изменение рН.
Длина одного рида: до 400 нуклеотидов, в современных приборах чаще до 200.
Используется для всех возможных задач NGS, но только если геном не очень большой. На большие геномы метода не хватает.
Основное достоинство: относительно низкая цена, быстрота.
Основной недостаток: невысокая точность прочтения гомополимерных участков, невысокая производительность.
Первый прибор: компания Ion Torrent Systems, 2010 год.
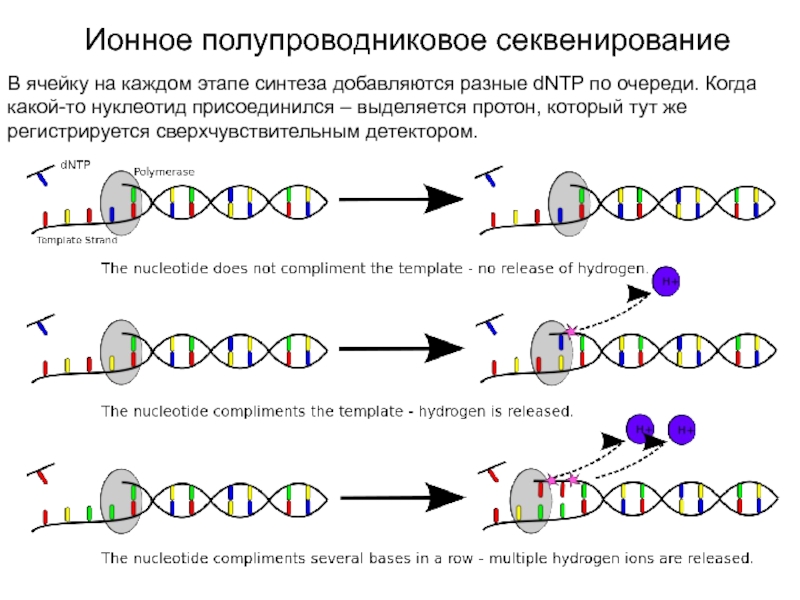
Слайд 22Ионное полупроводниковое секвенирование
В ячейку на каждом этапе синтеза добавляются разные
dNTP по очереди. Когда какой-то нуклеотид присоединился – выделяется протон,
который тут же регистрируется сверхчувствительным детектором.
Слайд 23Ионное полупроводниковое секвенирование
Вот у вас и получается нуклеотидная последовательность!
Слайд 24Циклическое лигазное секвенирование
(Sequencing By Ligation)
Метод основан на детекции флюорофора
при лигировании олигонуклеотида к секвенируемой цепи ДНК.
Длина одного рида: до
75 нуклеотидов.
Широко использовался для всех задач, связанных с секвенированием. В настоящее время (c 2016 года) поддержка технологии основными игроками на рынке прекращена, то есть технология не выдержала конкуренции.
Основное достоинство: высокая точность.
Основной недостаток: слишком короткие риды.
Первый прибор: SOLiD от компании Applied Biosystems, 2006 год.
Слайд 25Циклическое лигазное секвенирование
Первые этапы технологии аналогичны таковым для пиросеквенирования:
Фрагментация ДНК
Пришивание
адаптеров
Распределение фрагментов ДНК по магнитным бусинам
Эмульсионная ПЦР
По окончании эПЦР все
3’-концы амплифицированных фрагментов ДНК специальным образом модифицируются…
Слайд 26Циклическое лигазное секвенирование
… и посредством этой модификации закрепляются в проточных
ячейках.
В ячейки сначала добавляется обычный олигонуклеотид, комплементарный адаптеру, который использовался
для эПЦР. После этого добавляется смесь хитрых олигонуклеотидов, меченных четырьмя разными флюорофорами, длиной 8 нуклеотидов каждый:
X-Y-n-n-n-Z-Z-Z-флюорофор
X, Y – два каких-то конкретных нуклеотида,
n – вырожденный нуклеотид (любой из четырех),
Z – некое производное нуклеотида, которое не может никуда отжечься (что это такое именно – производители не раскрывают, скорее всего инозин).
Слайд 27Циклическое лигазное секвенирование
Универсальный олигонуклеотид отжегся на секвенируемую ДНК.
Среди хитрых нуклеотидов
предпочтительно будут отжигаться те, которые максимально комплементарны секвенируемой ДНК, то
есть те, у которых X и Y комплементарны двум нуклеотидам ДНК, следующим сразу после адаптера.
Напоминаю: нуклеотиды n вырожденные, они в любом случае отожгутся.
Добавьте ДНК-лигазу – и она сошьет связавшийся с ДНК хитрый олиг с универсальным.
Отмойте все, что не связалось и не лигировалось, и детектируйте флюоресценцию.
X-Y-n-n-n-Z-Z-Z-флюорофор
Слайд 28Циклическое лигазное секвенирование
Итак, X-Y-n-n-n у вас теперь лигированы к универсальному
олигонуклеотиду и находятся в составе дуплекса с секвенируемой ДНК. Для
Z-Z-Z второе неверно – они висят наружу, так как изначально не могли никуда отжечься. Добавьте специальное «режущее вещество», и оно (1) отрежет Z-Z-Z вместе с флюорофором и (2) оставит 5’-концевой фосфат на части олигонуклеотида в составе дуплекса.
«Режущее вещество» производителями не раскрывается, скорее всего – соль серебра.
X-Y-n-n-n-Z-Z-Z-флюорофор
Повторите всю процедуру сначала!
В результате вы будете знать каждые два нуклеотида из пяти, то есть нуклеотиды в положениях 1, 2, 6, 7, 11, 12 и т.д.
Слайд 29Циклическое лигазное секвенирование
Когда вы дошли до конца секвенируемой молекулы, уберите
с нее все, что там отожглось и лигировалось, а затем
отожгите на нее новый универсальный праймер. Если принять положение, на котором кончался предыдущий, за n, то новый должен кончаться на n-1, то есть быть на один нуклеотид ближе к 5’-концу секвенируемой молекулы.
Повторите все сначала – и у вас определятся уже другие пары нуклеотидов! Для предыдущего универсального олига на первом шаге определялись нуклеотиды n+1 и n+2, а для этого – n и n+1.
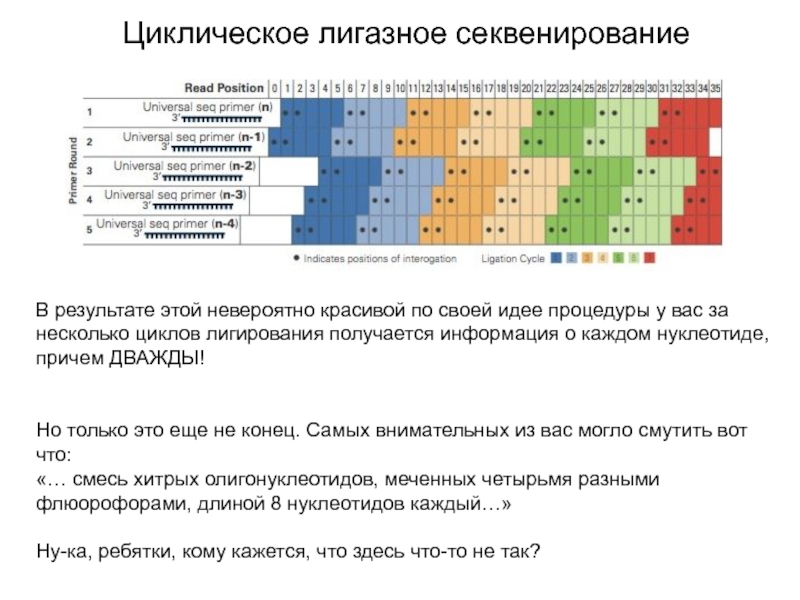
Слайд 30Циклическое лигазное секвенирование
В результате этой невероятно красивой по своей идее
процедуры у вас за несколько циклов лигирования получается информация о
каждом нуклеотиде, причем ДВАЖДЫ!
Но только это еще не конец. Самых внимательных из вас могло смутить вот что:
«… смесь хитрых олигонуклеотидов, меченных четырьмя разными флюорофорами, длиной 8 нуклеотидов каждый…»
Ну-ка, ребятки, кому кажется, что здесь что-то не так?
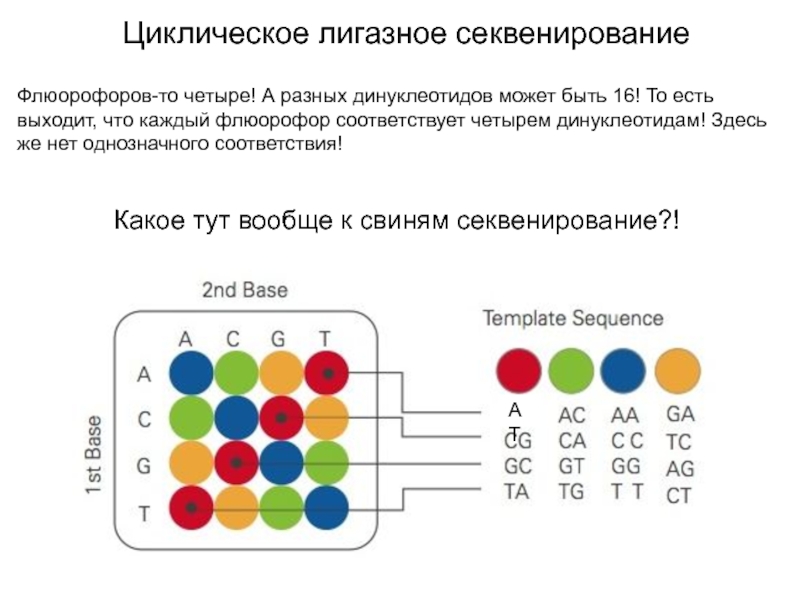
Слайд 31Циклическое лигазное секвенирование
Флюорофоров-то четыре! А разных динуклеотидов может быть 16!
То есть выходит, что каждый флюорофор соответствует четырем динуклеотидам! Здесь
же нет однозначного соответствия!
Какое тут вообще к свиням секвенирование?!
АТ
Слайд 32Циклическое лигазное секвенирование
Секвенирование очень даже хорошее!
Вы же знаете нуклеотид в
положении n-1! Это же последний нуклеотид универсального олига! И при
этом вы его отсеквенировали в составе пары n-1/n, и эта пара дала вам, допустим, зеленый цвет. И вы знаете, что n-1 – это G. Значит, n – это T! А пара n/n+1 дала вам желтый цвет. Значит, n+1 – это А!
И так далее.
АТ