Слайд 1Гомологическая рекомбинация
Встречается гораздо чаще, чем сайт-специфическая рекомбинация. Надо полагать, что
она происходит вообще в любом живом организме на Земле.
Гомологическая рекомбинация
происходит между ЛЮБЫМИ двумя последовательностями ДНК, которые либо совершенно одинаковы, либо почти-почти одинаковы.
Минимальная известная на сегодняшний день длина участка эффективной гомологической рекомбинации – 23 нуклеотида.
Гомологическая рекомбинация обладает фундаментальной важностью для нормального функционирования клетки, в особенности бактериальной. В наши дни бытует мнение, что важнейшая роль этого процесса заключается в участии в бактериальной репликации, а именно в поддержании нормальной структуры вилки репликации. Ведь ориджин у бактерий один, и если что-то там пойдет не так, никакого запасного варианта нет!
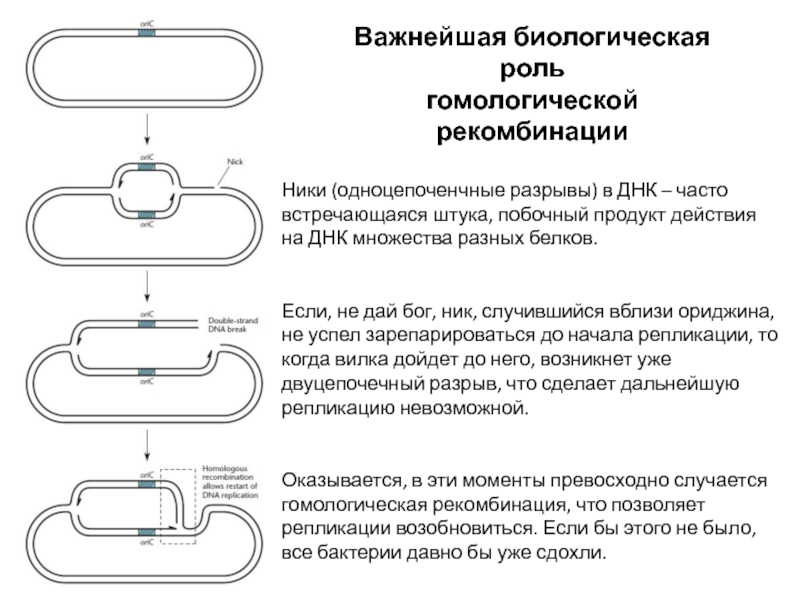
Слайд 2Ники (одноцепоченчные разрывы) в ДНК – часто встречающаяся штука, побочный
продукт действия на ДНК множества разных белков.
Если, не дай бог,
ник, случившийся вблизи ориджина, не успел зарепарироваться до начала репликации, то когда вилка дойдет до него, возникнет уже двуцепочечный разрыв, что сделает дальнейшую репликацию невозможной.
Оказывается, в эти моменты превосходно случается гомологическая рекомбинация, что позволяет репликации возобновиться. Если бы этого не было, все бактерии давно бы уже сдохли.
Важнейшая биологическая роль
гомологической рекомбинации
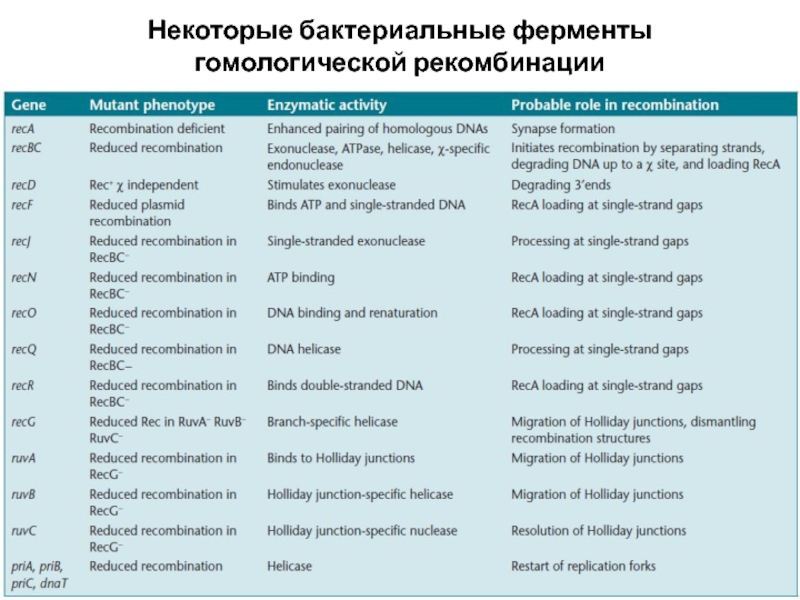
Слайд 3Некоторые бактериальные ферменты
гомологической рекомбинации
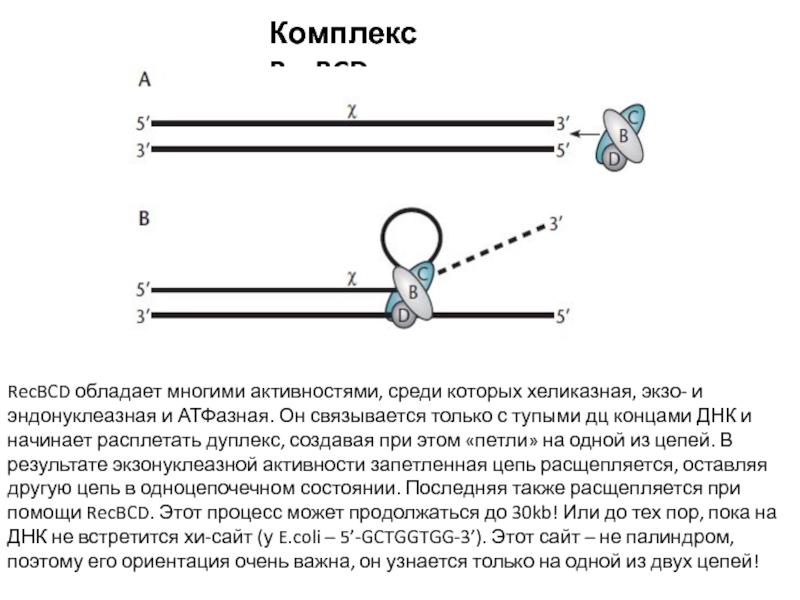
Слайд 4Комплекс RecBCD
RecBCD обладает многими активностями, среди которых хеликазная, экзо- и
эндонуклеазная и АТФазная. Он связывается только с тупыми дц концами
ДНК и начинает расплетать дуплекс, создавая при этом «петли» на одной из цепей. В результате экзонуклеазной активности запетленная цепь расщепляется, оставляя другую цепь в одноцепочечном состоянии. Последняя также расщепляется при помощи RecBCD. Этот процесс может продолжаться до 30kb! Или до тех пор, пока на ДНК не встретится хи-сайт (у E.coli – 5’-GCTGGTGG-3’). Этот сайт – не палиндром, поэтому его ориентация очень важна, он узнается только на одной из двух цепей!
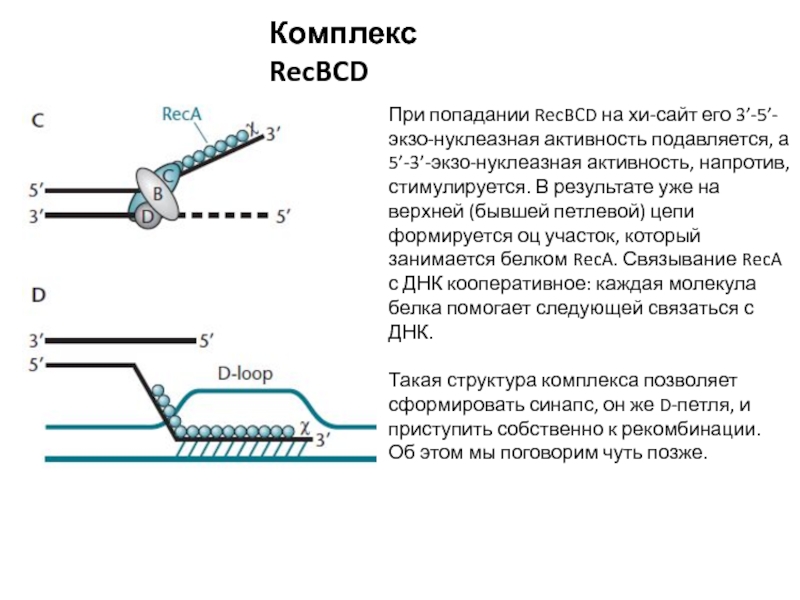
Слайд 5Комплекс RecBCD
При попадании RecBCD на хи-сайт его 3’-5’-экзо-нуклеазная активность подавляется,
а 5’-3’-экзо-нуклеазная активность, напротив, стимулируется. В результате уже на верхней
(бывшей петлевой) цепи формируется оц участок, который занимается белком RecA. Связывание RecA с ДНК кооперативное: каждая молекула белка помогает следующей связаться с ДНК.
Такая структура комплекса позволяет сформировать синапс, он же D-петля, и приступить собственно к рекомбинации. Об этом мы поговорим чуть позже.
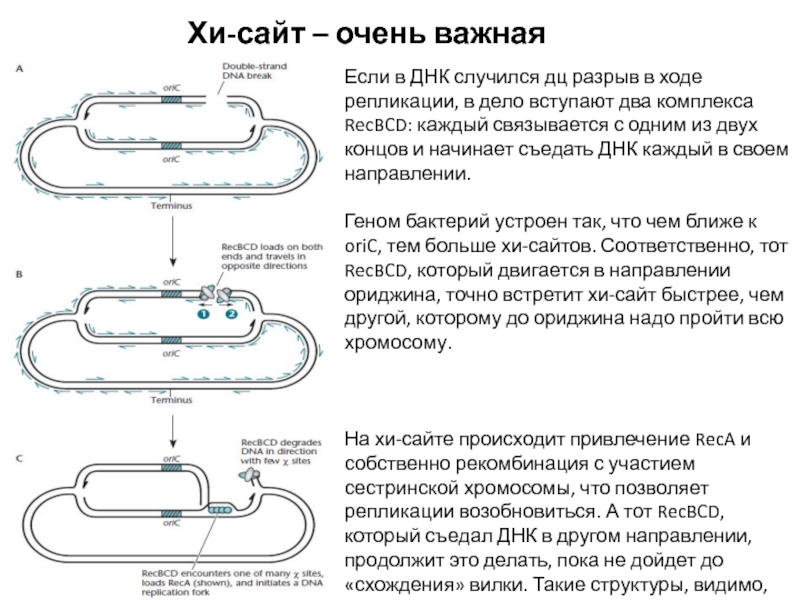
Слайд 6Хи-сайт – очень важная штука
Если в ДНК случился дц разрыв
в ходе репликации, в дело вступают два комплекса RecBCD: каждый
связывается с одним из двух концов и начинает съедать ДНК каждый в своем направлении.
Геном бактерий устроен так, что чем ближе к oriC, тем больше хи-сайтов. Соответственно, тот RecBCD, который двигается в направлении ориджина, точно встретит хи-сайт быстрее, чем другой, которому до ориджина надо пройти всю хромосому.
На хи-сайте происходит привлечение RecA и собственно рекомбинация с участием сестринской хромосомы, что позволяет репликации возобновиться. А тот RecBCD, который съедал ДНК в другом направлении, продолжит это делать, пока не дойдет до «схождения» вилки. Такие структуры, видимо, для него непреодолимы, и он отваливается. И репликация продолжается!
Слайд 7RecF-рекомбинация
Используется в случае оц разрывов ДНК, но редко – их
проще сразу достроить
Хеликаза RecQ и нуклеаза RecJ связываются с
местом ника и подъедают одну цепочку ДНК (как они понимают, когда надо остановиться – не очень понятно).
Оц ДНК покрывается белков SSB.
После остановки RecQ и RecJ отваливаются, но приходит комплекс RecFOR, который осуществляет замену SSB на RecA, помогая связаться первой молекуле RecA, а уж дальше проявляется кооперативность связывания.
ДНК, покрытая RecA – субстрат для формирования синапса.
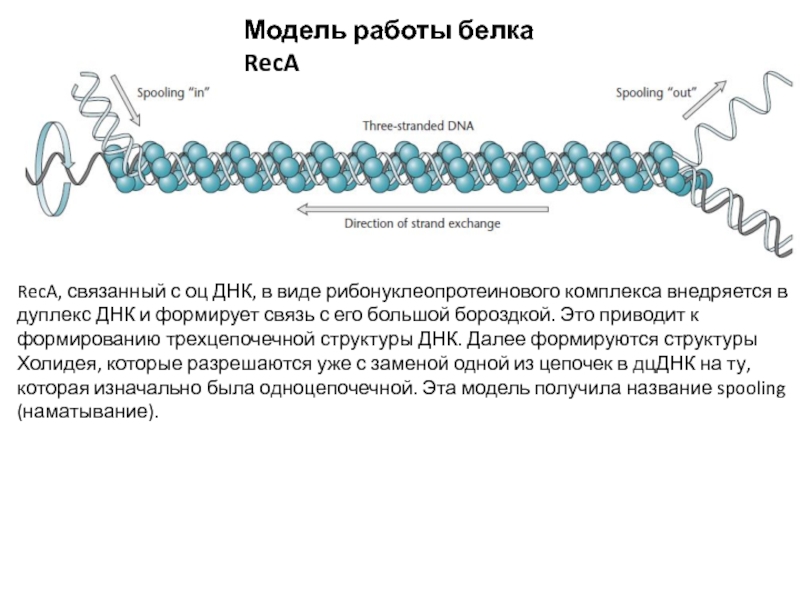
Слайд 8Модель работы белка RecA
RecA, связанный с оц ДНК, в виде
рибонуклеопротеинового комплекса внедряется в дуплекс ДНК и формирует связь с
его большой бороздкой. Это приводит к формированию трехцепочечной структуры ДНК. Далее формируются структуры Холидея, которые разрешаются уже с заменой одной из цепочек в дцДНК на ту, которая изначально была одноцепочечной. Эта модель получила название spooling (наматывание).
Слайд 9Структуры Холидея, образующиеся после спулинга
Та самая трехцепочечная структура способна сформировать
структуру Холидея. Она может либо разрешиться сразу, приведя к замене
определенного участка ДНК, либо через изомеризацию (происходящую спонтанно) принять другую конформацию и уж потом разрешиться, что приведет к обмену «половинами» ДНК, то есть к существенно большим изменениям, чем разрешение первой структуры.
Пожалуйста, не спрашивайте меня о подробностях, у меня совсем нет пространственного воображения, я 20 лет не понимаю, как работают структуры Холидея!!!
Слайд 10Ruv-белки формируют структуры Холидея
Все начинается с той же структуры, с
которой все начиналось на предыдущем слайде и которая сформировалась в
результате спулинга RecA. С ней связываются два тетрамера RuvA, что приводит к формированию структуры Холидея.
После этого приходят гексамеры RuvB, каждый из которых связывается с одним тетрамером RuvA и формирует кольцо вокруг одной цепочки ДНК. После этого ДНК АТФ-зависимым образом протаскивается через такое кольцо, что приводит к удлинению гетеродуплексов, то есть участков, в которых между собой спарены цепочки разных исходных ДНК. Это называется миграция структуры Холидея.
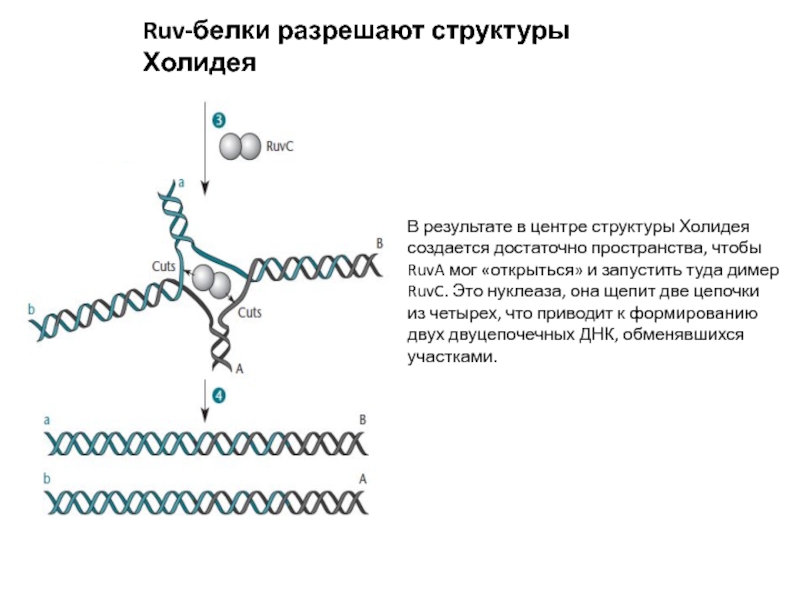
Слайд 11Ruv-белки разрешают структуры Холидея
В результате в центре структуры Холидея создается
достаточно пространства, чтобы RuvA мог «открыться» и запустить туда димер
RuvC. Это нуклеаза, она щепит две цепочки из четырех, что приводит к формированию двух двуцепочечных ДНК, обменявшихся участками.
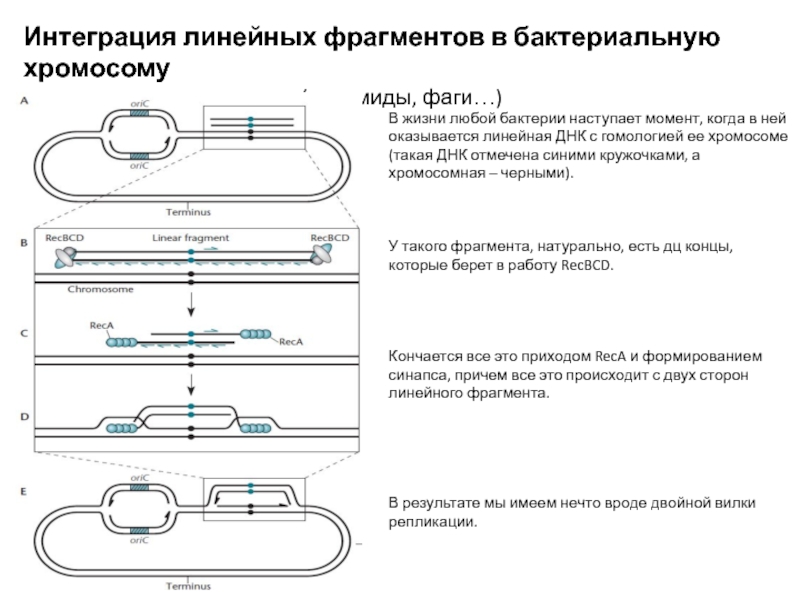
Слайд 12Интеграция линейных фрагментов в бактериальную хромосому
(плазмиды, фаги…)
В жизни любой бактерии
наступает момент, когда в ней оказывается линейная ДНК с гомологией
ее хромосоме (такая ДНК отмечена синими кружочками, а хромосомная – черными).
У такого фрагмента, натурально, есть дц концы, которые берет в работу RecBCD.
Кончается все это приходом RecA и формированием синапса, причем все это происходит с двух сторон линейного фрагмента.
В результате мы имеем нечто вроде двойной вилки репликации.
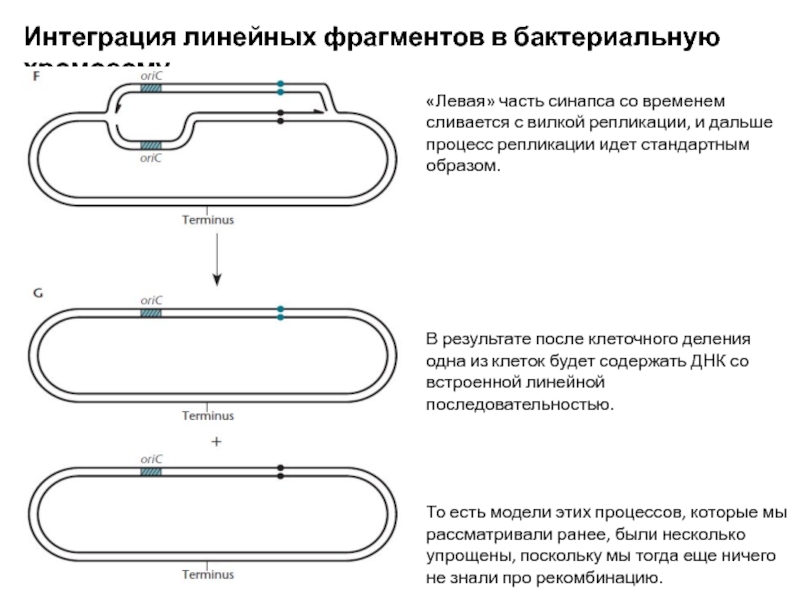
Слайд 13Интеграция линейных фрагментов в бактериальную хромосому
«Левая» часть синапса со временем
сливается с вилкой репликации, и дальше процесс репликации идет стандартным
образом.
В результате после клеточного деления одна из клеток будет содержать ДНК со встроенной линейной последовательностью.
То есть модели этих процессов, которые мы рассматривали ранее, были несколько упрощены, поскольку мы тогда еще ничего не знали про рекомбинацию.
Слайд 14Репарация ДНК
Репарация есть процесс исправления повреждений в ДНК.
ДНК подвержена воздействиям
многих факторов, среди которых УФ-облучение, разнообразные химические вещества и т.п.
Все это приводит к ее повреждениям. А ведь любое повреждение в ДНК – это мутация! А значит, ее надо исправить, иначе клетка может погибнуть.
Помимо этого, репликационный аппарат клетки ведь тоже не идеально точен. Ошибки репликации, то есть неправильно вставленные нуклеотиды, тоже надо исправлять.
Вот всем этим и занимаются клеточные системы репарации.
Сейчас мы довольно поверхностно пройдемся по основным типам повреждений ДНК и по путям их репарации.
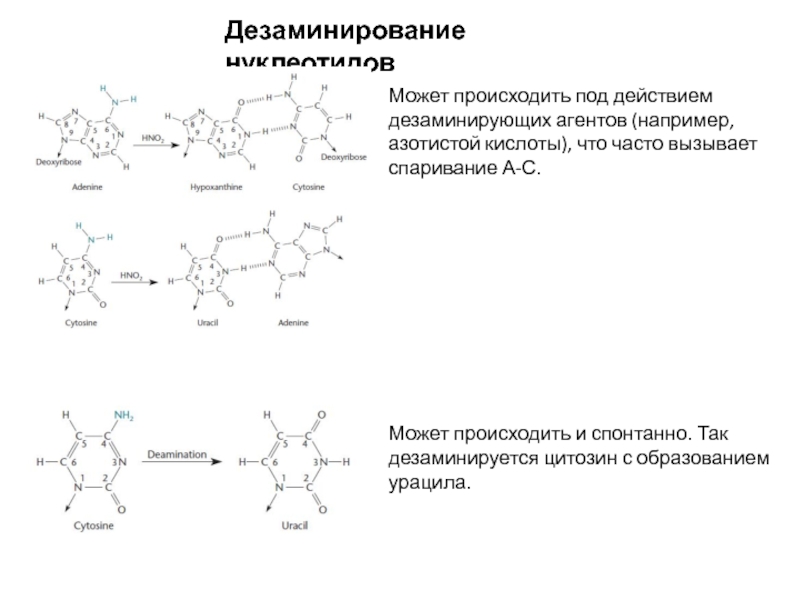
Слайд 15Дезаминирование нуклеотидов
Может происходить под действием дезаминирующих агентов (например, азотистой кислоты),
что часто вызывает спаривание А-С.
Может происходить и спонтанно. Так дезаминируется
цитозин с образованием урацила.
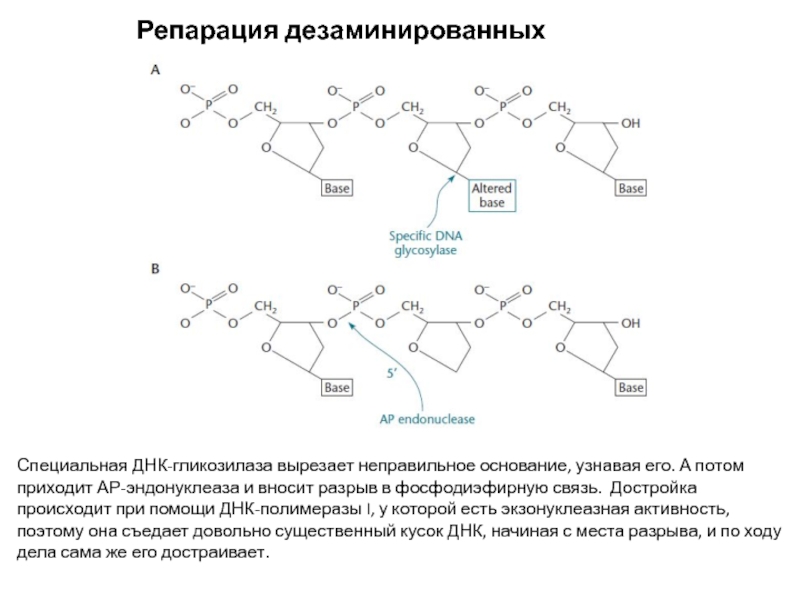
Слайд 16Репарация дезаминированных нуклеотидов
Специальная ДНК-гликозилаза вырезает неправильное основание, узнавая его. А
потом приходит АР-эндонуклеаза и вносит разрыв в фосфодиэфирную связь. Достройка
происходит при помощи ДНК-полимеразы I, у которой есть экзонуклеазная активность, поэтому она съедает довольно существенный кусок ДНК, начиная с места разрыва, и по ходу дела сама же его достраивает.
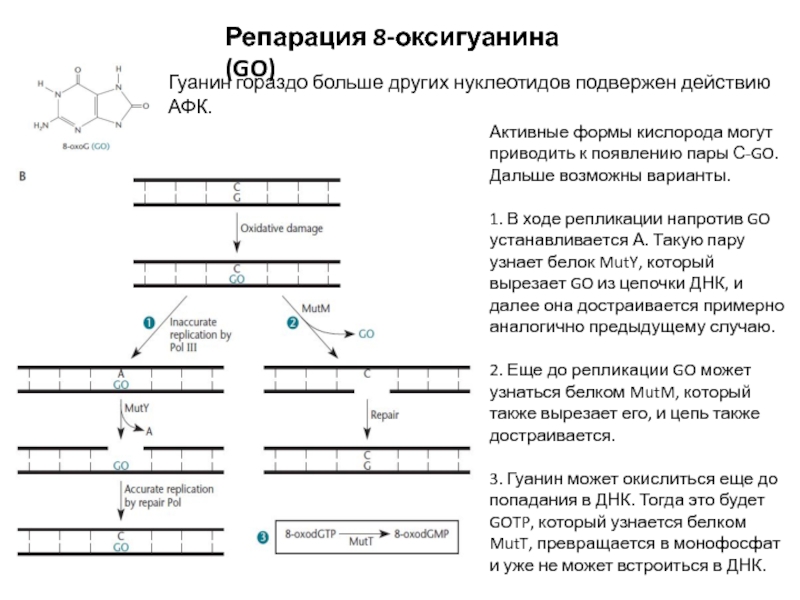
Слайд 17Репарация 8-оксигуанина (GO)
Гуанин гораздо больше других нуклеотидов подвержен действию АФК.
Активные
формы кислорода могут приводить к появлению пары С-GO. Дальше возможны
варианты.
1. В ходе репликации напротив GO устанавливается А. Такую пару узнает белок MutY, который вырезает GO из цепочки ДНК, и далее она достраивается примерно аналогично предыдущему случаю.
2. Еще до репликации GO может узнаться белком MutM, который также вырезает его, и цепь также достраивается.
3. Гуанин может окислиться еще до попадания в ДНК. Тогда это будет GOTP, который узнается белком MutT, превращается в монофосфат и уже не может встроиться в ДНК.
Слайд 18Алкилирование гуанина
Здесь представлен самый частый случай алкилирования, приводящий к образованию
О6-метилгуанина. Он может спариваться с тимином.
Репарация таких нуклеотидов может происходить
по-разному. Самый простой способ – специфические N-гликозилазы, узнающие неправильные пары с участием алкилированных нуклеотидов и вырезающие эти нуклеотиды. Цепочка потом достраивается полимеразой I, как в случаях выше.
Однако есть более интересный вариант, связанный с метилтрансферазами.
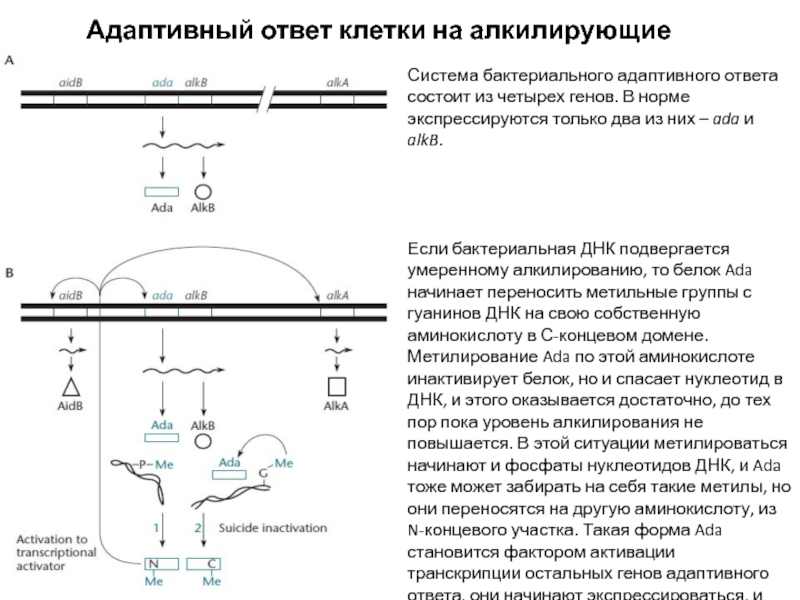
Слайд 19Адаптивный ответ клетки на алкилирующие агенты
Система бактериального адаптивного ответа состоит
из четырех генов. В норме экспрессируются только два из них
– ada и alkB.
Если бактериальная ДНК подвергается умеренному алкилированию, то белок Ada начинает переносить метильные группы с гуанинов ДНК на свою собственную аминокислоту в С-концевом домене. Метилирование Ada по этой аминокислоте инактивирует белок, но и спасает нуклеотид в ДНК, и этого оказывается достаточно, до тех пор пока уровень алкилирования не повышается. В этой ситуации метилироваться начинают и фосфаты нуклеотидов ДНК, и Ada тоже может забирать на себя такие метилы, но они переносятся на другую аминокислоту, из N-концевого участка. Такая форма Ada становится фактором активации транскрипции остальных генов адаптивного ответа, они начинают экспрессироваться, и адаптивный ответ включается на полную катушку!
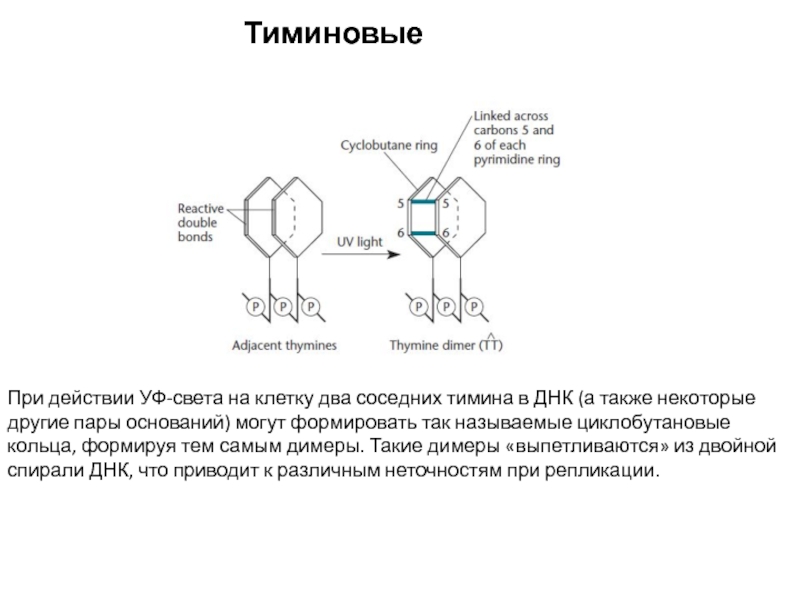
Слайд 20Тиминовые димеры
При действии УФ-света на клетку два соседних тимина в
ДНК (а также некоторые другие пары оснований) могут формировать так
называемые циклобутановые кольца, формируя тем самым димеры. Такие димеры «выпетливаются» из двойной спирали ДНК, что приводит к различным неточностям при репликации.
Слайд 21Репарация (фотореактивация) тиминовых димеров
Процесс осуществляется специальными фотолиазами, которые активируются светом
видимого диапазона. После такой активации они способны разрезать связи, сформировавшие
циклобутановый димер. Это полностью восстанавливает первоначальную структуру ДНК.
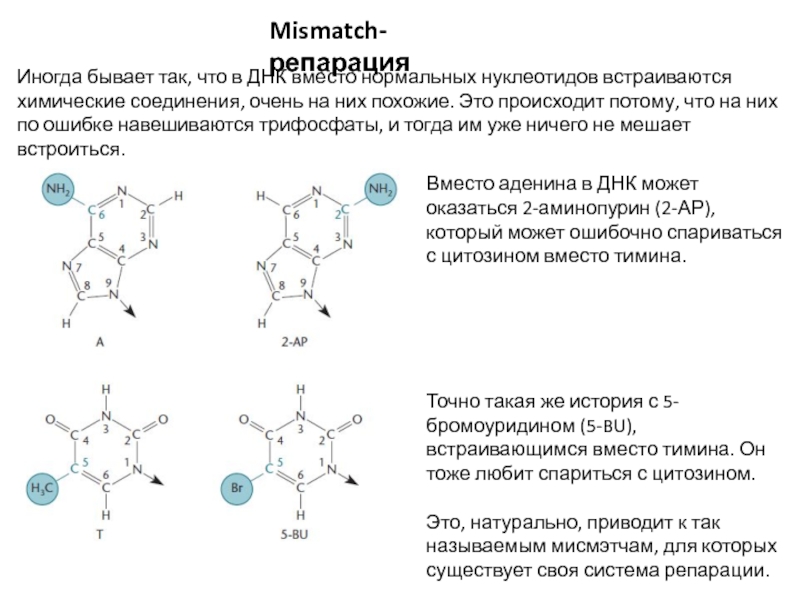
Слайд 22Mismatch-репарация
Иногда бывает так, что в ДНК вместо нормальных нуклеотидов встраиваются
химические соединения, очень на них похожие. Это происходит потому, что
на них по ошибке навешиваются трифосфаты, и тогда им уже ничего не мешает встроиться.
Вместо аденина в ДНК может оказаться 2-аминопурин (2-АР), который может ошибочно спариваться с цитозином вместо тимина.
Точно такая же история с 5-бромоуридином (5-BU), встраивающимся вместо тимина. Он тоже любит спариться с цитозином.
Это, натурально, приводит к так называемым мисмэтчам, для которых существует своя система репарации.
Слайд 23Mismatch-репарация
Основной вопрос, встающий перед системой: в какой цепи ошибочный нуклеотид?
Ответить на него помогает метилирование. Ведь ошибки случаются при репликации,
значит, они сосредоточены во вновь синтезированной цепи. Если подобраться к ней сразу после репликации, то она еще не успеет метилироваться по GATC-сайтам, как ей положено. А родительская цепь метилирована! Поэтому система musmatch-репарации работает с неметилированной цепью , то есть вырезает из нее неправильный нуклеотид. Цепь достраивается и только потом, уже будучи правильной, метилируется.
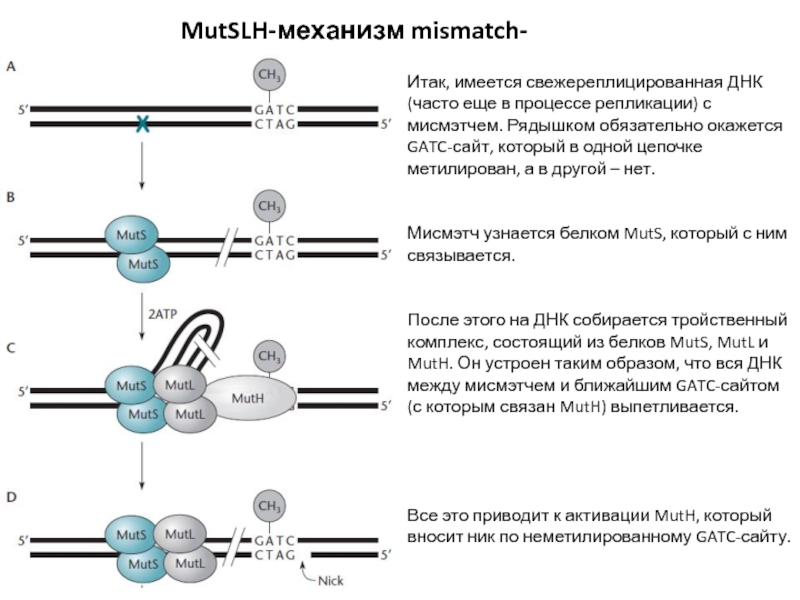
Слайд 24MutSLH-механизм mismatch-репарации
Итак, имеется свежереплицированная ДНК (часто еще в процессе репликации)
с мисмэтчем. Рядышком обязательно окажется GATC-сайт, который в одной цепочке
метилирован, а в другой – нет.
Мисмэтч узнается белком MutS, который с ним связывается.
После этого на ДНК собирается тройственный комплекс, состоящий из белков MutS, MutL и MutH. Он устроен таким образом, что вся ДНК между мисмэтчем и ближайшим GATC-сайтом (с которым связан MutH) выпетливается.
Все это приводит к активации MutH, который вносит ник по неметилированному GATC-сайту.
Слайд 25MutSLH-механизм mismatch-репарации
Здесь в дело вступают экзонуклеазы, превращающие ник в «gap»,
и хеликаза UvrD, чувствующая, где сидят Mut-белки, и в зависимости
от этого определяющая направление работы нуклеаз. Дойдя до MutS, нуклеазы останавливаются, а Mut-белки после этого уходят из комплекса с ДНК.
Приходят полимераза III и лигаза, которые восстанавливают нормальную двуцепочечную структуру ДНК. Поскольку мисмэтч – это вероятностный процесс (аналоги нуклеотидов чаще связываются с нормальной парой, чем с ошибочной), здесь все надеются, что мисмэтча на том же месте не повторится.
И в самом конце появляется Dam-метилаза, которая метилирует дочернюю цепь по GATC-сайтам.
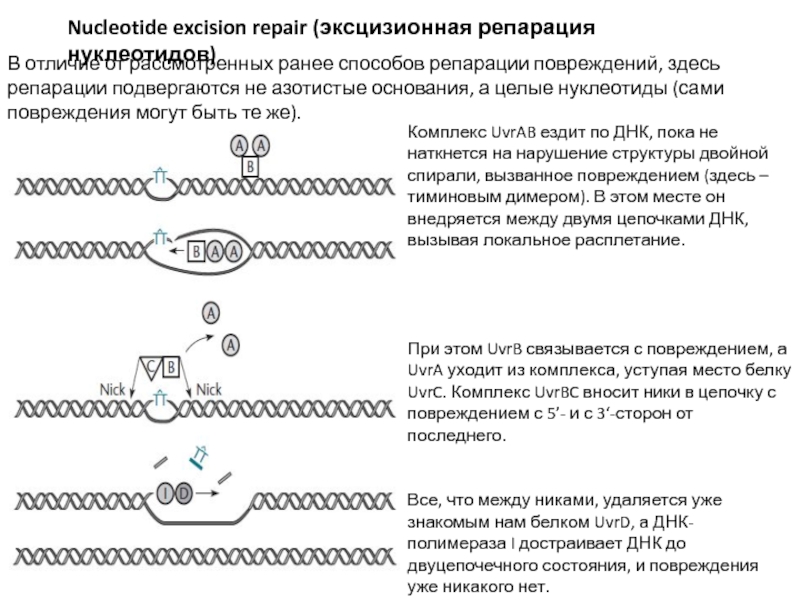
Слайд 26Nucleotide excision repair (эксцизионная репарация нуклеотидов)
В отличие от рассмотренных ранее
способов репарации повреждений, здесь репарации подвергаются не азотистые основания, а
целые нуклеотиды (сами повреждения могут быть те же).
Комплекс UvrAB ездит по ДНК, пока не наткнется на нарушение структуры двойной спирали, вызванное повреждением (здесь – тиминовым димером). В этом месте он внедряется между двумя цепочками ДНК, вызывая локальное расплетание.
При этом UvrB связывается с повреждением, а UvrA уходит из комплекса, уступая место белку UvrC. Комплекс UvrBC вносит ники в цепочку с повреждением с 5’- и с 3‘-сторон от последнего.
Все, что между никами, удаляется уже знакомым нам белком UvrD, а ДНК-полимераза I достраивает ДНК до двуцепочечного состояния, и повреждения уже никакого нет.
Слайд 27Связь репарации и репликации
Мы говорили о тех случаях, когда репарация
случилась до репликации. Тогда все действи-тельно в порядке: повреждение успевает
исправиться до того, как ДНК реплицировалась. А как быть, если оно не успело исправиться, а вилка репликации уже тут как тут?
Если повреждение на отстающей цепи, то все достаточно просто (ситуацию с лидирующей цепью мы рассматривать не будем, она очень сложная и не до конца понятная).
Ну то есть сначала все плохо. Получается, что полимераза останавливается на повреждении.
Репликация продолжается только со следующего фрагмента Окадзаки, оставляя нереплицированной кусок отстающей цепи.
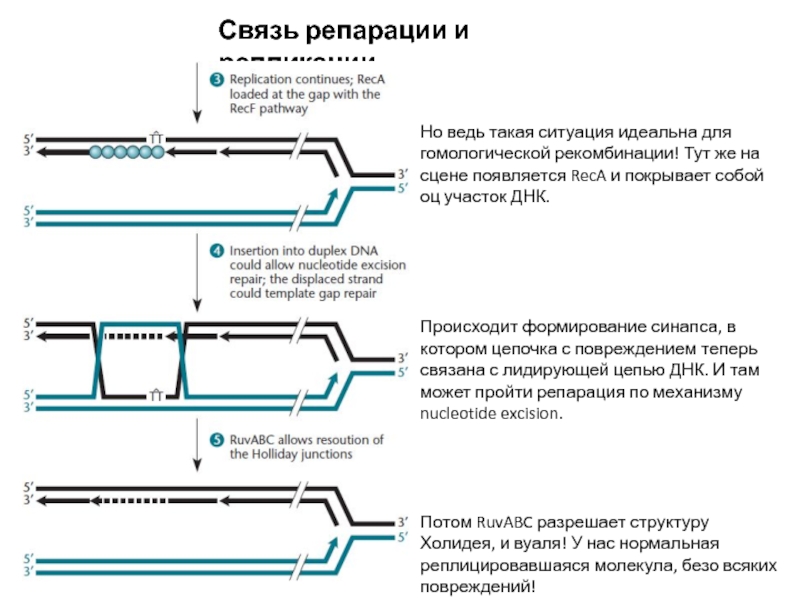
Слайд 28Связь репарации и репликации
Но ведь такая ситуация идеальна для гомологической
рекомбинации! Тут же на сцене появляется RecA и покрывает собой
оц участок ДНК.
Происходит формирование синапса, в котором цепочка с повреждением теперь связана с лидирующей цепью ДНК. И там может пройти репарация по механизму nucleotide excision.
Потом RuvABC разрешает структуру Холидея, и вуаля! У нас нормальная реплицировавшаяся молекула, безо всяких повреждений!
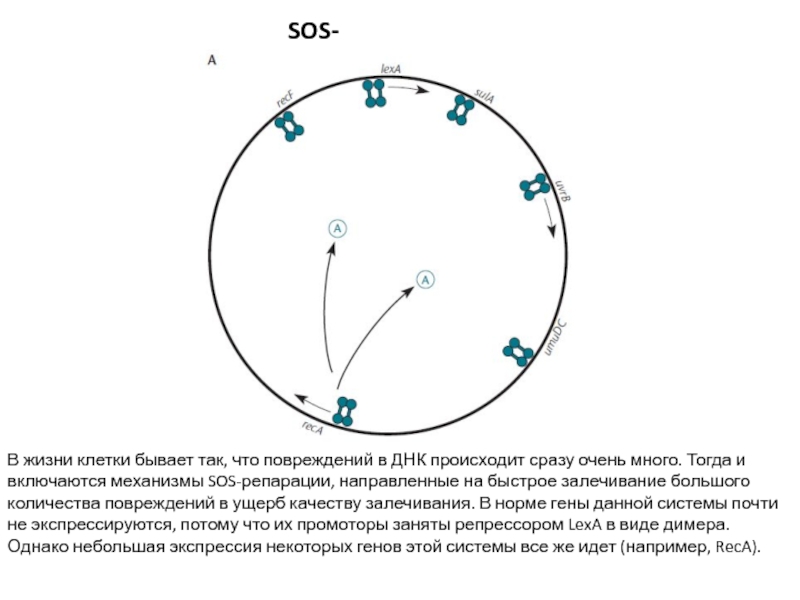
Слайд 29SOS-репарация
В жизни клетки бывает так, что повреждений в ДНК происходит
сразу очень много. Тогда и включаются механизмы SOS-репарации, направленные на
быстрое залечивание большого количества повреждений в ущерб качеству залечивания. В норме гены данной системы почти не экспрессируются, потому что их промоторы заняты репрессором LexA в виде димера. Однако небольшая экспрессия некоторых генов этой системы все же идет (например, RecA).
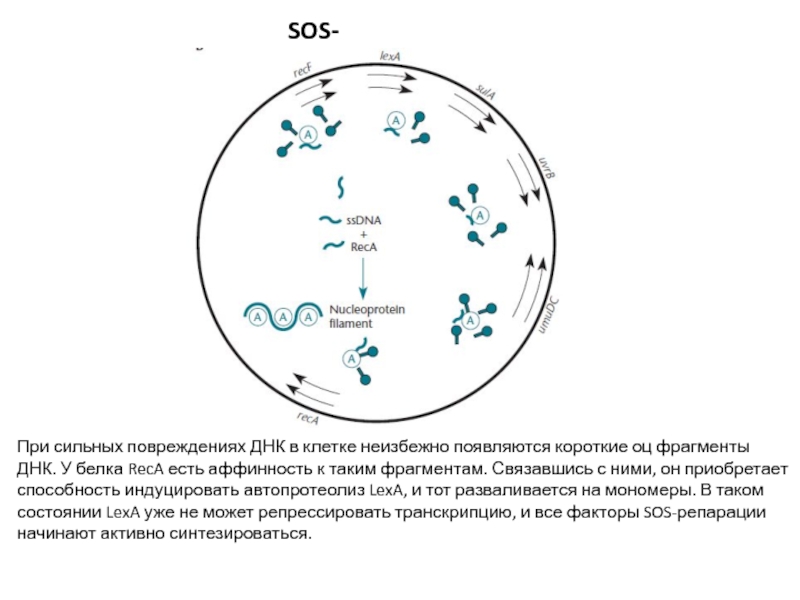
Слайд 30SOS-репарация
При сильных повреждениях ДНК в клетке неизбежно появляются короткие оц
фрагменты ДНК. У белка RecA есть аффинность к таким фрагментам.
Связавшись с ними, он приобретает способность индуцировать автопротеолиз LexA, и тот разваливается на мономеры. В таком состоянии LexA уже не может репрессировать транскрипцию, и все факторы SOS-репарации начинают активно синтезироваться.
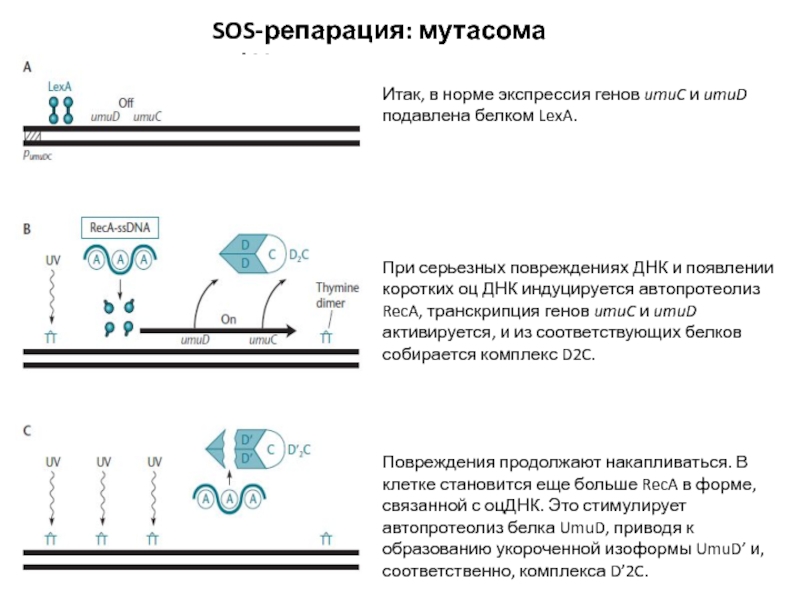
Слайд 31SOS-репарация: мутасома pol V
Итак, в норме экспрессия генов umuC и
umuD подавлена белком LexA.
При серьезных повреждениях ДНК и появлении коротких
оц ДНК индуцируется автопротеолиз RecA, транскрипция генов umuC и umuD активируется, и из соответствующих белков собирается комплекс D2C.
Повреждения продолжают накапливаться. В клетке становится еще больше RecA в форме, связанной с оцДНК. Это стимулирует автопротеолиз белка UmuD, приводя к образованию укороченной изоформы UmuD’ и, соответственно, комплекса D’2C.
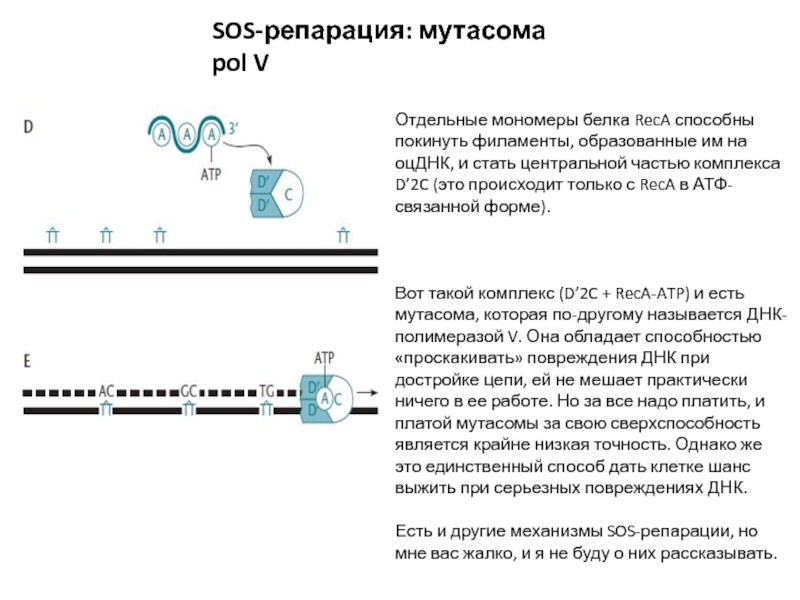
Слайд 32SOS-репарация: мутасома pol V
Отдельные мономеры белка RecA способны покинуть филаменты,
образованные им на оцДНК, и стать центральной частью комплекса D’2C
(это происходит только с RecA в АТФ-связанной форме).
Вот такой комплекс (D’2C + RecA-ATP) и есть мутасома, которая по-другому называется ДНК-полимеразой V. Она обладает способностью «проскакивать» повреждения ДНК при достройке цепи, ей не мешает практически ничего в ее работе. Но за все надо платить, и платой мутасомы за свою сверхспособность является крайне низкая точность. Однако же это единственный способ дать клетке шанс выжить при серьезных повреждениях ДНК.
Есть и другие механизмы SOS-репарации, но мне вас жалко, и я не буду о них рассказывать.
Слайд 33Обзор типов бактериальной репарации и ее участников