Разделы презентаций
- Разное
- Английский язык
- Астрономия
- Алгебра
- Биология
- География
- Геометрия
- Детские презентации
- Информатика
- История
- Литература
- Математика
- Медицина
- Менеджмент
- Музыка
- МХК
- Немецкий язык
- ОБЖ
- Обществознание
- Окружающий мир
- Педагогика
- Русский язык
- Технология
- Физика
- Философия
- Химия
- Шаблоны, картинки для презентаций
- Экология
- Экономика
- Юриспруденция
PSI-BLAST. Множественное выравнивание. Профили. Домены
Содержание
- 1. PSI-BLAST. Множественное выравнивание. Профили. Домены
- 2. BLAST не может решить две проблемы[1] При
- 3. Position specific iterated BLAST: PSI-BLASTЦель PSI-BLAST -
- 4. Поиск в PSI-BLAST выполняется в пять шагов[1]
- 5. Проверка вывода BLASTP для выявления эмпирических "правил"
- 6. Кафедра биоинформатики МБФ РНИМУ
- 7. Кафедра биоинформатики МБФ РНИМУ
- 8. Кафедра биоинформатики МБФ РНИМУПоиск в PSI-BLAST выполняется
- 9. Кафедра биоинформатики МБФ РНИМУ
- 10. Кафедра биоинформатики МБФ РНИМУПоиск в PSI-BLAST выполняется
- 11. Результаты поиска PSI-BLASTКафедра биоинформатики МБФ РНИМУ Кол. посл.Итерация Кол. посл. > threshold 1 104 49 2 173 96 3 236 178 4 301 240 5 344 283 6 342 298 7 378 310 8 382 320
- 12. Поиск PSI-BLAST: RBP4 человека по RefSeq БД, итерация 1Кафедра биоинформатики МБФ РНИМУ
- 13. Кафедра биоинформатики МБФ РНИМУПоиск PSI-BLAST: RBP4 человека по RefSeq БД, итерация 2
- 14. Кафедра биоинформатики МБФ РНИМУПоиск PSI-BLAST: RBP4 человека по RefSeq БД, итерация 3
- 15. Парное выравнивание RBP4 с ApoD, PSI-BLAST итерация 1, E value 3e-07Кафедра биоинформатики МБФ РНИМУ
- 16. Кафедра биоинформатики МБФ РНИМУПарное выравнивание RBP4 с ApoD, PSI-BLAST итерация 2, E value 1e-42!!!
- 17. Кафедра биоинформатики МБФ РНИМУПарное выравнивание RBP4 с ApoD, PSI-BLAST итерация 3, E value 6e-34
- 18. Кафедра биоинформатики МБФ РНИМУВселенная липокалинов (каждая точка - белок)retinol-binding proteinodorant-binding proteinapolipoprotein D
- 19. Кафедра биоинформатики МБФ РНИМУСкоринг матрицы позволяют сосредоточиться на большой (или маленькой) картинеretinol-binding proteinЗапрос RBP
- 20. Кафедра биоинформатики МБФ РНИМУretinol-binding proteinretinol-binding proteinPAM250 PAM30
- 21. Кафедра биоинформатики МБФ РНИМУretinol-binding proteinretinol-binding proteinPSI-BLAST создает скоринг матрицы более мощные чем PAM или BLOSUM
- 22. PSI-BLAST: оценка эффективностиPSI-BLAST полезeн для обнаружения слабых, но биологически значимых связей между белками (
- 23. Искажение определяется как присутствие, по меньшей мере, одного ложно-положительного выравнивания со значением E
- 24. Множественное выравнивание последовательностейЭволюционный анализопределение гомологиифилогенетические построенияэволюционные тестовые
- 25. Набор из трех или более белковых (или
- 26. Кафедра биоинформатики МБФ РНИМУМножественное выравнивание последовательностейN. Provart & D. Guttman. Bioinformatic Methods I. Coursera
- 27. Кафедра биоинформатики МБФ РНИМУHomoloGene включает группы эукариотических белков, парные и множественные выравнивания, и много другое
- 28. Пример. Шаг 1: в NCBI выберете меню
- 29. Кафедра биоинформатики МБФ РНИМУПример. Шаг 2: проверить
- 30. Кафедра биоинформатики МБФ РНИМУПример. Шаг 3: проверим
- 31. Кафедра биоинформатики МБФ РНИМУДругое множественное выравнивание, Rac:Эта вставка может быть альтернативным сплайсингом
- 32. Пример: 5 выравниваний 5 глобиновДавайте посмотрим на
- 33. Кафедра биоинформатики МБФ РНИМУClustalWОбратите внимание как участок консервативного гистидина (▼) изменяется в зависимости от используемого алгоритма
- 34. Кафедра биоинформатики МБФ РНИМУPraline
- 35. Кафедра биоинформатики МБФ РНИМУProbcons
- 36. Кафедра биоинформатики МБФ РНИМУTCoffee
- 37. Не обязательно, что существует одно "правильное" выравнивание
- 38. некоторые выровненные остатки, такие как цистеина, образующие
- 39. ДоменыКафедра биоинформатики МБФ РНИМУДомен белка — элемент
- 40. Кафедра биоинформатики МБФ РНИМУЛейциновая молнияСвязка из 4 спиралейГлобиновая укладка-ДОМЕНЫ
- 41. Кафедра биоинформатики МБФ РНИМУ-ДОМЕНЫ Up and down barrel Баррел на основе греческих ключейJelly roll barrel
- 42. Кафедра биоинформатики МБФ РНИМУ/-ДОМЕНЫПодкова/-пропеллерУкладка РоссманаTIM-укладкаМетилмалонил-коА-мутаза
- 43. Более чувствительно, чем попарное выравнивания для выявления
- 44. Методы множественного выравнивания Точные методы Прогрессивный (ClustalW) Итеративный (MUSCLE) Согласованный (ProbCons) Основанный на структуре (Expresso)Кафедра биоинформатики МБФ РНИМУ
- 45. Прогрессивный метод (ClustalW)Прогрессивные методы: используют направляющей дерево
- 46. Шаг 1. Построение попарных выравниванийКафедра биоинформатики МБФ
- 47. Кафедра биоинформатики МБФ РНИМУ 5 близко родственных глобиновКонвертация баллов сходства в баллы расстояния
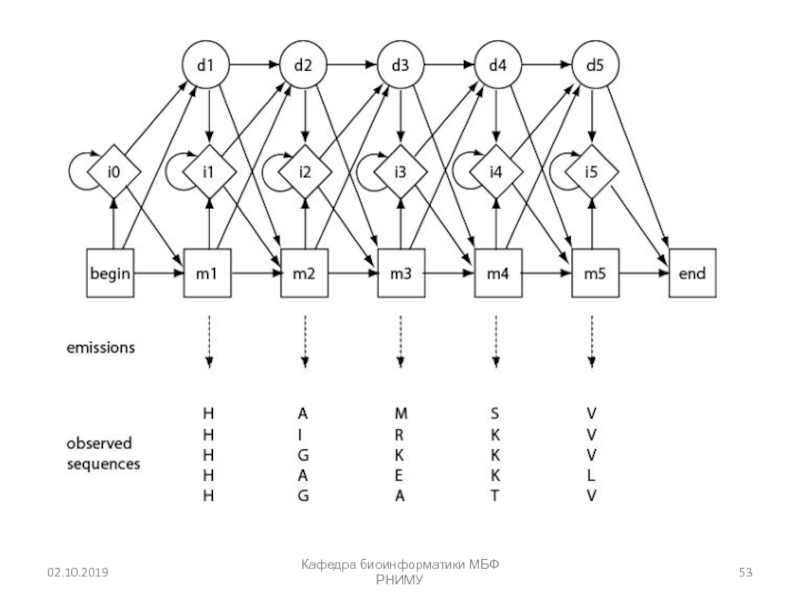
- 48. Множественное выравнивание для профилей скрытых Марковских моделей
- 49. Простая Марковская модельКафедра биоинформатики МБФ РНИМУДождь =
- 50. Простая скрытая Марковская модельКафедра биоинформатики МБФ РНИМУНаблюдение:
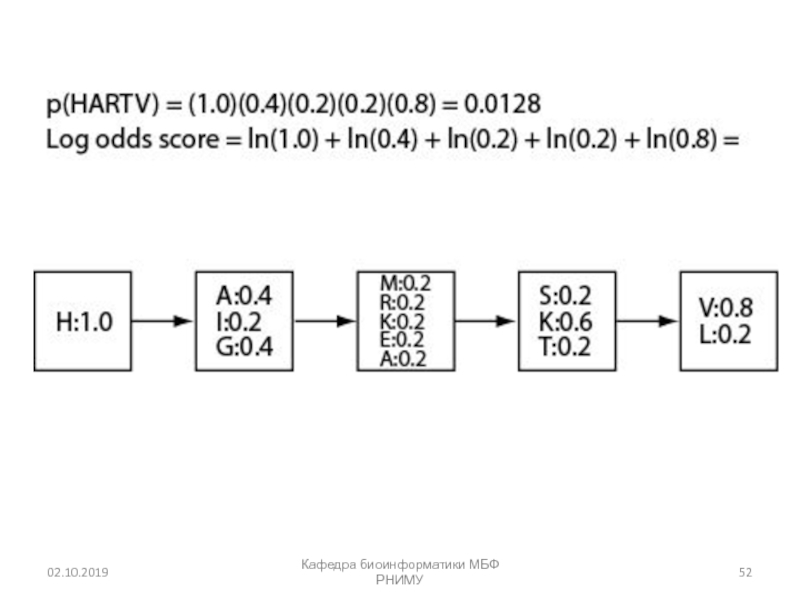
- 51. Кафедра биоинформатики МБФ РНИМУ
- 52. Кафедра биоинформатики МБФ РНИМУ
- 53. Кафедра биоинформатики МБФ РНИМУ
- 54. МотивыМотив в молекулярной биологии — это характерная
- 55. Регулярные выраженияАлфавит — совокупность отдельных символов, обозначающих определенную
- 56. PROSITE – база данных для поиска мотивов
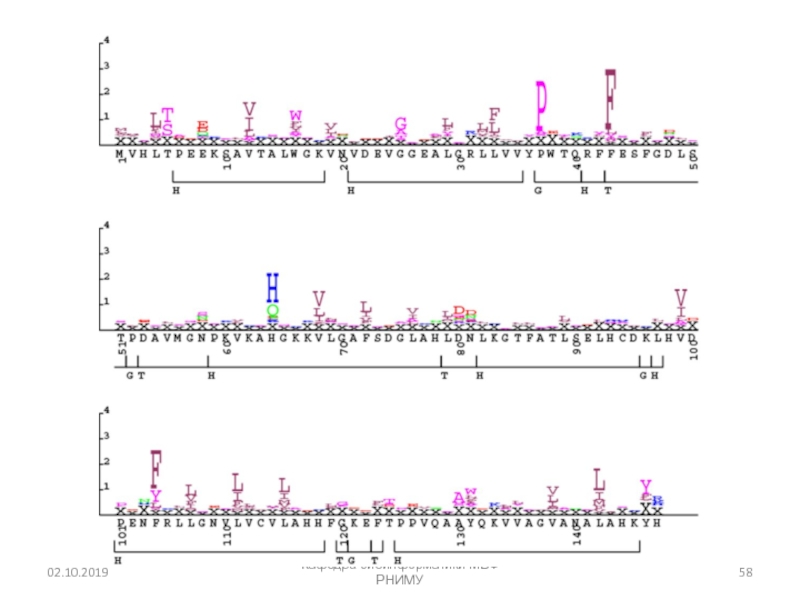
- 57. Мотив домена цинковый палец:C-х (2,4)-C-х (3)-[LIVMFYWC]- х(8)-H-x(3,5)-HКафедра биоинформатики МБФ РНИМУ
- 58. Кафедра биоинформатики МБФ РНИМУ
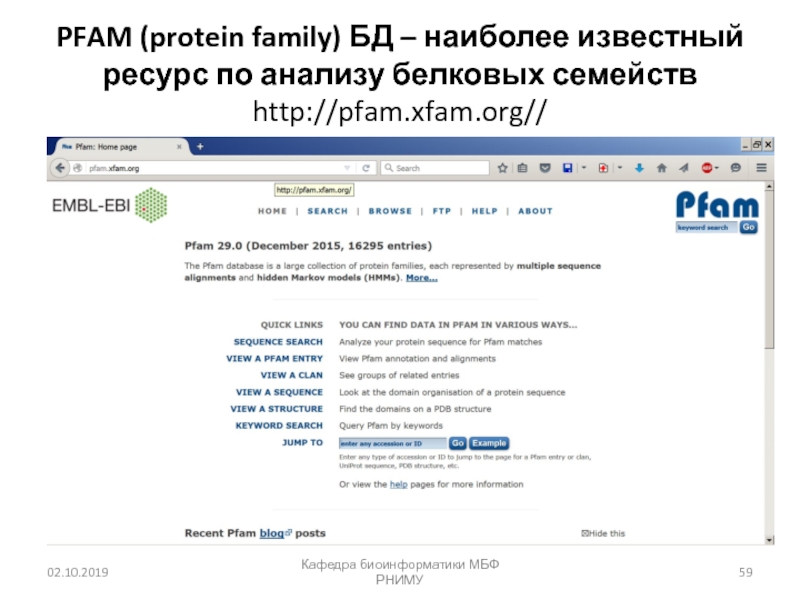
- 59. Кафедра биоинформатики МБФ РНИМУPFAM (protein family) БД – наиболее известный ресурс по анализу белковых семействhttp://pfam.xfam.org//
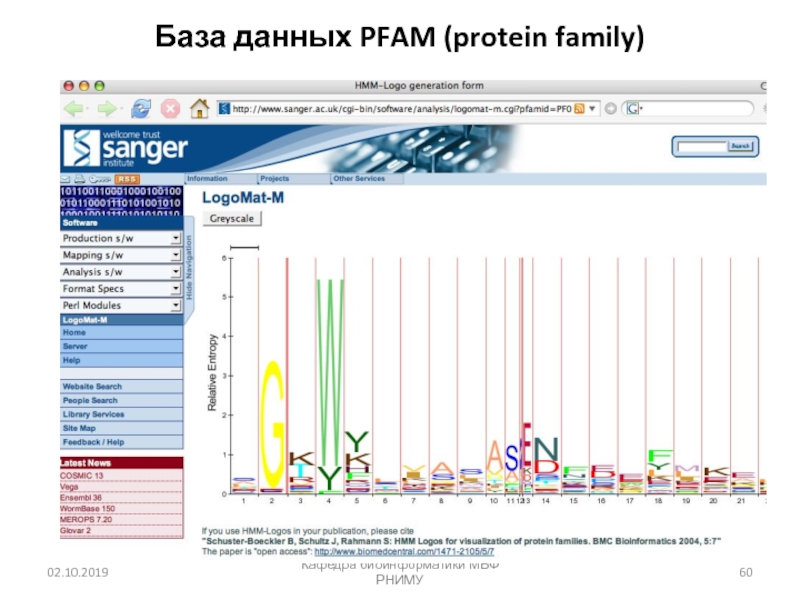
- 60. Кафедра биоинформатики МБФ РНИМУБаза данных PFAM (protein family)
- 61. Скачать презентанцию
Слайды и текст этой презентации
Слайд 1PSI-BLAST. Множественное выравнивание. Профили. Домены
Кафедра биоинформатики МБФ РНИМУ
Многие слайды и
Слайд 2BLAST не может решить две проблемы
[1] При использовании человеческого бета-глобина
в виде запроса для белков RefSeq, BLASTP не "найдет" миоглобин
человека. Потому что эти два белка имеют слишком отдаленное родство. PSI-BLAST в NCBI, а также скрытые Марковские модели легко решают эту проблему.[2] Нельзя задавать запрос для поиска в виде 10 000 пар оснований или миллионов пар оснований. Многие BLAST подобные инструменты для геномной ДНК имеют такие возможности: PatternHunter, Megablast, BLAT и BLASTZ.
Кафедра биоинформатики МБФ РНИМУ
Слайд 3Position specific iterated BLAST: PSI-BLAST
Цель PSI-BLAST - посмотреть глубже в
базу данных в поисках совпадений с вашей последовательностью белка путем
использования оценочной матрицы, которая настроена на ваш запрос.Общая идея : заменяем сиквенс белка вероятностной моделью семейства белков
Кафедра биоинформатики МБФ РНИМУ
Слайд 4Поиск в PSI-BLAST выполняется в пять шагов
[1] Выберите последовательность и
запустите поиск в базе данных последовательностей белков [2] PSI-BLAST строит множественное
выравнивание последовательностей затем создает «профиль» или специализированную позиционно-специфическую оценочную матрицу (PSSM - position-specific scoring matrix).Кафедра биоинформатики МБФ РНИМУ
Слайд 5Проверка вывода BLASTP для выявления эмпирических "правил" в отношении изменчивости
аминокислот в каждой позиции
Кафедра биоинформатики МБФ РНИМУ
R,I,K
C
D,E,T
K,R,T
N,L,Y,G
Слайд 6Кафедра биоинформатики МБФ РНИМУ
A
R N D C Q E G H I L
K M F P S T W Y V1 M -1 -2 -2 -3 -2 -1 -2 -3 -2 1 2 -2 6 0 -3 -2 -1 -2 -1 1
2 K -1 1 0 1 -4 2 4 -2 0 -3 -3 3 -2 -4 -1 0 -1 -3 -2 -3
3 W -3 -3 -4 -5 -3 -2 -3 -3 -3 -3 -2 -3 -2 1 -4 -3 -3 12 2 -3
4 V 0 -3 -3 -4 -1 -3 -3 -4 -4 3 1 -3 1 -1 -3 -2 0 -3 -1 4
5 W -3 -3 -4 -5 -3 -2 -3 -3 -3 -3 -2 -3 -2 1 -4 -3 -3 12 2 -3
6 A 5 -2 -2 -2 -1 -1 -1 0 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 0
7 L -2 -2 -4 -4 -1 -2 -3 -4 -3 2 4 -3 2 0 -3 -3 -1 -2 -1 1
8 L -1 -3 -3 -4 -1 -3 -3 -4 -3 2 2 -3 1 3 -3 -2 -1 -2 0 3
9 L -1 -3 -4 -4 -1 -2 -3 -4 -3 2 4 -3 2 0 -3 -3 -1 -2 -1 2
10 L -2 -2 -4 -4 -1 -2 -3 -4 -3 2 4 -3 2 0 -3 -3 -1 -2 -1 1
11 A 5 -2 -2 -2 -1 -1 -1 0 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 0
12 A 5 -2 -2 -2 -1 -1 -1 0 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 0
13 W -2 -3 -4 -4 -2 -2 -3 -4 -3 1 4 -3 2 1 -3 -3 -2 7 0 0
14 A 3 -2 -1 -2 -1 -1 -2 4 -2 -2 -2 -1 -2 -3 -1 1 -1 -3 -3 -1
15 A 2 -1 0 -1 -2 2 0 2 -1 -3 -3 0 -2 -3 -1 3 0 -3 -2 -2
16 A 4 -2 -1 -2 -1 -1 -1 3 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 -1
...
37 S 2 -1 0 -1 -1 0 0 0 -1 -2 -3 0 -2 -3 -1 4 1 -3 -2 -2
38 G 0 -3 -1 -2 -3 -2 -2 6 -2 -4 -4 -2 -3 -4 -2 0 -2 -3 -3 -4
39 T 0 -1 0 -1 -1 -1 -1 -2 -2 -1 -1 -1 -1 -2 -1 1 5 -3 -2 0
40 W -3 -3 -4 -5 -3 -2 -3 -3 -3 -3 -2 -3 -2 1 -4 -3 -3 12 2 -3
41 Y -2 -2 -2 -3 -3 -2 -2 -3 2 -2 -1 -2 -1 3 -3 -2 -2 2 7 -1
42 A 4 -2 -2 -2 -1 -1 -1 0 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 0
20 аминокислот
Все аминокислоты от позиции 1 до последней позиции белковой последовательности запроса в PSI-BLAST
Слайд 7Кафедра биоинформатики МБФ РНИМУ
A
R N D C Q E G H I L
K M F P S T W Y V1 M -1 -2 -2 -3 -2 -1 -2 -3 -2 1 2 -2 6 0 -3 -2 -1 -2 -1 1
2 K -1 1 0 1 -4 2 4 -2 0 -3 -3 3 -2 -4 -1 0 -1 -3 -2 -3
3 W -3 -3 -4 -5 -3 -2 -3 -3 -3 -3 -2 -3 -2 1 -4 -3 -3 12 2 -3
4 V 0 -3 -3 -4 -1 -3 -3 -4 -4 3 1 -3 1 -1 -3 -2 0 -3 -1 4
5 W -3 -3 -4 -5 -3 -2 -3 -3 -3 -3 -2 -3 -2 1 -4 -3 -3 12 2 -3
6 A 5 -2 -2 -2 -1 -1 -1 0 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 0
7 L -2 -2 -4 -4 -1 -2 -3 -4 -3 2 4 -3 2 0 -3 -3 -1 -2 -1 1
8 L -1 -3 -3 -4 -1 -3 -3 -4 -3 2 2 -3 1 3 -3 -2 -1 -2 0 3
9 L -1 -3 -4 -4 -1 -2 -3 -4 -3 2 4 -3 2 0 -3 -3 -1 -2 -1 2
10 L -2 -2 -4 -4 -1 -2 -3 -4 -3 2 4 -3 2 0 -3 -3 -1 -2 -1 1
11 A 5 -2 -2 -2 -1 -1 -1 0 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 0
12 A 5 -2 -2 -2 -1 -1 -1 0 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 0
13 W -2 -3 -4 -4 -2 -2 -3 -4 -3 1 4 -3 2 1 -3 -3 -2 7 0 0
14 A 3 -2 -1 -2 -1 -1 -2 4 -2 -2 -2 -1 -2 -3 -1 1 -1 -3 -3 -1
15 A 2 -1 0 -1 -2 2 0 2 -1 -3 -3 0 -2 -3 -1 3 0 -3 -2 -2
16 A 4 -2 -1 -2 -1 -1 -1 3 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 -1
...
37 S 2 -1 0 -1 -1 0 0 0 -1 -2 -3 0 -2 -3 -1 4 1 -3 -2 -2
38 G 0 -3 -1 -2 -3 -2 -2 6 -2 -4 -4 -2 -3 -4 -2 0 -2 -3 -3 -4
39 T 0 -1 0 -1 -1 -1 -1 -2 -2 -1 -1 -1 -1 -2 -1 1 5 -3 -2 0
40 W -3 -3 -4 -5 -3 -2 -3 -3 -3 -3 -2 -3 -2 1 -4 -3 -3 12 2 -3
41 Y -2 -2 -2 -3 -3 -2 -2 -3 2 -2 -1 -2 -1 3 -3 -2 -2 2 7 -1
42 A 4 -2 -2 -2 -1 -1 -1 0 -2 -2 -2 -1 -1 -3 -1 1 0 -3 -2 0
обратите внимание, что данная аминокислота (например, аланин) в последовательности запроса может по разному оцениваться при совпадении с аланином - в зависимости от положения в белке
Слайд 8Кафедра биоинформатики МБФ РНИМУ
Поиск в PSI-BLAST выполняется в пять шагов
[1]
Выберите последовательность и запустите поиск в базе данных последовательностей белков [2]
PSI-BLAST строит множественное выравнивание последовательностей затем создает «профиль» или специализированную позиционно-специфическую оценочную матрицу (PSSM - position-specific scoring matrix).[3] PSSM используется при запросе для дальнейшего поиска в базе данных
[4] PSI-BLAST оценивает статистическую значимость (E values)
Слайд 10Кафедра биоинформатики МБФ РНИМУ
Поиск в PSI-BLAST выполняется в пять шагов
[1]
Выберите последовательность и запустите поиск в базе данных последовательностей белков [2]
PSI-BLAST строит множественное выравнивание последовательностей затем создает «профиль» или специализированную позиционно-специфическую оценочную матрицу (PSSM - position-specific scoring matrix).[3] PSSM используется как запрос для поиска в базе данных
[4] PSI-BLAST оценивает статистическую значимость (E values)
[5] Итеративное повторение шагов [3] и [4], обычно 5 раз.
При каждом новом поиске, новый профиль используется в качестве запроса.
Слайд 11Результаты поиска PSI-BLAST
Кафедра биоинформатики МБФ РНИМУ
Кол. посл.
Итерация Кол. посл. >
threshold
1 104 49
2 173 96
3 236 178
4 301 240
5 344 283
6 342 298
7 378 310
8 382 320
Слайд 15 Парное выравнивание RBP4 с ApoD, PSI-BLAST итерация 1, E value
3e-07
Кафедра биоинформатики МБФ РНИМУ
Слайд 16Кафедра биоинформатики МБФ РНИМУ
Парное выравнивание RBP4 с ApoD, PSI-BLAST итерация
2,
E value 1e-42!!!
Слайд 17Кафедра биоинформатики МБФ РНИМУ
Парное выравнивание RBP4 с ApoD, PSI-BLAST итерация
3,
E value 6e-34
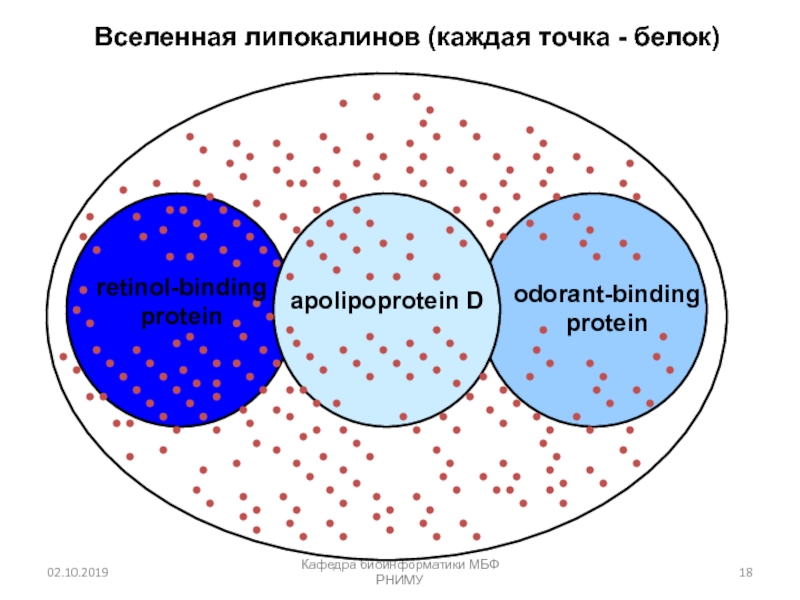
Слайд 18Кафедра биоинформатики МБФ РНИМУ
Вселенная липокалинов (каждая точка - белок)
retinol-binding
protein
odorant-binding
protein
apolipoprotein D
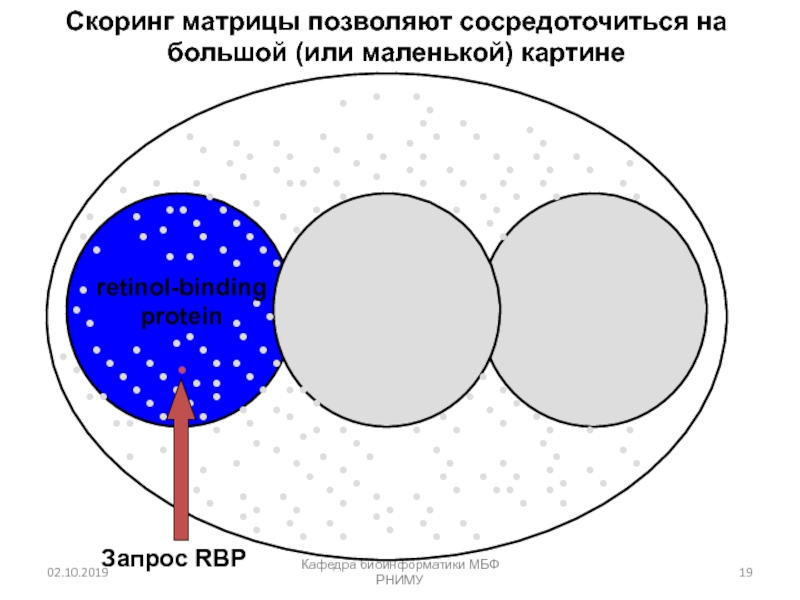
Слайд 19Кафедра биоинформатики МБФ РНИМУ
Скоринг матрицы позволяют сосредоточиться на большой (или
маленькой) картине
retinol-binding
protein
Запрос RBP
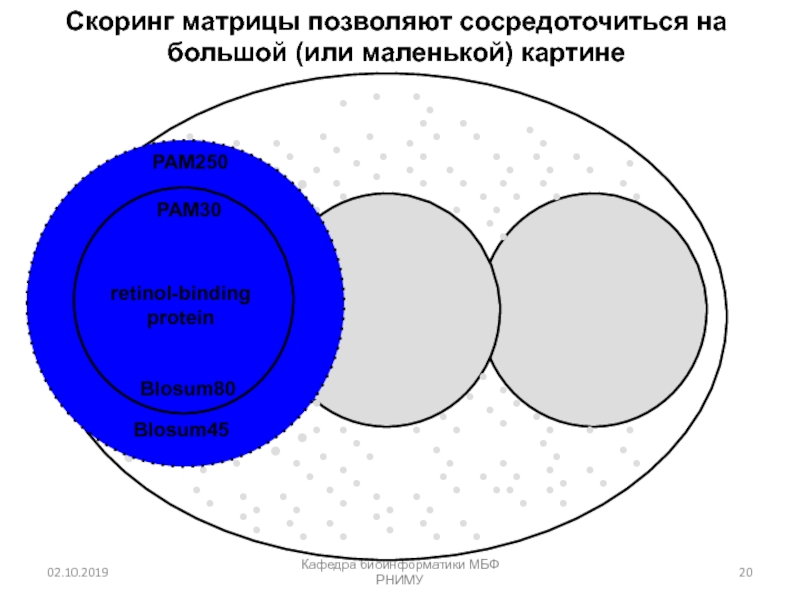
Слайд 20Кафедра биоинформатики МБФ РНИМУ
retinol-binding
protein
retinol-binding
protein
PAM250
PAM30
Blosum45
Blosum80
Скоринг
матрицы позволяют сосредоточиться на большой (или маленькой) картине
Слайд 21Кафедра биоинформатики МБФ РНИМУ
retinol-binding
protein
retinol-binding
protein
PSI-BLAST создает скоринг матрицы более
мощные чем PAM или BLOSUM
Слайд 22PSI-BLAST: оценка эффективности
PSI-BLAST полезeн для обнаружения слабых, но биологически значимых
связей между белками (
ложное усиление последовательностей, не связанных с запросом. Например, запрос с биспиральным (coiled-coil) мотивом может выявить тысячи других негомологичных белков с этим мотивом.Даже однажды вошедший выше порога в результат поиска PSI-BLAST ложный белок останется при последующих итерациях – проблема искажения (corruption)
Кафедра биоинформатики МБФ РНИМУ
Слайд 23Искажение определяется как присутствие, по меньшей мере, одного ложно-положительного выравнивания
со значением E
с искажением: [1] Применить фильтрацию искажающих участков профиля сгенерированного PSI-BLAST (например, программа SEG: http://www.ncbi.nlm.nih.gov/Education/BLASTinfo/Seg.html [2] Настроить порог E значения ниже 0,001 (по умолчанию), например E = 0,0001. [3] Просмотреть результаты каждой из итерации. Удалить подозрительные хиты, сняв флажок.Кафедра биоинформатики МБФ РНИМУ
PSI-BLAST: проблема искажения
Слайд 24Множественное выравнивание последовательностей
Эволюционный анализ
определение гомологии
филогенетические построения
эволюционные тестовые модели
Функциональный анализ
определить консервативные
участки
идентификация белковых семейств
Структурный анализ
определить последовательность ковариация
моделирование гомологии
Практическое применение
определить консервативные сайты
связывания праймеровконструирование мутагенетических экспериментов
анализ мутантов
Кафедра биоинформатики МБФ РНИМУ
В 1990-х исследователи начали понимать, что выравнивание нескольких последовательностей (профилей) дает гораздо больше информации, чем парные выравнивания.
Слайд 25Набор из трех или более белковых (или нуклеотидных) последовательностей, которые
частично или полностью выровнены
Гомологичные остатки выровнены в столбцах по всей
длине последовательностейОстатки гомологичны в эволюционном смысле
Остатки гомологичны в структурном смысле
Кафедра биоинформатики МБФ РНИМУ
Множественное выравнивание последовательностей
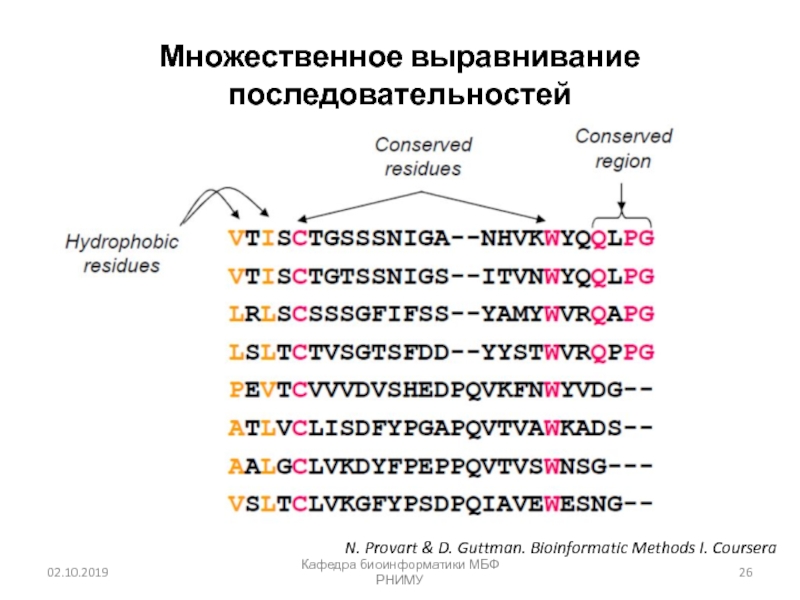
Слайд 26Кафедра биоинформатики МБФ РНИМУ
Множественное выравнивание последовательностей
N. Provart & D. Guttman.
Bioinformatic Methods I. Coursera
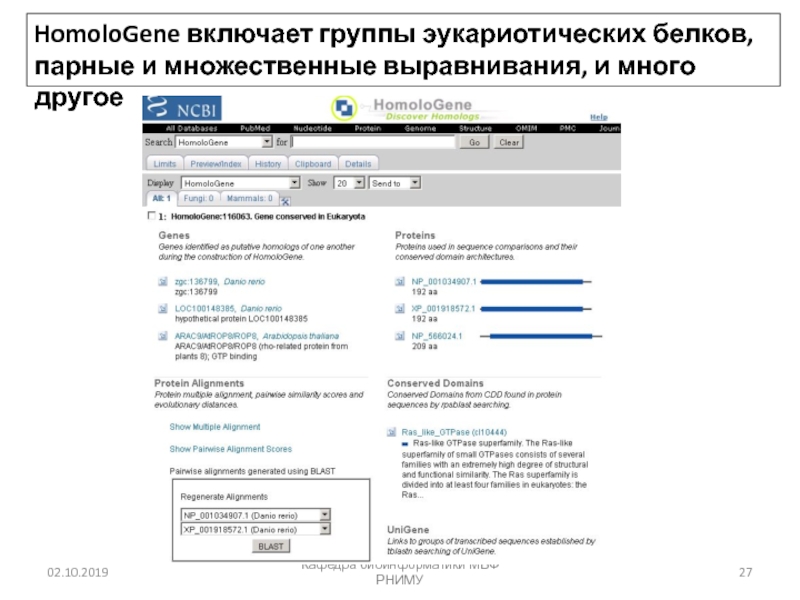
Слайд 27Кафедра биоинформатики МБФ РНИМУ
HomoloGene включает группы эукариотических белков, парные и
множественные выравнивания, и много другое
Слайд 28Пример. Шаг 1: в NCBI выберете меню HomoloGene и введите
caveolin в поле поиска
Кафедра биоинформатики МБФ РНИМУ
Слайд 29Кафедра биоинформатики МБФ РНИМУ
Пример. Шаг 2: проверить результаты. Возьмем первый
набор кавеолинов. Изменить Display на Multiple alignment.
Слайд 30Кафедра биоинформатики МБФ РНИМУ
Пример. Шаг 3: проверим множественное выравнивание. Восемь
белков хорошо выравнены, хотя пробелы также включены.
Слайд 31Кафедра биоинформатики МБФ РНИМУ
Другое множественное выравнивание, Rac:
Эта вставка может быть
альтернативным сплайсингом
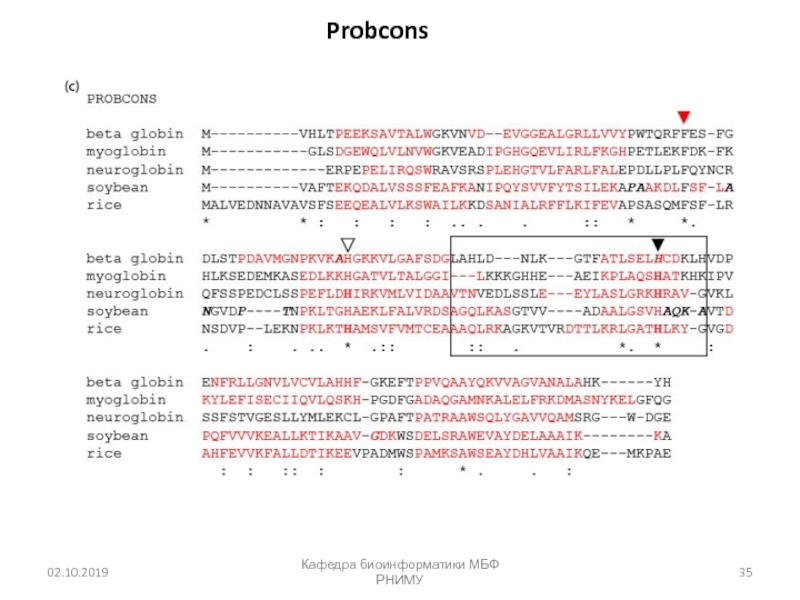
Слайд 32Пример: 5 выравниваний 5 глобинов
Давайте посмотрим на множественное выравнивание последовательности
(MSA) пяти глобинов белков. Мы будем использовать пять известных программ
MSA: ClustalW, Praline, MUSCLE (используется в HomoloGene), ProbCons и TCoffee. Каждая программа имеет уникальные особенности.Мы сосредоточимся на остатках гистидина (H), который имеет важную роль в связывании кислорода в глобинах, и должны быть выровнены. Но часто выравнивание не совпадает, и все пять программ дают разные ответы.
Вывод: не существует единственно верного подхода к MSA. Десятки новых программ были разработаны в последние годы.
Кафедра биоинформатики МБФ РНИМУ
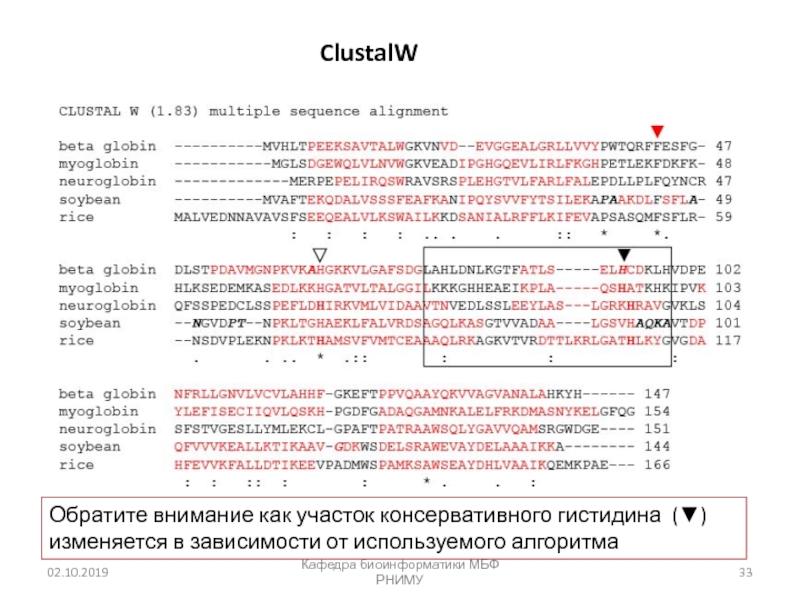
Слайд 33Кафедра биоинформатики МБФ РНИМУ
ClustalW
Обратите внимание как участок консервативного гистидина (▼)
изменяется в зависимости от используемого алгоритма
Слайд 37Не обязательно, что существует одно "правильное" выравнивание семейства белков
Эволюционируют белковые
последовательности …
Соответствующие трехмерные структуры белков также эволюционируют…
может оказаться невозможным идентификации
аминокислотных остатков, которые выравниваются должным образом (структурно) в течение множественного выравнивания последовательностейДля двух белков, с 30% идентичностью аминокислотной последовательности, совмещается около 50% отдельных аминокислот в двух структурах
Кафедра биоинформатики МБФ РНИМУ
Свойства множественного выравнивания последовательностей
Слайд 38некоторые выровненные остатки, такие как цистеина, образующие дисульфидные мостики, могут
быть высоко консервативны
может быть консервативные домены, такие как трансмембранный домен
может
быть консервативны особенности вторичной структурыможет быть участки в последовательностях являются паттернами вставок или делеций
Кафедра биоинформатики МБФ РНИМУ
Особенности множественного выравнивания последовательностей
Слайд 39Домены
Кафедра биоинформатики МБФ РНИМУ
Домен белка — элемент третичной структуры белка,
представляющий собой достаточно стабильную и независимую подструктуру белка, фолдинг которой
проходит независимо от остальных частей [wikipedia].Домен – это часть полипептидной цепи (или вся цепочка), которая сворачивается независимо в стабильную третичную структуру [C.Brenden & John Tooze]
Доменами в белках называют области в третичной структуре, которым свойственна определенная автономия структурной организации [Степанов В.М.]
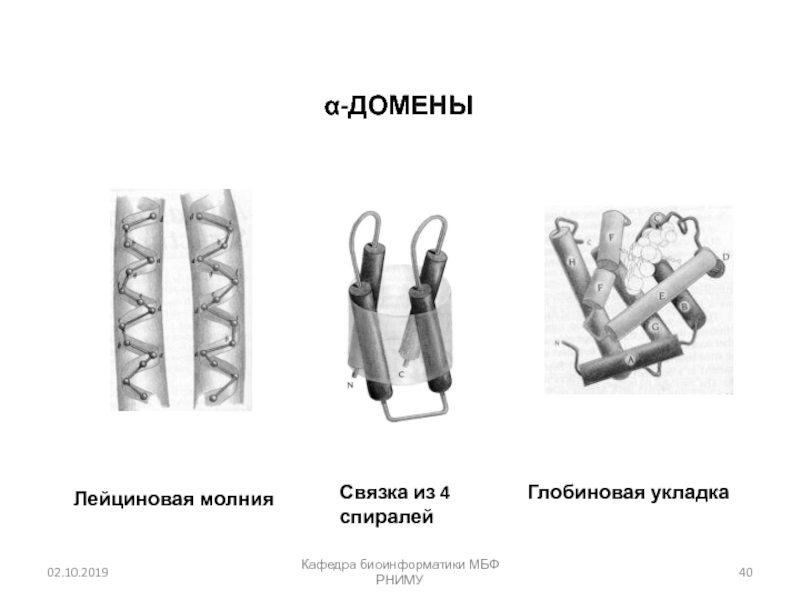
Слайд 40Кафедра биоинформатики МБФ РНИМУ
Лейциновая молния
Связка из 4
спиралей
Глобиновая укладка
-ДОМЕНЫ
Слайд 41Кафедра биоинформатики МБФ РНИМУ
-ДОМЕНЫ
Up and down barrel
Баррел на
основе
греческих ключей
Jelly roll barrel
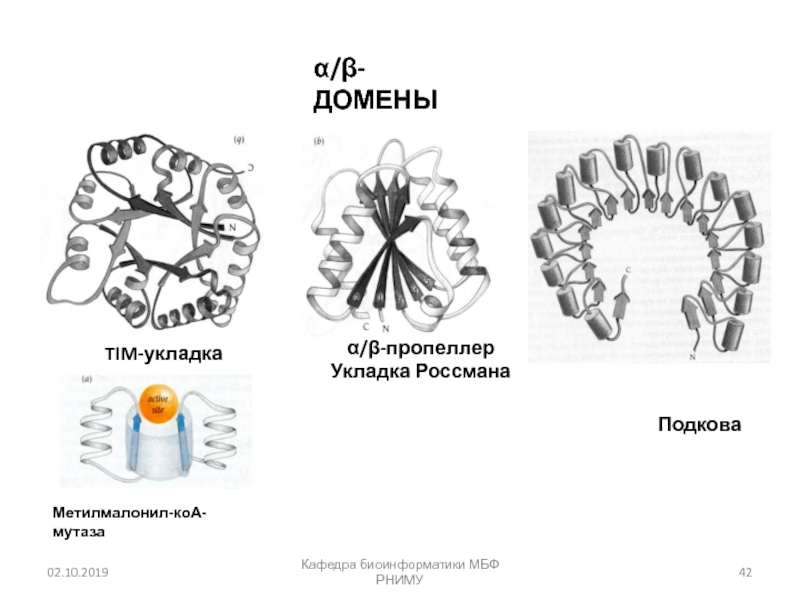
Слайд 42Кафедра биоинформатики МБФ РНИМУ
/-ДОМЕНЫ
Подкова
/-пропеллер
Укладка Россмана
TIM-укладка
Метилмалонил-коА-мутаза
Слайд 43Более чувствительно, чем попарное выравнивания для выявления гомологов
Результат BLAST может
принять форму множественного выравнивания, и может раскрыть консервативные остатки или
мотивыДемографические данные могут быть проанализированы в множественном выравнивании (PopSet)
Отдельный запрос может быть использован для поиска в базе данных множественных выравниваний (например, PFAM)
Регуляторные области генов могут быть консенсусными последовательностями идентифицируемыми множественным выравниванием
Кафедра биоинформатики МБФ РНИМУ
Использование множественного выравнивания
Слайд 44Методы множественного выравнивания
Точные методы
Прогрессивный (ClustalW)
Итеративный (MUSCLE)
Согласованный (ProbCons)
Основанный
на структуре (Expresso)
Кафедра биоинформатики МБФ РНИМУ
Слайд 45Прогрессивный метод (ClustalW)
Прогрессивные методы: используют направляющей дерево (связанное с филогенетическим
деревом), чтобы определить, как объединить попарные выравнивания по одному для
создания множественного выравнивания.[1] Сделать ряд глобальных попарных выравниваний (Needleman и Wunsch динамический алгоритм программирования)
[2] Создать направляющее дерево
[3] Постепенно выровнять последовательности
Кафедра биоинформатики МБФ РНИМУ
Слайд 46Шаг 1. Построение попарных выравниваний
Кафедра биоинформатики МБФ РНИМУ
best
Score
(% идентичности)
Для
n последовательностей, (n-1)(n) / 2
Для 5 последовательностей, (4)(5) / 2
= 10Для 200 последовательностей, (199)(200) / 2 = 19,900
Слайд 47Кафедра биоинформатики МБФ РНИМУ
5 близко родственных глобинов
Конвертация баллов сходства
в баллы расстояния
Слайд 48Множественное выравнивание для профилей скрытых Марковских моделей (HMMs - Hidden
Markov models)
Скрытые Марковские модели (HMMs) являются "состояниями", которые описывают вероятность
наличия конкретного аминокислотного остатка расположенного в колонке множественного выравнивания последовательностейHMMs являются вероятностными моделями
HMMs может дать более чувствительные выравнивания, чем традиционные методы, такие как прогрессивное выравнивание
Кафедра биоинформатики МБФ РНИМУ
Слайд 49Простая Марковская модель
Кафедра биоинформатики МБФ РНИМУ
Дождь = собака может не
захотеть выйти на улицу
Солнце = собака, вероятно, выйдет на улицу
Д
С
0.15
0.85
0.2
0.8
Марковское
состояние = не зависимость от ближайшего предыдущего состояния ("Без памяти")courtesy of
Sarah Wheelan
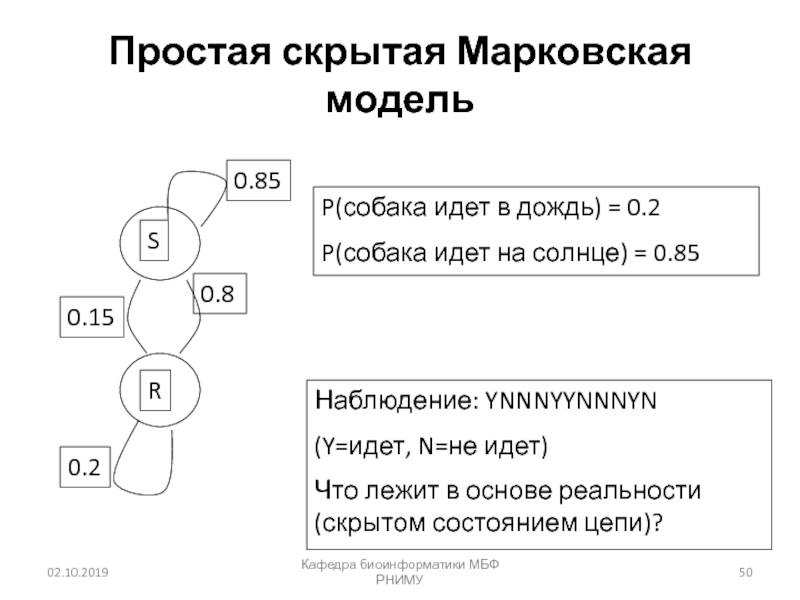
Слайд 50Простая скрытая Марковская модель
Кафедра биоинформатики МБФ РНИМУ
Наблюдение: YNNNYYNNNYN
(Y=идет, N=не идет)
Что
лежит в основе реальности (скрытом состоянием цепи)?
R
S
0.15
0.85
0.2
0.8
P(собака идет в дождь)
= 0.2P(собака идет на солнце) = 0.85
Слайд 54Мотивы
Мотив в молекулярной биологии — это характерная последовательность нуклеотидов (в
ДНК, РНК) или аминокислот (в белках), которые имеют существенное биологическое
значение. Мотивы в белках позволяют найти участки белков, отвечающие за определённые свойства.Для обозначения мотива используют стандартные обозначения регулярных выражений
Кафедра биоинформатики МБФ РНИМУ
Слайд 55Регулярные выражения
Алфавит — совокупность отдельных символов, обозначающих определенную аминокислоту или набор
аминокислот.
Строка из символов алфавита — обозначающая последовательность соответствующих аминокислот.
[ABC] — любая строка
символов, взятых из алфавита в квадратных скобках соответствует любому из соответствующих аминокислот; например [ABC] соответствует любому из аминокислот, из представленных: или a или b или c.{ABC} — любая строка символов, взятых из алфавита соответствует любой аминокислоте кроме тех, что находятся в фигурных скобках; например {ABC} соответствует любой аминокислоте, кроме: a, b и c.
Главная идея, лежащая в этих обозначениях — принцип соответствия: последовательность элементов паттерна совпадает с последовательностью аминокислот, если и только если последнюю последовательность можно разбить на подпоследовательности таким образом, что каждый элемент массива соответствует соответствующий подпоследовательности в свою очередь.
Например, модель [AB] [ CDE ] F соответствует шести последовательности аминокислот: ACF, ADF, AEF, BCF, BDF и BEF.
Кафедра биоинформатики МБФ РНИМУ
Слайд 56PROSITE – база данных для поиска мотивов в белках (prosite.expasy.org)
PROSITE дополняет список выражений, описанных выше:
1. «х» — шаблон элемента обозначают
любую аминокислоту.2. '<' — шаблон ограничивается N-концом последовательности.
3. '>' — шаблон ограничивается C-концом последовательности.
Также символ ' >' может находиться внутри квадратных скобок, например: S [ T> ] соответствует как " ST " и « S >».
4. Если е — шаблон элемента, и m и n два целых десятичных числа и m < = n, то:
- е (m) эквивалентно повторению е ровно m раз - е ( m, n) эквивалентно повторению е ровно k раз для любого целого k удовлетворяющей : m < = k < = n Например:
х (3) эквивалентно Х-Х-Х.
х (2,4) соответствует любой последовательности, которая соответствует хх или ххх или хххх.
Кафедра биоинформатики МБФ РНИМУ

![PSI-BLAST. Множественное выравнивание. Профили. Домены BLAST не может решить две проблемы[1] При использовании человеческого бета-глобина в BLAST не может решить две проблемы[1] При использовании человеческого бета-глобина в виде запроса для белков RefSeq, BLASTP](/img/thumbs/52b3a75a595ca45906ee6e51a958dc4e-800x.jpg)

![PSI-BLAST. Множественное выравнивание. Профили. Домены Поиск в PSI-BLAST выполняется в пять шагов[1] Выберите последовательность и запустите Поиск в PSI-BLAST выполняется в пять шагов[1] Выберите последовательность и запустите поиск в базе данных последовательностей белков](/img/thumbs/2e7dc66031b11df72eebe5af8f7435f4-800x.jpg)



![PSI-BLAST. Множественное выравнивание. Профили. Домены Кафедра биоинформатики МБФ РНИМУПоиск в PSI-BLAST выполняется в пять шагов[1] Выберите Кафедра биоинформатики МБФ РНИМУПоиск в PSI-BLAST выполняется в пять шагов[1] Выберите последовательность и запустите поиск в базе](/img/thumbs/6ebff38b83e1ba51559c70fd1ddb8222-800x.jpg)

![PSI-BLAST. Множественное выравнивание. Профили. Домены Кафедра биоинформатики МБФ РНИМУПоиск в PSI-BLAST выполняется в пять шагов[1] Выберите Кафедра биоинформатики МБФ РНИМУПоиск в PSI-BLAST выполняется в пять шагов[1] Выберите последовательность и запустите поиск в базе](/img/thumbs/82d76b2e6ddb608e2cf087836e1622f8-800x.jpg)

































![PSI-BLAST. Множественное выравнивание. Профили. Домены Мотив домена цинковый палец:C-х (2,4)-C-х (3)-[LIVMFYWC]- х(8)-H-x(3,5)-HКафедра биоинформатики МБФ РНИМУ Мотив домена цинковый палец:C-х (2,4)-C-х (3)-[LIVMFYWC]- х(8)-H-x(3,5)-HКафедра биоинформатики МБФ РНИМУ](/img/thumbs/afdc81710a643884c167acf746476bcf-800x.jpg)