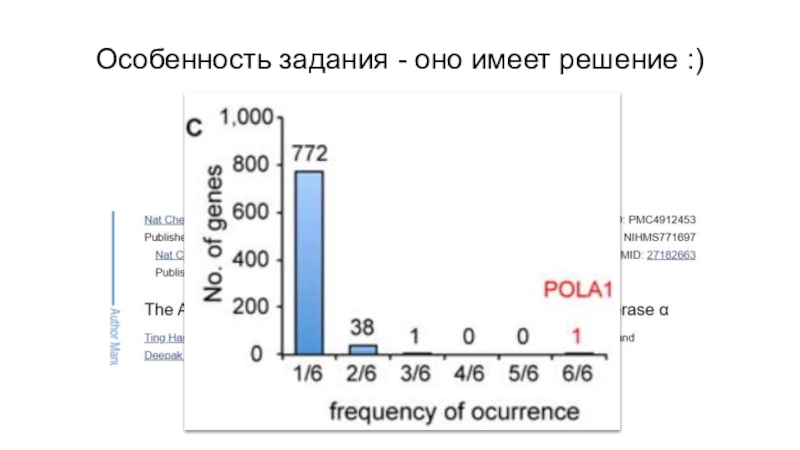
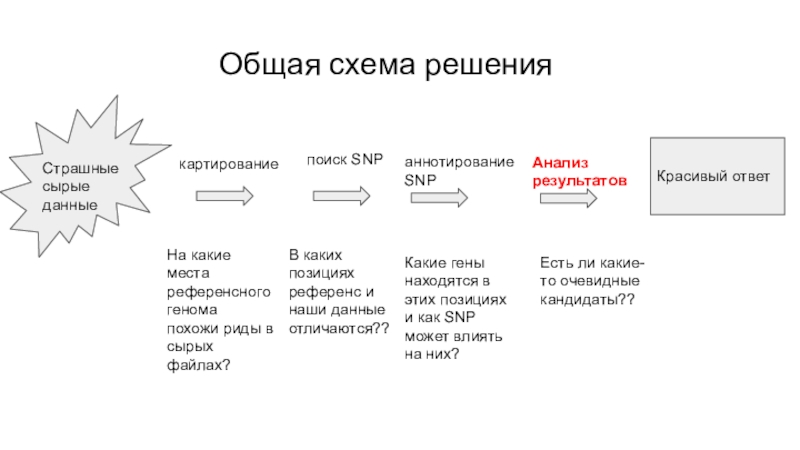
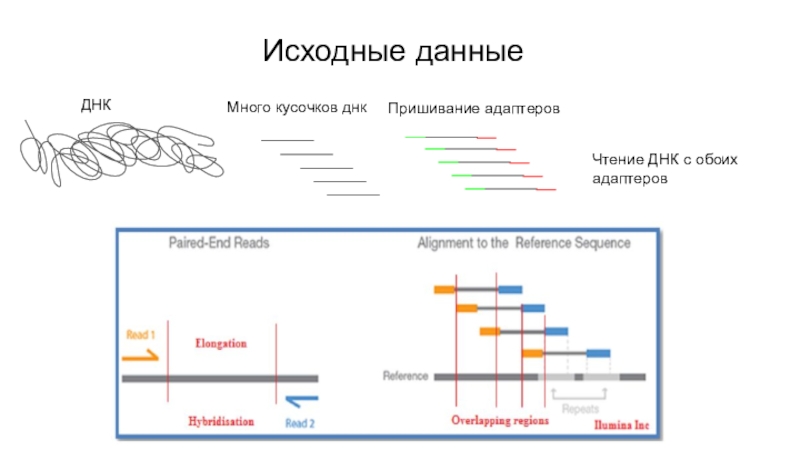
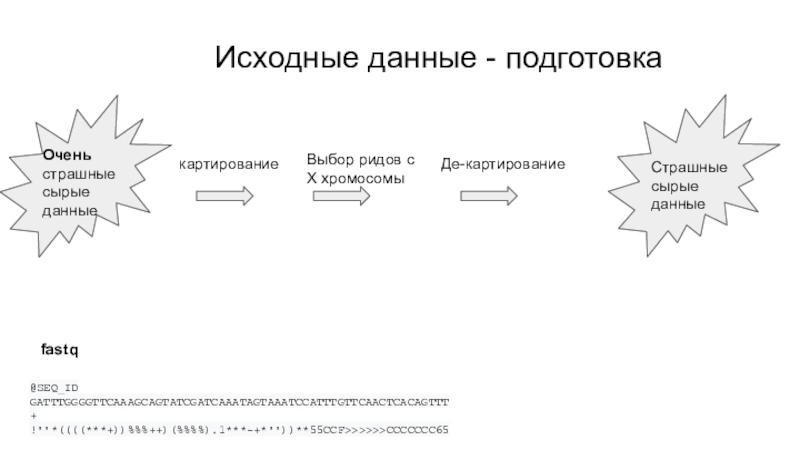
$p
echo "#########################Started mapping#######################"
bwa mem -R "@RG\tID:4\tSM:$p\tPL:illumina\tLB:lib1\tPU:unit1" ./Homo_sapiens_assembly38.fasta "$p"_1.fastq "$p"_2.fastq -t 25 > "$p".sam
echo "####################Deleting fastq files###################"
#rm "$p"_1.fastq
#rm "$p"_2.fastq
echo "##################### Converting files to Bam and sort ###################"
/samtools view -Su "$p".sam | samtools sort -@25 - -o "$p".sorted.bam
echo "##############Deleting sam files###############"
rm "$p".sam
echo "#################mark duplicates################"
java -Dpicard.useLegacyParser=false -Xmx16G -jar ../picard.jar MarkDuplicates\
-I "$p".sorted.bam\
-O "$p".MD.bam\
-METRICS_FILE metrics.txt\
-CREATE_INDEX true
echo "#############Deleting sorted.bam################"
rm "$p".sorted.bam
done < samp_list
resist_1
resist_2
resist_3
resist_4
resist_5
resist_6
sensitive_1
sensitive_2
sensitive_3
sensitive_4
sensitive_5
sensitive_6
samp_list